《统计学习方法(第二版)》学习笔记 第三章 k近邻法 代码实现
文章目录
- 1. kd树节点定义
- 2. 数据处理过程
- 3. 基于kd树的最近邻算法实现和k近邻算法实现
- 3.1 kd树的构建
- 3.2 基于kd树的最近邻算法实现
- 3.3 基于kd树的k近邻算法的实现
这篇博客给出基于kd树的最近邻算法实现和k近邻算法的实现
1. kd树节点定义
Node.py:kd树中的节点定义
class KdNode:
"""
@:param value: 节点值
@:param dimension: 当前节点的划分维度
@:param left: 左子树根节点
@:param right: 右子树根节点
"""
def __init__(self, value):
self.value = value
self.partition_dimension = None
self.left = None
self.right = None
def set_value(self, value):
self.value = value
def __str__(self):
return str(self.value)
2. 数据处理过程
labeled.txt:数据集中包含十个样本,其中前三维是数据维,最后一维为样本标签
40920 8.326976 0.953952 3
14488 7.153469 1.673904 2
26052 1.441871 0.805124 1
75136 13.147394 0.428964 1
38344 1.669788 0.134296 1
72993 10.141740 1.032955 1
35948 6.830792 1.213192 3
42666 13.276369 0.543880 3
67497 8.631577 0.749278 1
35483 12.273169 1.508053 3
DataProcess.py:读入数据集,并对样本的每一维都进行数据归一化,以防止不同维度的量纲对结果的影响。
import numpy as np
def file2matrix(filename):
"""
将数据从文件中读出为矩阵
:param filename: 文件名[.txt]
:return: 返回数据列表
"""
data_matrix = []
with open(filename) as file_hanlder:
for line in file_hanlder.readlines():
sample = line.strip().split(' ')
# print(sample)
# 将sample中的每一个str类型的元素转化为float类型
for i, data in enumerate(sample):
sample[i] = float(data)
# print(i, '+', sample[i])
data_matrix.append(sample)
return data_matrix
def auto_norm(data):
"""
将数据归一化,避免数据各维度间的差异过大对结果造成影响
:param data: 原始带标签的数据
:return: 经过归一化后带标签的数据
"""
# 将样本中的data和label类别拆开,分别存储
samples = []
labels = []
for sample in data:
samples.append(sample[:-1])
labels.append([sample[-1]])
# 先将samples和labels列表转化为numpy数组类型,然后调用其中的min和max方法在0维上(即每一列上)找到最大和最小值
samples = np.array(samples)
labels = np.array(labels)
min_vals = samples.min(0) # 各列的最小值
max_vals = samples.max(0) # 各列的最大值
# print(min_vals)
# print(max_vals)
# 数据归一化公式为:(x - min_vals) / (max_vals - min_vals)
ranges = max_vals - min_vals
norm_dataset = np.zeros(np.shape(samples))
m = samples.shape[0] # 总共有多少行
# np.tile()函数将矩阵按横向和纵向进行复制,这里是将min_val向量在纵向上复制m次,在横向上不变
norm_dataset = samples - np.tile(min_vals, (m, 1))
norm_dataset = norm_dataset / np.tile(ranges, (m, 1))
# 将样本和对应的标签进行拼接,得到数据归一化后带标签的样本
norm_dataset = np.hstack((norm_dataset, labels))
print("经过数据归一化后得到的数据集为:")
for i, sample in enumerate(norm_dataset):
print("sample ", i+1, ': ', sample)
return norm_dataset
if __name__ == '__main__':
# Test
data = file2matrix('labeled_data.txt')
data = auto_norm(data)
print(data)
3. 基于kd树的最近邻算法实现和k近邻算法实现
3.1 kd树的构建
读入数据,顺序选择数据的维度作为kd树的每一层的排序标准构建kd树,并通过前序遍历输出构建好的kd树,验证构建过程的正确性。
from MachineLearningImplementation.KNN.Node import KdNode
from MachineLearningImplementation.KNN.DataProcess import file2matrix, auto_norm
import numpy as np
import sys
import matplotlib.pyplot as plt
def create_kdtree(data_in, k, depth):
"""
根据给定的数据集,创建kdTree
:param data_in: 输入数据集
:param k: 表示存储的数据维度为k维
:param depth: 当前节点的深度
:return: 将已经创建好的kdTree的根节点root返回
"""
if data_in.shape[0] > 0: # 如果该数据区域中还有数据的话,继续划分
# 根据当前深度对应的列,对样本进行排序
# np.argsort函数返回经过排序后的索引
data_in = data_in[np.argsort(data_in[:, int(depth % k)])]
# 找到当前数据域中的中位数的数据,根据这个中位数样本进行划分
mid = data_in.shape[0] // 2
root = KdNode(data_in[mid, :]) # 得到当前数据域的中位数,也是当前子树的根节点中要存的数据样本
root.partition_dimension = depth % k # 数据的划分维度
# 深度加1,同时分别递归构建左右子树
depth = depth + 1
root.left = create_kdtree(data_in[:mid], k, depth)
root.right = create_kdtree(data_in[mid+1:], k, depth)
return root
elif data_in.shape[0] <= 0: # 如果当前数据域中已经没有元素,则直接返回
return None
def preorder_traversal(root, depth):
"""
前序遍历构建好的KdTree,用于测试构建过程是否成功
:param root: 当前子树的根节点
:param depth: 当前子树的深度
:return: None
"""
print(str(root.value) + ' : ' + str(root.partition_dimension) + ' : ' + str(depth))
if root.left is not None:
preorder_traversal(root.left, depth+1)
if root.right is not None:
preorder_traversal(root.right, depth+1)
3.2 基于kd树的最近邻算法实现
def find_closest_node(root, closest_point, x, min_dis, depth=0):
"""
找到当前预测点的最近邻点
:param root: 构造的kd tree中的一棵子树的根节点
:param closest_point: 与预测点最近的点
:param x: 不带标签的预测点
:param min_dis: 当前最短距离
:param depth: 当前遍历的深度,也是当前在数据样本中比较的维度
:return:
"""
if root is None:
return
# 先计算预测点与当前节点中存储的样本的欧氏距离
cur_distance = (sum((root.value[:-1] - x) ** 2)) ** 0.5
if min_dis[0] < 0 or cur_distance < min_dis:
depth += 1
min_dis[0] = cur_distance
# 注意赋值的时候不能直接赋值,要不按元素值赋值,要不用np.copy函数进行赋值,而为了使用可变类型的特性,我们选择值传递,这样在
# 子函数中改变可变类型变量的值,在子函数外访问该变量,值也会改变
#closest_point = np.copy(root.value)
for i in range(len(root.value)):
closest_point[i] = root.value[i]
# 递归查找叶节点,首先查找到预测点属于kd tree划分的哪个数据域
if x[root.partition_dimension] <= root.value[root.partition_dimension]:
find_closest_node(root.left, closest_point, x, min_dis, depth)
else:
find_closest_node(root.right, closest_point, x, min_dis, depth)
# 进行回溯,计算测试点和分割超平面的距离,如果相交则进入叶节点的右子树进行遍历查找最近点
distance = abs(x[root.partition_dimension] - root.value[root.partition_dimension])
if distance > min_dis[0]: # 如果当前分离超平面和预测点的距离小于当前最小点到预测点的距离,说明当前超平面的另一半区域内不存在距离更小的点,直接舍弃
return
elif distance <= min_dis[0]: # 说明相交,则需要遍历回溯回来的另一个子树
# 可以看到这里的遍历顺序和上面正好相反,因为是要往另一棵子树遍历,直到叶节点再往上回溯
if x[root.partition_dimension] <= root.value[root.partition_dimension]:
find_closest_node(root.right, closest_point, x, min_dis, depth)
elif x[root.partition_dimension] > root.value[root.partition_dimension]:
find_closest_node(root.left, closest_point, x, min_dis, depth)
# return min_dis, np.copy(closest_point)
return
if __name__ == '__main__':
"""
加载数据,得到训练集和测试集
"""
data = file2matrix('labeled_data.txt')
data = np.array(data) # 将数据类型由list转换为array
norm_dataset = auto_norm(data)
sys.setrecursionlimit(10000) # 设置最大递归深度为10000
train_set, test_set = norm_dataset[:-1], norm_dataset[-1] # 划分训练集和测试集
kd_tree = create_kdtree(train_set, train_set.shape[1] - 1, 0) # 构建kd tree
test_sample = test_set[:-1] # 得到不带标签的测试样本
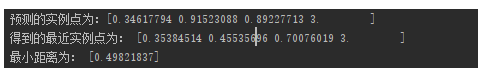
print("预测的实例点为:" + str(test_set))
"""
寻找最近邻点
"""
closest_point = np.zeros(4) # 初始化最近点
min_dis = np.array([-1.0]) # 先设置初始值为负
find_closest_node(kd_tree, closest_point, test_sample, min_dis)
print("得到的最近实例点为:", closest_point)
print("最小距离为:", min_dis)
输出结果为:
3.3 基于kd树的k近邻算法的实现
首先,必须明确的一点是,kd树中的k指的是数据的维度,而k近邻中的k指的是要选择k个近邻点,两者的意义是不同的。
从下面的代码中可以看出,k近邻算法其实只比最近邻算多了一个初始化和对k个候选点重排序的过程,即将递归过程中首先遇到的k个数据点作为k近邻点的初始化值,之后每次的更新其实只是更新当前的k近邻点中离目标点最远的那个待选点的值,然后对k个候选点重新排序选出新的距离最大的点用于下一轮更新,这和最近邻算法的过程其实是类似的。
def find_k_closest_nodes(root, closest_points, x, k):
"""
对构建的kd tree进行遍历,得到k个近邻点
:param root: 当前数据域对应子树的根节点
:param closest_points:
:param x: 不带标签的样本
:param k: 要选择k个邻近点
:return:
"""
if root is None:
return
# 计算预测点和当前节点的欧式距离
cur_distance = (sum((root.value[:-1] - x) ** 2)) ** 0.5
# 将closest_points中的点按最后一列的距离升序排序,不过一定要注意同样为了能在函数外访问这个元素已经改变了的可变对象,我们要进行值传递,而不是对象直接赋值
temp_points = closest_points[np.argsort(closest_points[:, -1])]
for i in range(len(closest_points)):
closest_points[i] = temp_points[i]
# 每次取最后一行元素进行操作,因为最后一行是距离最大的点,所以每次替换必定是替换掉这个点
if closest_points[k-1][-1] >= 10000 or closest_points[k-1][-1] > cur_distance:
closest_points[k-1][-1] = cur_distance
closest_points[k-1, :-1] = root.value # 这种替换值的方式
# 递归搜索叶节点
if x[root.partition_dimension] <= root.value[root.partition_dimension]:
find_k_closest_nodes(root.left, closest_points, x, k)
else:
find_k_closest_nodes(root.right, closest_points, x, k)
# 计算测试点和分割超平面之间的距离,如果相交则进入另一个叶节点进行搜索
distance = abs(x[root.partition_dimension] - root.value[root.partition_dimension])
if distance > closest_points[-1][-1]: # 如果不相交,则不用遍历另一棵子树
return
else: # 如果相交,则遍历回溯到当前节点的另外一棵子树
# 如果是这种情况,说明是从当前节点的左孩子节点回溯回来的,那我们还需要遍历右孩子节点
if x[root.partition_dimension] <= root.value[root.partition_dimension]:
find_k_closest_nodes(root.right, closest_points, x , k)
else:
find_k_closest_nodes(root.left, closest_points, x, k)
return
if __name__ == '__main__':
"""
加载数据,得到训练集和测试集
"""
data = file2matrix('labeled_data.txt')
data = np.array(data) # 将数据类型由list转换为array
norm_dataset = auto_norm(data)
sys.setrecursionlimit(10000) # 设置最大递归深度为10000
train_set, test_set = norm_dataset[:-1], norm_dataset[-1] # 划分训练集和测试集
kd_tree = create_kdtree(train_set, train_set.shape[1] - 1, 0) # 构建kd tree
test_sample = test_set[:-1] # 得到不带标签的测试样本
print("预测的实例点为:" + str(test_set))
"""
寻找k近邻点
"""
k = 3
# 这个array类型值中的最后一列为每个最近点到预测点的距离
closest_points = np.zeros((k, train_set.shape[1]+1))
closest_points[:, -1] = 10000.0
find_k_closest_nodes(kd_tree, closest_points, test_sample, k)
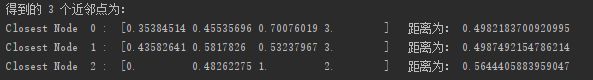
print("得到的", k, "个近邻点为:")
for i, point in enumerate(closest_points):
print("Closest Node ", i, ': ', point[:-1], ' 距离为:', point[-1])
输出结果为: