计算机视觉编程——图像聚类
文章目录
- 图像聚类
- 1 K-means聚类
- 1.1 SciPy聚类包
- 1.2 图像聚类
- 1.3 像素聚类
- 2 层次聚类
- 2.1 图像聚类
- 3 谱聚类
图像聚类
将物理或抽象对象的集合分成由类似的对象组成的多个类的过程被称为聚类。由聚类所生成的簇是一组数据对象的集合,这些对象与同一个簇中的对象彼此相似,与其他簇中的对象相异。聚类可以用于识别、划分图像数据集、组织和导航,以及对聚类后的图像进行相似性可视化。
1 K-means聚类
K-means聚类是一种将输入数据划分成k个簇的简单的聚类方法。其算法要反复提炼初始评估的类中心:
- 以随机或猜测的方式初始化类中心
- 将每个数据点归并到离它最近的类中心所属的类
- 对所有属于该类的数据点求平均,将平均值作为新的类中心
- 重复步骤2,3直到收敛
K-means算法最大的缺陷在于必须预先设定聚类数k,如果选择不恰当则会导致聚类出的结果很差。其优点是容易实现可以并行计算,并且对于很多别的问题不需要任何调整就能够直接使用。
1.1 SciPy聚类包
SciPy矢量量化包scipy.cluster.vq中有K-means的实现,下面是使用方法:
from scipy.cluster.vq import *
class1 = 1.5 * randn(100, 2)
class2 = randn(100, 2) + array([5, 5])
features = vstack((class1, class2))
centroids, variance = kmeans(features, 2)
code, distance = vq(features, centroids)
figure()
ndx = where(code == 0)[0]
plot(features[ndx, 0], features[ndx, 1], '*')
ndx = where(code == 1)[0]
plot(features[ndx, 0], features[ndx, 1], 'r')
plot(centroids[:, 0], centroids[:, 1], 'go')
axis('off')
show()
上面的代码生成两类二维正态分布数据,用k=2对这些数据进行聚类。由于SciPy中实现的K-means会计算若干次,并选择方差最小的结果,所以这里返回的方差并不是真正需要的。接下来使用SciPy包中的矢量量化函数对每个数据点进行归类。为了将其可视化,可以画出这些数据点以及最终的聚类中心。绘制的结果如下图所示:

1.2 图像聚类
使用来自字体数据集的fontinages的图像的前40个主成分进行投影,用投影系数作为作为每幅图像的向量描述。用pickle模块载入模型文件,在主成分上对图像进行投影,然后用下面的方法聚类:
import imtools
import pickle
from scipy.cluster.vq import *
imlist = imtools.get_imlist('selected_fontimages/')
imnbr = len(imlist)
with open('a_pca_modes.pkl', 'rb') as f:
immean = pickle.load(f)
V = pickle.load(f)
immatrix = array([array(Image.open(im)).flatten() for im in imlist], 'f')
immean = immean.flatten()
projected = array([dot(V[:40], immatrix[i] - immean) for i in range(imnbr)])
projected = whiten(projected)
centroids, distortion = kmean(projected, 4)
code, distance = vq(projected, centroids)
上述代码中的code变量中包含的是每幅图像属于哪个簇。设定聚类数为4,同时用SciPy的whiten()函数对数据“白化”处理,并进行归一化操作,使每个特征具有单位方差。利用下面的代码可视化聚类后的结果:
for k in range(4):
ind = where(code == k)[0]
figure()
gray()
for i in range(minimum(len(ind), 40)):
subplot(4, 10, i + 1)
imshow(immatrix[ind[i]].reshape((25, 25)))
axis('off')
show()
这里将每个簇显示在一个独立图像窗口中,且在该图形窗口中最多可以显示40幅图像。用Pylab的subplot()函数设定网格,聚成4类的可视化结果如图所示:

我们用PIL中的ImageDraw模块进行可视化:
h, w = 1200, 1200
img = Image.new('RGB', (w, h), (255, 255, 255))
draw = ImageDraw.Draw(img)
draw.line((0, h / 2, w, h / 2), fill = (255, 0, 0))
draw.line((w / 2, 0, w / 2, h), fill = (255, 0, 0))
scale = abs(projected).max(0)
scaled = floor(array([(p / scale) * (w / 2 - 20, h / 2 - 20) + (w / 2, h / 2) for p in projected]))
for i in range(imnbr):
nodeim = Image.open(imlist[i])
nodeim.thumbnail((25, 25))
ns = nodeim.size
box = (int(scaled[i][0] - ns[0] // 2), int(scaled[i][1] - ns[1] // 2),
int(scaled[i][0] + ns[0] // 2 + 1), int(scaled[i][1] + ns[1] // 2 + 1))
img.paste(nodeim, box)
img.save('pca_font.jpg')
这个图像说明了,这些字体图像在40维里的分布情况,二维投影后相似的字体图像距离较近。
1.3 像素聚类
将图像区域或像素合并成有意义的部分称为图像分割。除了在一些简单的图像上,单纯在像素水平上应用K-means得出的结论往往是毫无意义的。要产生有意义的结果,往往需要更复杂的类模型而非平均像素色彩或空间一致性。
下面的代码示例载入一幅图像,用一个步长为steps的方形网格在图像上滑动,每滑一次对网格中图像区域像素求平均值,将其作为新生成的低分辨率图像对应位置处的像素值,并用K-means进行聚类:
from scipy.cluster.vq import *
from scipy.misc import imresize
steps = 50
im = array(Image.open('jimei_grey.jpg'))
dx = im.shape[0]
dy = im.shape[1]
features = []
for x in range(steps):
for y in range(steps):
R = mean(im[x * dx: (x + 1) * dx, y * dy: (y + 1) * dy, 0])
G = mean(im[x * dx: (x + 1) * dx, y * dy: (y + 1) * dy, 1])
B = mean(im[x * dx: (x + 1) * dx, y * dy: (y + 1) * dy, 2])
features.append([R, G, B])
features = array(features, 'f')
centroids, variance = kmeans(features, 3)
code, distance = vq(features, centroids)
codeim = code.reshape(steps, steps)
codeim = imresize(codeim, im.shape[:2], interp = 'nearest')
figure()
imshow(codeim)
show()
K-means的输入是一个有steps*steps行的数组,数组的每一行有3列,各列分别为区域块R、G、B三个通道的像素平均值。为可视化最后的结果,我们用Scipy的imresize()函数在原图像坐标中显示这幅图像。参数interp指定插值方法采用最近邻差值,以便在类间进行变换时不需要引入新的像素值。结果如下图所示:

2 层次聚类
层次聚类是另一种简单但有效的聚类算法,其思想是基于样本间成对距离建立一个简相似性树。该算法首先将特征向量距离最近的两个样本归并为一组,并在树中创建一个“平均”节点,将这两个距离最近的样本作为该“平均”节点的下的儿子节点。然后在剩下的包含任意平均节点的样本中寻找下一个最近的对,重复进行前面的操作。在每一个节点处保存了两个子节点之间的距离。遍历整个树,通过设定的阈值,遍历过程可以在比阈值大的节点位置终止,从而提取出聚类簇。
层次聚类有若干优点。例如,利用树结构可以可视化数据间的关系,并显示这些簇是如何关联的。在树中,一个好的特征向量可以给出一个很好的分离结果。另外一个优点是,对于给定的不同阈值,可以直接利用原来的树而不需要重新计算。不足之处是,对于实际需要的聚类簇需要给出一个合适的阈值。
from itertools import combinations
class ClusterNode(object):
def __init__(self,vec,left,right,distance=0.0,count=1):
self.left = left
self.right = right
self.vec = vec
self.distance = distance
self.count = count
def extract_clusters(self,dist):
""" Extract list of sub-tree clusters from
hcluster tree with distance
if self.distance < dist:
return [self]
return self.left.extract_clusters(dist) + self.right.extract_clusters(dist)
def get_cluster_elements(self):
""" Return ids for elements in a cluster sub-tree. """
return self.left.get_cluster_elements() + self.right.get_cluster_elements()
def get_height(self):
""" Return the height of a node,
height is sum of each branch. """
return self.left.get_height() + self.right.get_height()
def get_depth(self):
""" Return the depth of a node, depth is
max of each child plus own distance. """
return max(self.left.get_depth(), self.right.get_depth()) + self.distance
class ClusterLeafNode(object):
def __init__(self,vec,id):
self.vec = vec
self.id = id
def extract_clusters(self,dist):
return [self]
def get_cluster_elements(self):
return [self.id]
def get_height(self):
return 1
def get_depth(self):
return 0
def draw(self,draw,x,y,s,imlist,im):
nodeim = Image.open(imlist[self.id])
nodeim.thumbnail([20,20])
ns = nodeim.size
im.paste(nodeim,[int(x),int(y-ns[1]//2),int(x+ns[0]),int(y+ns[1]-ns[1]//2)])
def L2dist(v1,v2):
return sqrt(sum((v1-v2)**2))
def L1dist(v1,v2):
return sum(abs(v1-v2))
def hcluster(features,distfcn=L2dist):
distances = {}
node = [ClusterLeafNode(array(f),id=i) for i,f in enumerate(features)]
while len(node)>1:
closest = float('Inf')
for ni,nj in combinations(node,2):
if (ni,nj) not in distances:
distances[ni,nj] = distfcn(ni.vec,nj.vec)
d = distances[ni,nj]
if d<closest:
closest = d
lowestpair = (ni,nj)
ni,nj = lowestpair
new_vec = (ni.vec + nj.vec) / 2.0
new_node = ClusterNode(new_vec,left=ni,right=nj,distance=closest)
node.remove(ni)
node.remove(nj)
node.append(new_node)
return node[0]
import hcluster
from numpy.random import randn
from pylab import *
from PIL import Image
class1 = 1.5 * randn(100, 2)
class2 = randn(100, 2) + array([5, 5])
features = vstack((class1, class2))
tree = hcluster.hcluster(features)
clusters = tree.extract_clusters(5)
print('number of clusters', len(clusters))
for c in clusters:
print(c.get_cluster_elements())
理想情况下应该得到一个聚类簇,但是在实际数据中,可能会得到三类或者更多。这主要依赖于实际生成的二维数据。
2.1 图像聚类
import os
import hcluster
path = 'flickr-sunsets/'
imlist = [os.path.join(path, f) for f in os.listdir(path) if f.endswith('.jpg')]
features = zeros([len(imlist), 512])
for i, f in enumerate(imlist):
im = array(Image.open(f))
h, edges = histogramdd(im.reshape(-1, 3), 8, normed = True, range = [(0, 255), (0, 255), (0, 255)])
features[i] = h.flatten()
tree = hcluster.hcluster(features)
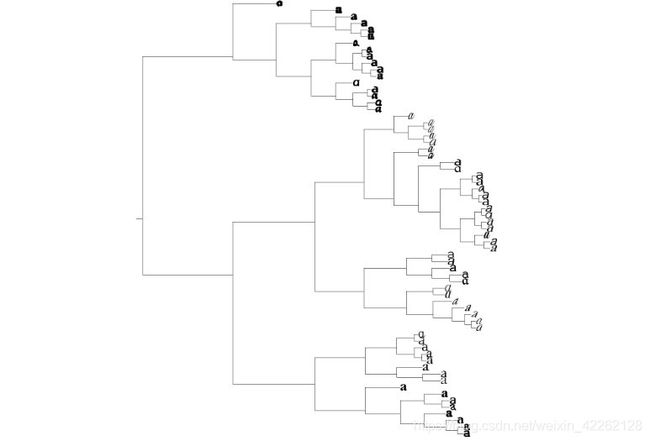
将R、G、B三个颜色通道作为特征向量,将其传递到Numpy的histogramdd()中。该函数能够计算多维直方图。为了可视化聚类树,可以画出树状图,这样在判定给出的描述子向量好坏以及在特征场合考虑什么是相似的时候提供有用的信息。
tree = hcluster.hcluster(projected)
hcluster.draw_dendrogram(tree, imlist, filename = 'fonts.jpg')
clusters = tree.extract_clusters(0.23 * tree.distance)
for c in clusters:
elements = c.get_cluster_elements()
nbr_elements = len(elements)
if nbr_elements > 3:
figure()
for p in range(minimum(nbr_elements, 20)):
subplot(4, 5, p + 1)
im = array(Image.open(imlist[elements[p]]))
imshow(im)
axis('off')
show()
3 谱聚类
谱聚类与K-means和层次聚类方法截然不同。
对于n个元素,相似矩阵是一个n*n的矩阵,矩阵每个元素表示两两之间的相似性分数。谱聚类是由相似性矩阵构建谱矩阵而得名的。对该谱矩阵进行特征分解得到的特征向量可以用于降维,然后聚类。其优点在于仅需输入相似性矩阵,并且可以采用任意度量方式构建相似性矩阵。
n = len(projected)
S = array([[sqrt(sum((projected[i] - projected[j]) ** 2)) for i in range(n)] for j in range(n)], 'f')
rowsum = sum(S, axis = 0)
D = diag(1 / sqrt(rowsum))
I = identity(n)
L = I - dot(D, dot(S, D))
U, sigma, V = linalg.svd(L)
k = 5
features = array(V[:k]).T
features = whiten(features)
centroids,distortion = kmeans(projected,k)
code,distance = vq(projected,centroids)
for c in range(k):
ind = where(code==c)[0]
figure()
for i in range(minimum(len(ind),39)):
im = Image.open('selected_fontimages/' + imlist[ind[i]])
subplot(4,10,i+1)
imshow(array(im))
axis('equal')
axis('off')
show()
上述代码中使用两两间的欧式距离创建矩阵S,并对k个特征向量用常规的K-means进行聚类,最后绘制出这些聚类簇。