聚类算法(k_means)
聚类算法
- 1 聚类算法分类
- 2 k-means
-
- 2.1 算法描述
- 2.2 优缺点
- 2.3 k-means算法调优及改进
-
- 2.3.1 K均值算法调优
- 2.3.2 K均值算法改进
-
- 克服算法收敛于局部最小值问题的K-means算法
- K-Means++:初始值选择的改进
- elkan K-Means:距离计算优化
- Mini Batch K-Means:大样本优化
- K-Medians:异常值影响
- ISODATA算法:K值的大小不确定
- 2.4 确定合理的K值
-
- 2.4.1 肘部法则
- 2.4.2 Gap Statistic
- 2.5 EM算法证明 K-means 算法的收敛性
- 2.6 K-means 附加问题
- 2.7 K-means 在 sklearn方法
- 3 轮廓图定量分析聚类质量
聚类:
- 通过一定的算法将原始数据划分成多个数据簇
- 没有预先定义好的类别
- 同一簇内部数据样本之间有很大的相似性,不同簇内部的数据相似性很小
相似度与距离度量:
定义距离来度量表示相似度:欧式距离,曼哈顿距离( D i j = ∣ x i − x j ∣ + ∣ y i − y j ∣ D_{ij}=|x_i-x_j|+|y_i-y_j| Dij=∣xi−xj∣+∣yi−yj∣),闵可夫斯基距离(n次欧式距离),切比雪夫距离( D i j = m a x ∣ x i − x j ∣ D_{ij}=max|x_i-x_j| Dij=max∣xi−xj∣)
1 聚类算法分类
1.基于划分
给定一个有N个元组或者纪录的数据集,分裂法将构造K个分组,每一个分组就代表一个聚类,K
算法:K-means算法、K-medians算法、CLARANS算法
2.基于层次
对给定的数据集进行层次似的分解,直到某种条件满足为止。具体又可分为“自底向上”和“自顶向下”两种方案。
特点:较小的计算开销。然而这种技术不能更正错误的决定。
算法:BIRCH算法、CURE算法、CHAMELEON算法
3.基于密度
只要一个区域中的点的密度大过某个阈值,就把它加到与之相近的聚类中去。
特点:能克服基于距离的算法只能发现“类圆形”的聚类的缺点。
算法:DBSCAN算法、OPTICS算法、DENCLUE算法
4.基于网格
将数据空间划分成为有限个单元(cell)的网格结构,所有的处理都是以单个的单元为对象的。
特点:处理速度很快,通常这是与目标数据库中记录的个数无关的,只与把数据空间分为多少个单元有关。
算法:STING算法、CLIQUE算法、WAVE-CLUSTER算法
硬聚类和软聚类
硬聚类(hard clustering)是指数据集中的样本只能划分到一个簇中,如k-means算法。软聚类(soft clustering)或模糊聚类(fuzzy clustering)可以将一个样本划分到多个不同的簇中,如C-means(FCM)算法,FCM的计算步骤与k-means相似,只是FCM是使用样本属于不同簇的概率来代替k-means中的类标。样本属于不同簇的概率之和为1。
2 k-means
K-Means算法的思想很简单,对于给定的样本集,按照样本之间的距离大小,将样本集划分为K个簇。让簇内的点尽量紧密的连在一起,而让簇间的距离尽量的大。
2.1 算法描述

迭代收敛定义:
(1)聚类中心不再有变化
(2)每个样本到对应聚类中心的距离之和不再有很大变化
损失函数:
假定 u 1 , u 2 , . . . , u k u_1,u_2,...,u_k u1,u2,...,uk 为K个聚类中心,用 r n k ∈ 0 , 1 r_{nk}\in{0,1} rnk∈0,1 表示 x n x_n xn 是否属于聚类K,则损失函数定义如下(簇内误差平方和SSE): S S E = J ( u , r ) = ∑ n = 1 N ∑ k = 1 K r n k ∣ x n − u k ∣ 2 SSE = J(u,r) = \sum_{n=1}^N \sum_{k=1}^K r_{nk}|x_n-u_k|^2 SSE=J(u,r)=n=1∑Nk=1∑Krnk∣xn−uk∣2由于SSE是一个非凸函数(non-convex function),所以SSE不能保证找到全局最优解,只能确保局部最优解。但是可以重复执行几次kmeans,选取SSE最小的一次作为最终的聚类结果。
2.2 优缺点
K-means算法优点:
- 计算复杂度低,为O(Nmq),其中N是数据总量,m是类别(即k),q是迭代次数
- 原理简单,实现容易,容易解释,收敛速度快
- 聚类效果不错
K-means算法缺点:
- 对噪音和异常点比较的敏感,可以通过一些调整(如中心值不直接取均值,而是找均值最近的样本点代替)
- 需要提前确定K值(提前确定多少类)
- 分类结果依赖于分类中心的初始化(可以通过进行多次K-means取最优来解决)
- 属于“硬聚类”,每个样本只能有一个类别。其他的一些聚类方法(GMM或者模糊K-means允许“软聚类”)
- 对于团状的数据点集区分度好,对于带状(环绕)等非凸形状不太好,不是凸的数据集比较难收敛。(用谱聚类或做特征映射)
- 采用迭代方法,得到的结果只是局部最优
- 如果各隐含类别的数据不平衡,比如各隐含类别的数据量严重失衡,或者各隐含类别的方差不同,则聚类效果不佳。
注意:由于k-means算法是基于欧式距离来计算的,所以k-means算法对于数据的范围比较敏感,所以在使用k-means算法之前,需要先对数据进行标准化,保证k-means算法不受特征量纲的影响。
2.3 k-means算法调优及改进
2.3.1 K均值算法调优
K均值算法的调优一般可以从以下几个角度出发。
- 对数据进行归一化和对离群点进行处理(利用损失函数SSE对离群点进行处理)
(1)对于所有的簇,某变量的SSE都很低,都意味着什么?说明该属性本质上为常量,不能作为聚类依据。
(2)如果只对一个簇很低,意味着什么?那么该属性有助于该簇的定义
(3)如果只对一个簇很高,意味着什么?那么该属性为噪声属性
(4)如果对所有簇都很高,意味着什么?那么该属性 与 定义该属性提供的信息不一致,也意味着该属性不利于簇的定义。
(5)如何使用每个变量的SSE信息改进聚类?消除对于所有簇都是低的SSE(高的SSE)的属性。因为这些属性对聚类没有帮助,这些属性在SSE的总和计算中引入了噪声。也可以对其中某些属性用加权概率来计算,使该属性有助于该簇的定义, 去除某些不利于该簇定义的影响因子(那些可能是噪声)。从而更有利于簇的聚类。 - 合理选择K值(2.4)
- 采用核函数:传统的欧式距离度量方式,使得K均值算法本质上假设了各个数据簇的数据具有一样的先验概率,并呈现球形或者高维球形分布,这种分布在实际生活中并不常见。面对非凸的数据分布形状时,可能需要引入核函数来优化,这时算法又称为核K均值算法,是核聚类方法的一种。核聚类方法的主要思想是通过一个非线性映射,将输入空间中的数据点映射到高位的特征空间中,并在新的特征空间中进行聚类。非线性映射增加了数据点线性可分的概率,从而在经典的聚类算法失效的情况下,通过引入核函数可以达到更为准确的聚类结果。
2.3.2 K均值算法改进
克服算法收敛于局部最小值问题的K-means算法
- 将所有点看成一个簇
- 当簇数目小于k时,对于每一个簇
- 计算总误差
- 在给定的簇上面进行k-均值聚类(k=2)(也可以选择SSE最大的簇进行划分)
- 计算将该簇一分为二之后的总误差
- 选择使得总误差最小的簇进行划分
K-Means++:初始值选择的改进
K-Means算法需要随机选择初始化的中心点,如果中心点选择不合适,可能会导致簇的效果不好或产生收敛速度慢等问题。解决这个问题一个比较合适的方法就是,在数据集上多次运行K-Means算法,根据簇内误差平方和(SSE)来选择性能最好的模型。除此之外,还可以通过K-Means++算法,让初始的中心点彼此的距离尽可能的远,相比K-Means算法,它能够产生更好的模型。
K-Means++有下面几个步骤组成:
- 初始化一个空的集合M,用于存储选定的k个中心点
- 从输入的样本中随机选择第一个中心点μ,并将其加入到集合M中
- 对于集合M之外的任意样本点x,计算每个样本与当前已有聚类中心的最短距离
- 使用加权概率分布来随机来随机选择下一个中心点μ (距离当前n个聚类中心越远的点会有更高的概率被选为第n+1个聚类中心)
- 重复步骤2和3,直到选定k个中心点
- 基于选定的中心点执行k-means算法
elkan K-Means:距离计算优化
elkan K-Means算法的目标是减少不必要的距离的计算,利用了两边之和大于等于第三边,以及两边之差小于第三边的三角形性质,来减少距离的计算。
- 第一种规律是对于一个样本点x和两个质心 μ 1 , μ 2 μ_1,μ_2 μ1,μ2。如果我们预先计算出了这两个质心之间的距离 D ( u 1 , u 2 ) D(u_1,u_2) D(u1,u2),则如果计算发现 2 D ( x , u 1 ) ≤ D ( u 1 , u 2 ) 2D(x,u_1)≤D(u_1,u_2) 2D(x,u1)≤D(u1,u2) ,我们立即就可以知道 D ( x , u 1 ) ≤ D ( x , u 2 ) D(x,u_1)≤D(x,u_2) D(x,u1)≤D(x,u2)。此时我们不需要再计算 D ( x , u 2 ) D(x,u_2) D(x,u2),也就是说省了一步距离计算。
- 第二种规律是对于一个样本点x和两个质心 u 1 , u 2 u_1,u_2 u1,u2。我们可以得到 D ( x , u 2 ) ≥ m a x ( 0 , D ( x , u 1 ) − D ( u 1 , u 2 ) ) D(x,u_2)≥max(0,D(x,u_1)−D(u_1,u_2)) D(x,u2)≥max(0,D(x,u1)−D(u1,u2))。这个从三角形的性质也很容易得到。
利用上边的两个规律,elkan K-Means比起传统的K-Means迭代速度有很大的提高。但是如果我们的样本的特征是稀疏的,有缺失值的话,这个方法就不使用了,此时某些距离无法计算,则不能使用该算法。
Mini Batch K-Means:大样本优化
如果样本量非常大,比如达到10万以上,特征有100以上,此时用传统的K-Means算法非常的耗时,就算加上elkan K-Means优化也依旧。Mini Batch,也就是用样本集中的一部分的样本来做传统的K-Means(一般是通过无放回的随机采样得到的),这样可以避免样本量太大时的计算难题,算法收敛速度大大加快。当然此时的代价就是我们的聚类的精确度也会有一些降低。一般来说这个降低的幅度在可以接受的范围之内。为了增加算法的准确性,我们一般会多跑几次Mini Batch K-Means算法,用得到不同的随机采样集来得到聚类簇,选择其中最优的聚类簇。
K-Medians:异常值影响
K-Medians是K-Means的一种变体,是用数据集的中位数而不是均值来计算数据的中心点。
K-Medians的优势是使用中位数来计算中心点不受异常值的影响;缺点是计算中位数时需要对数据集中的数据进行排序,速度相对于K-Means较慢。
ISODATA算法:K值的大小不确定
当K值的大小不确定时,可以使用ISODATA算法,全称是迭代自组织数据分析法。ISODATA算法的思想很直观,当属于某个类别的样本数过少时,把该类别去除;当属于某个类别的样本数过多、分散程度较大时,把该类别分为两个子类别。ISODATA算法在K均值算法的基础之上增加了两个操作,一是分裂操作,对应着增加聚类中心数;二是合并操作,对应着减少聚类中心数。ISODATA算法是一个比较常见的算法,其缺点是需要指定的参数比较多,不仅仅需要一个参考的聚类数量K,还需要制定3个阈值。下面介绍ISODATA算法的各个输入参数。
- 预期的聚类中心数目K。在ISODATA运行过程中聚类中心数可以变化,K是一个用户指定的参考值,该算法的聚类中心数目变动范围也由其决定。具体地,最终输出的聚类中心数目常见范围是从K的一半,到两倍K。
- 每个类所要求的最少样本数目 N m i n N_{min} Nmin。如果分裂后会导致某个子类别所包含样本数目小于该阈值,就不会对该类别进行分裂操作。
- 最大方差 Σ \Sigma Σ。用于控制某个类别中样本的分散程度。当样本的分散程度超过这个阈值时,且分裂后满足聚类中心范围,进行分裂操作。
- 两个聚类中心之间所允许最小距离 D m i n D_{min} Dmin。如果两个类靠得非常近(即这两个类别对应聚类中心之间的距离非常小),小于该阈值时,则对这两个类进行合并操作。
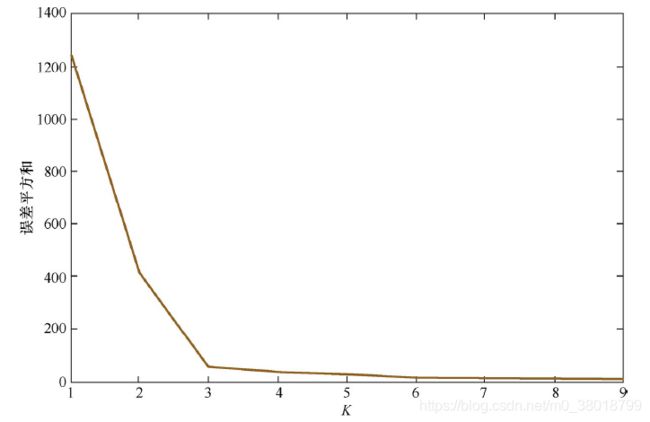
2.4 确定合理的K值
2.4.1 肘部法则
肘部法则(Elbow method):找到随着K值变大,损失函数的拐点。
2.4.2 Gap Statistic
手肘法是一个经验方法,缺点就是不够自动化。Gap Statistic方法的优点是,不再需要肉眼判断,而只需要找到最大的Gap statistic所对应的K即可,因此该方法也适用于批量化作业。在这里我们继续使用上面的损失函数,当分为K簇时,对应的损失函数记为 D k D_k Dk。Gap Statistic定义为 G a p ( K ) = E ( l o g D k ) − l o g D k Gap(K)=E(logD_k)−logD_k Gap(K)=E(logDk)−logDk 其中 E ( l o g D k ) E(logD_k) E(logDk) 是 l o g D k logD_k logDk 的期望,一般通过蒙特卡洛模拟产生。我们在样本所在的区域内按照均匀分布随机地产生和原始样本数一样多的随机样本,并对这个随机样本做K均值,得到一个 D k D_k Dk;重复多次就可以计算出 E ( l o g D k ) E(logD_k) E(logDk)的近似值。
那么Gap(K)有什么物理含义呢?它可以视为随机样本的损失与实际样本的损失之差。试想实际样本对应的最佳簇数为K,那么实际样本的损失应该相对较小,随机样本损失与实际样本损失之差也相应地达到最小值,从而Gap(K)取得最大值所对应的K值就是最佳的簇数。
2.5 EM算法证明 K-means 算法的收敛性
2.6 K-means 附加问题
- k值如何确定:
(1) 肘部法则
(2) 从较小值开始枚举,并计算当前k的平均轮廓系数,最后选取轮廓系数最大的值对应的k作为最终的集群数目。
(3) ISODATA算法针对这个问题进行了改进:当属于某个类别的样本数过少时把这个类别去除,当属于某个类别的样本数过多、分散程度较大时把这个类别分为两个子类别(类的自动合并和分裂) - 初始随机种子如何确定:
(1) K-means++
(2) 采用遗传算法(GA)进行初始化,以内部聚类准则作为评价指标 - 度量方式
传统K-means采用欧式距离进行样本间的相似度度量,显然并不是所有的数据集都适用于这种度量方式。kernel k-means参照支持向量机中核函数的思想,将所有样本映射到另外一个特征空间中再进行聚类,就有可能改善聚类效果 - 空簇
如果所有的点在指派步骤都未分配到某个簇,某个簇就会变成空簇。如果这种情况发生,则需要某种策略来选择一个替补聚类中心,否则的话,平方误差将会偏大(算法的目的就是使各个样本与所在类均值的误差平方和达到最小,这也是评价K-means算法最后聚类效果的评价标准)。一种方法是选择一个距离当前任何质心最远的点。这将消除当前对总平方误差影响最大的点 - 降低SSE
将大的分散的簇再次拆开;引入新的簇将之前的大簇拆分。 那么如何在保持簇数目不变的情况下提高簇的质量呢?将具有最大SSE值得簇划分为两个簇(因为,SSE最大的簇一般情况下,意味着簇内的数据点距离簇中心较远),具体地,可以将最大簇包含的点过滤出来并在这些点上运行K-means算法,其中k设为2。接着合并两个簇,一方面我们可以合并两个最近的质心所对应的簇,即计算所有质心之间的距离,合并质心距离最近的两个质心所对应的簇。另一方面,我们可以合并两个使得SSE增幅最小的簇,显然,合并两个簇之后SSE的值会有所上升,那么为了最好的聚类效果,应该尽可能使总的SSE值小,所以就选择合并两个簇后SSE涨幅最小的簇。具体地,就是计算合并任意两个簇之后的总得SSE,选取合并后最小的SSE对应的两个簇进行合并。这样,就可以满足簇的数目不变。 - 增量更新质心
再次在质心附近寻找测试点,看能否再次找到更优的质心。 - 噪声处理
k-means对离群值非常敏感,算法目标是簇内差异最小化,即SSE最小,所以单个噪音点也可以对整个簇造成很大的扰动,常用解决办法:
2.7 K-means 在 sklearn方法
sklearn.cluster.KMeans(
n_clusters = 8, #聚类个数,K值,默认8
init = 'k-means++',
n_init = 10,
max_iter = 300,
tol = 0.0001,
precompute_distances = 'auto',
verbose = 0,
random_state = None,
copy_x = True,
n_jobs = 1,
algorithm = 'auto'
)
# 一些重要的参数:
n_clusters = 8, #聚类个数,K值,默认8
init = 'k-means++', #初始化类中心点选择方法,可选:
{
'k-means++', #是一种优化选择方法,比较容易收敛
'random', #随机选择
an ndarray #可以通过输入ndarray数组手动指定中心点
}
max_iter: #最大迭代数
precompute_distances: #预计算距离,计算速度更快但占用更多内存。auto True
copy_x # True,原始数据不变,False直接在原始数据上做更改
3 轮廓图定量分析聚类质量
轮廓分析(silhouette analysis),使用图形工具来度量簇中样本的聚集程度,除k-means之外也适用于其他的聚类算法。通过三个步骤可以计算出当个样本的轮廓系数(silhouette coefficient)
1、将样本x与簇内的其他点之间的平均距离作为簇内的内聚度a
2、将样本x与最近簇中所有点之间的平均距离看作是与最近簇的分离度b
3、将簇的分离度与簇内聚度之差除以二者中比较大的数得到轮廓系数,计算公式如下: S i = b i − a i m a x ( b i , a i ) S^i = \frac{b^i-a^i}{max(b^i,a^i)} Si=max(bi,ai)bi−ai轮廓系数的取值在-1到1之间。当簇内聚度与分度离相等时,轮廓系数为0。当b>>a时,轮廓系数近似取到1,此时模型的性能最佳。计算所有x的轮廓系数,求出平均值即为当前聚类的整体轮廓系数