dandanwu90
2019年4月11日
不自我检测怎么知道我什么都不会?
把我盘倒的R语言中级10个题目在这里。
Q1:
根据R包org.Hs.eg.db找到下面ensembl 基因ID 对应的基因名(symbol)
ENSG00000000003.13
ENSG00000000005.5
ENSG00000000419.11
ENSG00000000457.12
ENSG00000000460.15
ENSG00000000938.11
提示:
library(org.Hs.eg.db)
g2s=toTable(org.Hs.egSYMBOL)
g2e=toTable(org.Hs.egENSEMBL)
suppressMessages(library(org.Hs.eg.db))
#查看包里面的内容
keytypes(org.Hs.eg.db)
columns(org.Hs.eg.db)
g2s=toTable(org.Hs.egSYMBOL)
g2e=toTable(org.Hs.egENSEMBL)
head(g2s)
head(g2e)
ensembl_id=c("ENSG00000000003.13", "ENSG00000000005.5","ENSG00000000419.11","ENSG00000000457.12","ENSG00000000460.15","ENSG00000000938.11")
ensembl_id=as.data.frame(ensembl_id)
library(stringr)
ensembl_id=str_split(ensembl_id$ensembl_id,pattern ="[.]",simplify = T)[,1]
ensembl_id=as.data.frame(ensembl_id)
b=merge(ensembl_id,g2e,by='ensembl_id',all.x=T)
d=merge(b,g2s,by="gene_id",all.x=T)
Q2:
根据R包hgu133a.db找到下面探针对应的基因名(symbol)
1053_at
117_at
121_at
1255_g_at
1316_at
1320_at
1405_i_at
1431_at
1438_at
1487_at
1494_f_at
1598_g_at
160020_at
1729_at
177_at
提示:
library(hgu133a.db)
ids=toTable(hgu133aSYMBOL)
head(ids)
suppressMessages(library(hgu133a.db))
keytypes(hgu133a.db)
ids=toTable(hgu133aSYMBOL)
a=read.csv(file='probe_id',header = F)
colnames(a)='probe_id'
mydata=merge(a,ids,by="probe_id",all.x=T)
Q3:
找到R包CLL内置的数据集的表达矩阵里面的TP53基因的表达量,并且绘制在 progres.-stable分组的boxplot图
提示:
suppressPackageStartupMessages(library(CLL))
data(sCLLex)
sCLLex
exprSet=exprs(sCLLex)
library(hgu95av2.db)
想想如何通过 ggpubr 进行美化。
suppressPackageStartupMessages(library(CLL))
data("sCLLex")
exprSet=as.data.frame(exprs(sCLLex))
pd=pData(sCLLex)
library(hgu95av2.db)
keytypes(hgu95av2.db)
gene2s=toTable(hgu95av2SYMBOL)
gene2s_filter=gene2s[gene2s$symbol=='TP53',]
exprSet$probe_id=rownames(exprSet)
exprSet2_filter=merge(gene2s_filter,exprSet,by='probe_id',all.x=T)
rownames(exprSet2_filter)=exprSet2_filter[,1]
exprSet2_filter=exprSet2_filter[,c(-1,-2)]
exprSet2_filter=as.data.frame(t(exprSet2_filter))
exprSet2_filter$Disease=pd$Disease
library(reshape)
exprSet2=melt(exprSet2_filter,id='Disease')
library(ggplot2)
ggplot(exprSet2, aes(x=variable, y=value, fill = Disease))+
geom_boxplot(position=position_dodge(1))+
geom_dotplot(binaxis='y', stackdir='center',
position=position_dodge(1),binwidth =0.05)
Q4:
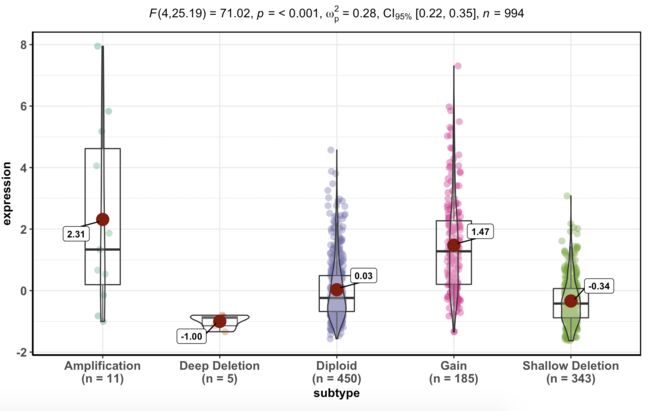
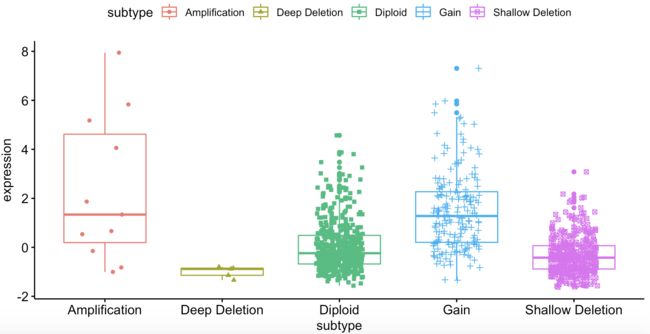
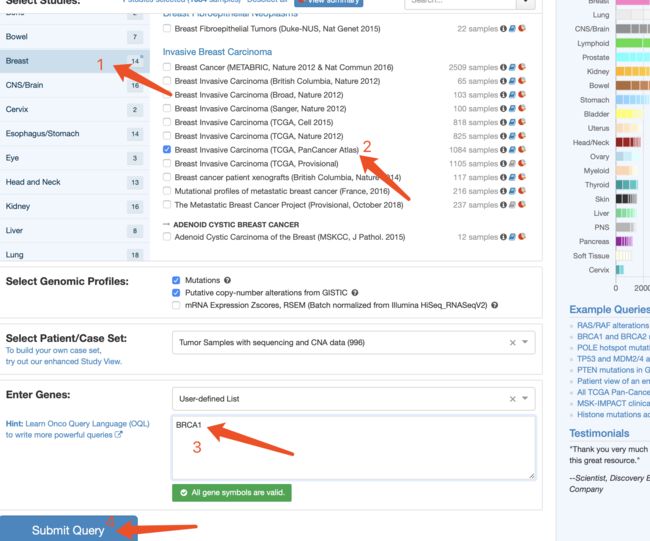
找到BRCA1基因在TCGA数据库的乳腺癌数据集(Breast Invasive Carcinoma (TCGA, PanCancer Atlas))的表达情况
提示:
使用http://www.cbioportal.org/index.do 定位数据集:http://www.cbioportal.org/datasets
a=read.csv(file='plot4.txt',sep="\t",header = T)
colnames(a)
colnames(a)=c("id","subtype","expression","mutant")
library("ggstatsplot")
ggbetweenstats(
data = a,
x = 'subtype',
y = 'expression')
library("ggpubr")
ggboxplot(data =a, x = 'subtype', y = 'expression',
color = "subtype",
add = "jitter", shape = "subtype")
数据网址在这里
步骤如下:Q4_1, Q4_2
Q5:
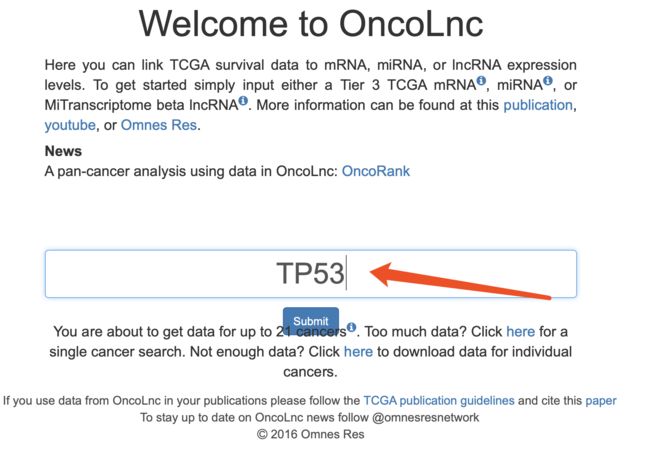
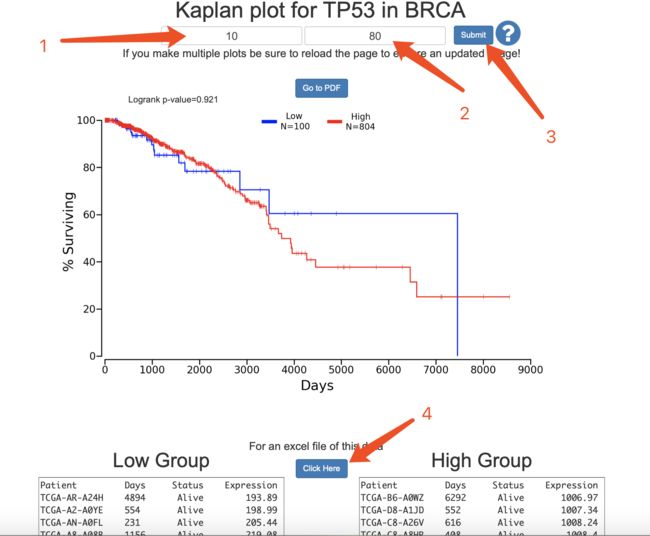
找到TP53基因在TCGA数据库的乳腺癌数据集的表达量分组看其是否影响生存
提示使用:http://www.oncolnc.org/
BRCA_7157_10_80=read.csv(file ='BRCA_7157_10_80.csv',header = T )
colnames(BRCA_7157_10_80)
library(ggstatsplot)
ggbetweenstats(
data = BRCA_7157_10_80,
x = 'Status',
y = 'Expression')
数据网址在这里
步骤如下:Q5_1,Q5_2
Q6:
下载数据集GSE17215的表达矩阵并且提取下面的基因画热图
ACTR3B ANLN BAG1 BCL2 BIRC5 BLVRA CCNB1 CCNE1 CDC20 CDC6 CDCA1 CDH3 CENPF CEP55 CXXC5 EGFR ERBB2 ESR1 EXO1 FGFR4 FOXA1 FOXC1 GPR160 GRB7 KIF2C KNTC2 KRT14 KRT17 KRT5 MAPT MDM2 MELK MIA MKI67 MLPH MMP11 MYBL2 MYC NAT1 ORC6L PGR PHGDH PTTG1 RRM2 SFRP1 SLC39A6 TMEM45B TYMS UBE2C UBE2T
提示:
根据基因名拿到探针ID,缩小表达矩阵绘制热图,没有检查到的基因直接忽略即可。
suppressMessages(library(GEOquery))
Q6=getGEO("GSE17215",AnnotGPL = F,getGPL = F)
show(Q6)
Series_m=Q6$GSE17215_series_matrix.txt.gz
Series_m=as.data.frame(exprs(Series_m))
head(Series_m)
dim(Series_m)
suppressMessages(library(hgu133a.db))
keytypes(hgu133a.db)
ids=toTable(hgu133aSYMBOL)
#加戏疑问:如果不过滤没有检测到的基因,那么统计有多少个基因没有检测到
Q6_gene=read.csv(file="Q6.txt",sep="\t",header = F)
colnames(Q6_gene)="symbol"
Q6_mydata=merge(Q6_gene,ids,by="symbol")
Series_m$probe_id=rownames(Series_m)
Series_m_filter=merge(Q6_mydata,Series_m,by="probe_id")
rownames(Series_m_filter)=Series_m_filter[,1]
Series_m_filter=Series_m_filter[,c(-1,-2)]
library(pheatmap)
n=t(scale(t( Series_m_filter ))) #scale()函数去中心化和标准化
#对每个探针的表达量进行去中心化和标准化
n[n>2]=2 #矩阵n中归一化后,大于2的项,赋值使之等于2(相当于设置了一个上限)
n[n< -2]= -2 #小于-2的项,赋值使之等于-2(相当于设置了一个下限)
n[1:4,1:4]
pheatmap(n,show_rownames=F,clustering_distance_rows = "correlation")
Q7:
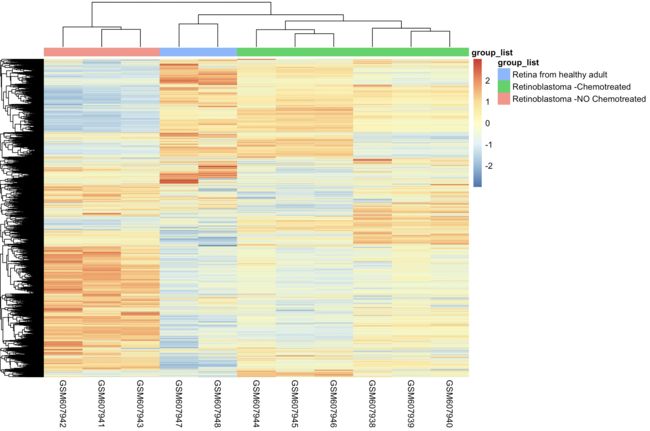
下载数据集GSE24673的表达矩阵计算样本的相关性并且绘制热图,需要标记上样本分组信息
suppressMessages(library(GEOquery))
Q7=getGEO("GSE24673",AnnotGPL = F,getGPL = F)
show(Q7)
Q7_m=Q7$GSE24673_series_matrix.txt.gz
Q7_exprs=as.data.frame(exprs(Q7_m))
head(Q7_exprs)
dim(Q7_exprs)
Q7_pd=pData(Q7_m)
Q7_group=Q7_pd[,"source_name_ch1"]
Q7_group=as.data.frame(Q7_group,row.names = rownames(Q7_pd))
colnames(Q7_group)="group_list"
library(pheatmap)
pheatmap(Q7_exprs,scale = 'row', show_rownames=F,annotation_col = Q7_group)
Q8:
找到 GPL6244 platform of Affymetrix Human Gene 1.0 ST Array 对应的R的bioconductor注释包,并且安装它!
options()BioC_mirror
options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")
options("repos" = c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
BiocManager::install("请输入自己找到的R包",ask = F,update = F)
options()BioC_mirror
答案在这里
hugene10sttranscriptcluster
BiocManager::install("hugene10sttranscriptcluster.db")
Q9:
下载数据集GSE42872的表达矩阵,并且分别挑选出 所有样本的(平均表达量/sd/mad/)最大的探针,并且找到它们对应的基因
suppressMessages(library(GEOquery))
Q9=getGEO("GSE42872",AnnotGPL = F,getGPL = F)
show(Q9)
Q9_m=Q9$GSE42872_series_matrix.txt.gz
Q9_exprs=as.data.frame(exprs(Q9_m))
head(Q9_exprs)
dim(Q9_exprs)
sort(apply(Q9_exprs,1,mean),decreasing = T)[1]
# 7978905
# 14.53288
sort(apply(Q9_exprs,1,sd),decreasing = T)[1]
# 8133876
# 3.166429
sort(apply(Q9_exprs,1,mad),decreasing = T)[1]
# 8133876
# 4.268561
suppressMessages(library("hugene10sttranscriptcluster.db"))
keytypes(hugene10sttranscriptcluster.db)
Q9_ids=toTable(hugene10sttranscriptclusterSYMBOL)
Q9_mean_g2s=Q9_ids[Q9_ids$probe_id%in%7978905,]
#没找到
Q9_sd_g2s=Q9_ids[Q9_ids$probe_id%in%8133876,]
#CD36
Q10:
下载数据集GSE42872的表达矩阵,并且根据分组使用limma做差异分析,得到差异结果矩阵
#与Q9数据一致,故不改数据名称
suppressMessages(library(GEOquery))
Q9=getGEO("GSE42872",AnnotGPL = F,getGPL = F)
show(Q9)
Q9_m=Q9$GSE42872_series_matrix.txt.gz
Q9_exprs=as.data.frame(exprs(Q9_m))
head(Q9_exprs)
dim(Q9_exprs)
Q9_pd=pData(Q9_m)
Q9_group=Q9_pd[,"source_name_ch1"]
Q9_group=as.data.frame(Q9_group,row.names = rownames(Q9_pd))
colnames(Q9_group)="group_list"
library("stringr")
Q9_group_list=as.data.frame(str_split(Q9_group$group_list,pattern = " ",simplify = T)[,6],row.names = rownames(Q9_group))
colnames(Q9_group_list)="group_list"
suppressMessages(library(limma))
design=model.matrix(~0+factor(Q9_group_list$group_list))
colnames(design)=c("vehicle","vemurafenib")
rownames(design)=rownames(Q9_group_list)
design
contrast.matrix=makeContrasts("vehicle-vemurafenib",levels = design)
contrast.matrix
fit=lmFit(Q9_exprs,design)
fit2=contrasts.fit(fit,contrast.matrix)
fit2=eBayes(fit2)
temOutput=topTable(fit2,coef = 1,n=Inf)
nrDEG=na.omit(temOutput)
head(nrDEG)
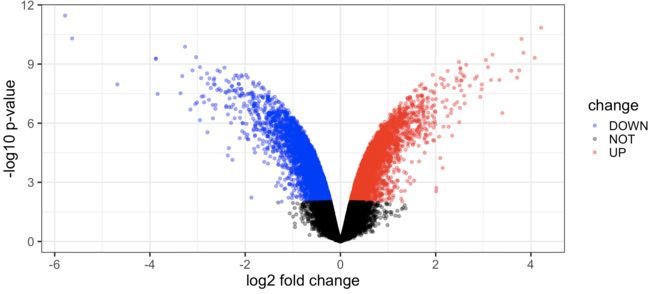
#加戏日常,差异分析都出来了,怎么不画个图?火山图来了
logFC_Cutof=with(nrDEG,mean(abs( logFC)) + 2*sd(abs( logFC)))
logFC_Cutof=0
nrDEG$change=as.factor(ifelse(nrDEG$P.Value<0.01 & abs(nrDEG$logFC)>logFC_Cutof,
ifelse(nrDEG$logFC>logFC_Cutof,'UP','DOWN'),'NOT'))
this_tile <- paste0('Cutoff for logFC is ',round(logFC_Cutof,3),'\nThe number of up gene is ',nrow(nrDEG[nrDEG$change =='UP',]) ,'\nThe number of down gene is ',nrow(nrDEG[nrDEG$change =='DOWN',]))
library(ggplot2)
g_volcano=ggplot(data=nrDEG,aes(x=logFC, y=-log10(P.Value),color=change))+
geom_point(alpha=0.4, size=1.75)+
theme_set(theme_set(theme_bw(base_size=20)))+
xlab("log2 fold change") + ylab("-log10 p-value") +
ggtitle( this_tile ) +
theme(plot.title = element_text(size=15,hjust = 0.5))+
scale_colour_manual(values = c('blue','black','red'))
print(g_volcano)
做完题了