聚类算法之DBSCAN
DBSCAN聚类算法
1. DBSCAN算法基本概念
DBSCAN是一种典型的基于密度的聚类算法,基于一组邻域 ( ϵ , M i n P t s ) (\epsilon, MinPts) (ϵ,MinPts)来描述样本集的紧密程度。其中 ϵ \epsilon ϵ描述了某一样本的邻域距离阈值, M i n P t s MinPts MinPts描述了某一样本的距离为 ϵ \epsilon ϵ的邻域中样本个数的阈值。
在DBSCAN算法中将数据点分为以下三类:
- 核心点:若样本 x i x_i xi的 ϵ \epsilon ϵ邻域内至少包含 M i n P t s MinPts MinPts样本,即 ∣ N ϵ ( x i ) ∣ ≥ M i n P t s |N_\epsilon(x_i)| \geq MinPts ∣Nϵ(xi)∣≥MinPts,则称样本点 x i x_i xi为核心点
- 边界点:若样本点 x i x_i xi的 ϵ \epsilon ϵ邻域内包含的样本数目小于 M i n P t s MinPts MinPts,但是它在其他核心点的邻域内,则称样本点 x i x_i xi为边界点
- 噪音点:既不是核心点也不是边界点的点
在DBSCAN算法中还定义了如下概念:
- 密度直达:若样本点 x j x_j xj在核心点 x i x_i xi的 ϵ \epsilon ϵ邻域内,则称样本点 x j x_j xj由 x i x_i xi密度直达。
- 密度可达:若在样本点 x i , 1 x_{i,1} xi,1和样本点 x i , n x_{i,n} xi,n之间存在序列 x i , 2 , . . . , x i , n − 1 x_{i,2},...,x_{i,n-1} xi,2,...,xi,n−1,且 x i , j + 1 x_{i,j+1} xi,j+1由 x i , j x_{i,j} xi,j密度直达,则称 x i , n x_{i,n} xi,n由 x i , 1 x_{i,1} xi,1密度可达。由密度直达的定义可知,样本点 x i , 1 , x i , 2 , . . . , x i , n − 1 x_{i,1},x_{i,2},...,x_{i,n-1} xi,1,xi,2,...,xi,n−1均为核心点
- 密度连接:对于样本点 x i x_i xi和样本点 x j x_j xj,若存在样本点 x k x_k xk,使得 x i x_i xi和 x j x_j xj都由 x k x_k xk密度可达,则称 x i x_i xi和 x j x_j xj密度相连
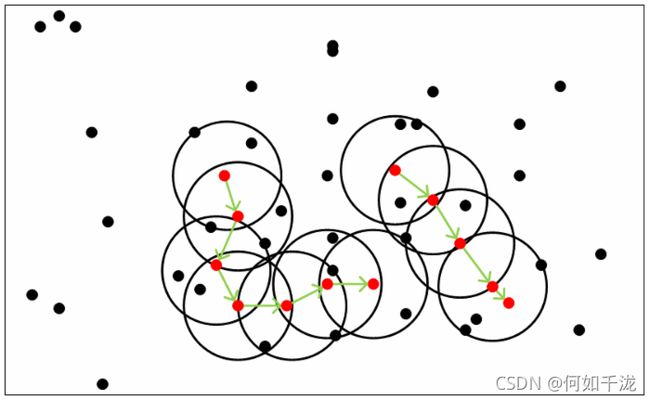
上图 M i n P t s = 5 MinPts=5 MinPts=5,红色的样本都是核心点,因为其 ϵ \epsilon ϵ邻域至少有5个样本。黑色的样本是非核心点,其中红色样本邻域内的黑色样本为边界点,其他黑色样本为噪音点。所有核心点密度直达的样本在以红色样本为中心的超球体内,如果不在超球体内,则不能密度直达。图中用绿色箭头连起来的核心点组成了密度可达的样本序列。在这些密度可达的样本序列的 ϵ \epsilon ϵ邻域内所有的样本相互都是密度相连的。
2. DBSCAN聚类算法流程
输 入 : 样 本 集 D = { x 1 , x 2 , . . . , x n } , 邻 域 参 数 ( ϵ , M i n P t s ) , 样 本 距 离 度 量 方 式 输入:样本集D=\{x_1,x_2,...,x_n\},邻域参数(\epsilon,MinPts),样本距离度量方式 输入:样本集D={ x1,x2,...,xn},邻域参数(ϵ,MinPts),样本距离度量方式
输 出 : 簇 划 分 C = { C 1 , C 2 , . . . , C k } 输出:簇划分C=\{C_1,C_2,...,C_k\} 输出:簇划分C={ C1,C2,...,Ck}
-
初始化核心点集合 Ω = ∅ \Omega=\varnothing Ω=∅,初始化聚类簇数 k = 0 k=0 k=0,初始化为访问集合 Γ = D \Gamma=D Γ=D,簇划分 C = ∅ C=\varnothing C=∅
-
对于 i = 1 , 2 , . . . , n i=1,2,...,n i=1,2,...,n,按下面步骤找出所有的核心点:
- 通过距离度量方式,找到样本 x i x_i xi的 ϵ \epsilon ϵ邻域子样本集 N ϵ ( x i ) N_\epsilon(x_i) Nϵ(xi)
- 如果子样本集样本个数满足 ∣ N ϵ ( x i ) ∣ ≥ M i n P t s |N_\epsilon(x_i)| \geq MinPts ∣Nϵ(xi)∣≥MinPts,将样本 x i x_i xi加入核心点集合: Ω = Ω ∪ { x i } \Omega=\Omega \cup \{x_i\} Ω=Ω∪{ xi}
-
如果核心点集合 Ω = ∅ \Omega=\varnothing Ω=∅,结束,否则转入步骤4
-
在核心点集合 Ω \Omega Ω中,随机选择一个核心点 o o o,初始化当前簇核心点队列 Ω c u r = { o } \Omega_{cur}=\{o\} Ωcur={ o},初始化类别序号 k = k + 1 k=k+1 k=k+1,初始化当前簇样本集合 C k = { o } C_k=\{o\} Ck={ o},更新为访问样本集合 Γ = Γ − { o } \Gamma=\Gamma-\{o\} Γ=Γ−{ o}
-
如果当前核心点队列 Ω c u r = ∅ \Omega_{cur}=\varnothing Ωcur=∅,则当前簇 C k C_k Ck生成完毕,更新簇划分 C = { C 1 , C 2 , . . . , C k } C=\{C_1,C_2,...,C_k\} C={ C1,C2,...,Ck},更新核心点集合 Ω = Ω − C k \Omega=\Omega-C_k Ω=Ω−Ck,转入步骤3。否则更新核心点集合 Ω = Ω − C k \Omega=\Omega-C_k Ω=Ω−Ck
-
在当前簇核心点队列 Ω c u r \Omega_{cur} Ωcur中取出一个核心点 o ′ o' o′,通过邻域阈值 ϵ \epsilon ϵ找出所有的 ϵ \epsilon ϵ邻域子样本集 N ϵ ( o ′ ) N_\epsilon(o') Nϵ(o′),令 Δ = N ϵ ( o ′ ) ∩ Γ \Delta=N_\epsilon(o') \cap \Gamma Δ=Nϵ(o′)∩Γ,更新当前簇样本集合 C k = C k ∪ Δ C_k=C_k \cup \Delta Ck=Ck∪Δ,更新为访问样本集合 Γ = Γ − Δ \Gamma = \Gamma - \Delta Γ=Γ−Δ,更新 Ω c u r = Ω c u r ∪ ( Δ ∩ Ω ) − { o ′ } \Omega_{cur}=\Omega_{cur} \cup (\Delta \cap \Omega)-\{o'\} Ωcur=Ωcur∪(Δ∩Ω)−{ o′},转入步骤5
简单来说:
- 根据给定的邻域参数 ϵ \epsilon ϵ和 M i n P t s MinPts MinPts确定所有的核心点
- 对每一个核心点
- 选择一个未处理过的核心点,找到由其密度可达的样本生成聚类‘簇’
- 重复以上过程
3. 实例演示
import numpy as np
import matplotlib.pyplot as plt
from sklearn import cluster, datasets
from sklearn.preprocessing import StandardScaler
np.random.seed(0)
# 构建数据
n_samples = 1500
noisy_circles = datasets.make_circles(n_samples=n_samples, factor=0.5, noise=0.05)
noisy_moons = datasets.make_moons(n_samples=n_samples, noise=0.05)
blobs = datasets.make_blobs(n_samples=n_samples, random_state=8)
data_sets = [
(
noisy_circles,
{
"eps": 0.3,
"min_samples": 5
}
),
(
noisy_moons,
{
"eps": 0.3,
"min_samples": 5
}
),
(
blobs,
{
"eps": 0.3,
"min_samples": 5
}
)
]
colors = ["#377eb8", "#ff7f00", "#4daf4a"]
plt.figure(figsize=(15, 5))
for i_dataset, (dataset, algo_params) in enumerate(data_sets):
# 模型参数
params = algo_params
# 数据
X, y = dataset
X = StandardScaler().fit_transform(X)
# 创建DBSCAN
dbscan = cluster.DBSCAN(eps=params["eps"], min_samples=params['min_samples'])
# 训练
dbscan.fit(X)
# 预测
y_pred = dbscan.labels_.astype(int)
y_pred_colors = []
for i in y_pred:
y_pred_colors.append(colors[i])
plt.subplot(1, 3, i_dataset+1)
plt.scatter(X[:, 0], X[:, 1], color=y_pred_colors)
plt.show()
4. DBSCAN小结
优点:
- 可以对任意形状的稠密数据集进行聚类,相对的,
K-Means、Mean Shift之类的聚类算法一般只适用于凸数据集 - 可以在聚类的同时发现异常点,对数据集中的异常点不敏感。
- 聚类结果没有偏倚,相对的,
K-Means之类的聚类算法初始值对聚类结果有很大影响。
缺点:
- 如果样本集的密度不均匀、聚类间距差相差很大时,聚类质量较差,这时用
DBSCAN聚类一般不适合。 - 如果样本集较大时,聚类收敛时间较长,此时可以对搜索最近邻时建立的
KD树或者球树进行规模限制来改进。 - 调参相对于传统的
K-Means之类的聚类算法稍复杂,主要需要对距离阈值 ϵ \epsilon ϵ,邻域样本数阈值 M i n P t s MinPts MinPts联合调参,不同的参数组合对最后的聚类效果有较大影响。