R语言glm logistic regression回归报错Warning messages: 1: glm.fit:算法没有聚合 2: glm.fit:拟合機率算出来是数值零或一的原因及解决方法
问题描述
在用R语言的glm函数做logistic回归时主要有以下两种报错:
Warning: glm.fit: algorithm did not converge
Warning: glm.fit: fitted probabilities numerically 0 or 1 occurred
Warning messages:
1: glm.fit:演算法沒有聚合
2: glm.fit:拟合概率算出來是数值零或一
glm.fit:演算法沒有聚合
对于第一个警告1: glm.fit:演算法沒有聚合 ,报错原因是用来拟合的数据在达到最大迭代次数时还没有收敛
(因为R在计算logistic回归时,是用极大似然估计法来进行迭代求解的,并且glm函数默认的最大迭代次数是 maxit=25,所以当拟合数据(matrix X或者说training set)不太好时,可能经过25次迭代后系数解还无法收敛)
解决方法
(1)增加最大迭代次数。比如在glm函数中设置 maxit = 200
(2)如果上述方法还无法解决问题,可能确实是 training data 不太ok,这时需要对 trainning data做进一步的处理,比如奇异值分解。
glm.fit:拟合概率算出來是数值零或一
要明白这个报错是怎么回事,我们可以先使用自带的数据集做可视化来帮助理解,先用如下函数导入数据
library("ggplot2")
data<-iris[1:100,]
samp<-sample(100,80)
names(data)<-c('sl','sw','pl','pw','species')
testdata<-data[samp,]
traindata<-data[-samp,]
lgst<-glm(testdata$species~pl,binomial(link='logit'),data=testdata)
先看看 training set 的logist回归結果,拟合出的每个样本属于’setosa’的概率是多少
lgst<-glm(testdata$species~pl,binomial(link='logit'),data=testdata,control=list(maxit=100))
p<-predict(lgst,type='response')
qplot(seq(-2,2,length=80),sort(p),col='predict')
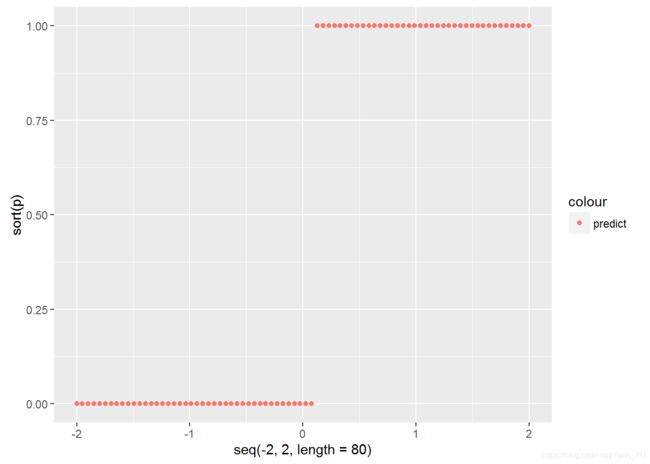
可以看出training set中拟合为’setosa’类别的概率不是几乎是0,就是几乎是1,并不是预想中的 logistic模型的S型曲线,这就是第二个报错/警告的意思。
那么问题来了,为什么会出现这种情况?
这种情况的出现可以理解为一种过拟合,由于training set的原因,在回归系数的优化搜寻过程中,使得分类的种类属于某一种类(y=1)的线性拟合值过大,分类种类为另一类(y=0)的线性拟合值过小。
由于在求解回归系数时,使用的是极大似然估计的原理,即回归系数在搜寻过程中使得似然函式极大化:
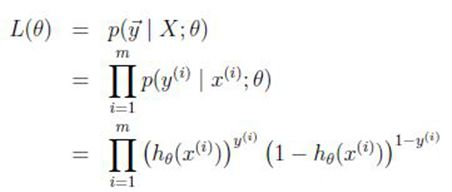
所以在搜寻过程中偏向于使得y=1的h(x)趋向于大,而使得y=0的h(x)趋向于小。
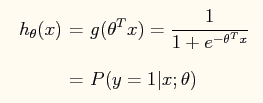
即系数Θ使得 Y=1类的 -ΘTX 趋向于大,使得Y=0类的 -ΘTX 趋向于小。而这样的结果就会导致P(y=1|x;Θ)–>1 ; P(y=0|x;Θ)–>0 .
那么问题又来了,什么样的training set会导致这样的过拟合产生呢?
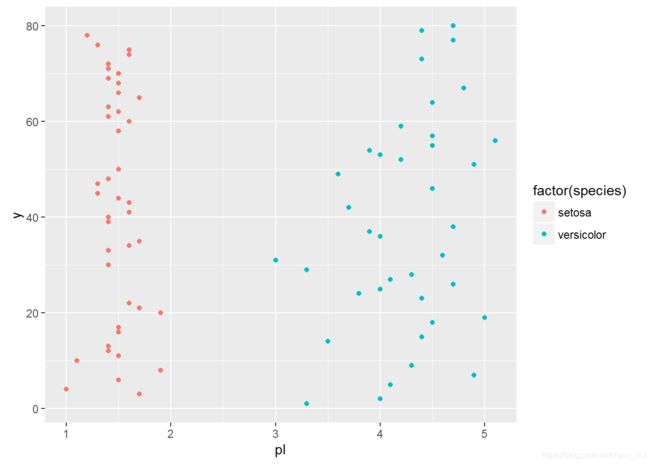
先来看看上述logistic回归中种类为setosa和versicolor的样本pl值的情况。 (横轴代表pl值,为了避免样本pl资料点叠加在一起,增加了一个无关的y值使样本点展开)
testdata$y <- c(1:80)
qplot(pl,y,data =testdata,colour =factor(species))
故在回归系数搜寻过程中只要使得一元线性函式h(x)的斜率的绝对值偏大,就可以实现y=1类的h(x)趋向大,y=0类的h(x)趋向小。
所以当training set完全可分时,logistic回归往往会导致过拟合的问题,即出现第二个警告:拟合概率算出来的概率为0或1。
解决方法
出现了第二个警告后的logistic模型进行预测时往往是不适用的,对于这种线性可分的样本资料,其实直接使用规则判断的方法则简单且适用(如当pl<2.5时则直接判断为setosa类,pl>2.5时判断为versicolor类)。
参考文献
https://blog.csdn.net/herokoking/article/details/88527082