1. 随机森林模型
随机森林是一种基于决策树(Decisiontree)的高效的机器学习算法,可以用于对样本进行分类(Classification),也可以用于回归分析(Regression)。
它属于非线性分类器,因此可以挖掘变量之间复杂的非线性的相互依赖关系。通过随机森林分析,可以找出能够区分两组样本间差异关键OTU。
Feature Importance Scores表格-来源于随机森林结果
记录了各OTU对组间差异的贡献值大小。
注:一般地,选取Mean_decrease_in_accuracy值大于0.05的OTU,作进一步分析;对于组间差异较小的样本,该值可能会降至0.03。
2. 交叉验证分析
交叉验证(Crossvalidation),是一种统计学上将数据样本切割成较小子集的实用方法。先在一个子集上做分析,而其它子集则用来做后续对此分析的确认及验证。一开始的子集被称为训练集。而其它的子集则被称为验证集或测试集。
其中最常见的为k-foldercross-validation,它指的是将所有数据分成k个子集,每个子集均做一次测试集,其余的作为训练集。交叉验证重复k次,每次选择一个子集作为测试集,并将k次的平均交叉验证识别正确率作为结果。
所有的样本都被作为了训练集和测试集,每个样本都被验证一次。
对随机森林方法筛选出的关键OTU的组合进行遍历,以期用最少的OTU数目组合构建一个错误率最低高效分类器。
一般地,对随机森林分析筛选出的关键OTU,按照不同组合进行10倍交叉验证分析,找出能够最准确区分组间差异的最少的OTU组合,再做进一步的分析,如ROC分析等。
注:图中横坐标表示不同数量的OTU组合,纵坐标表示该数量OTU组合下分类的错误率。OTU组合数越少,且错误率越低,则该OTU组合被认为是能够区分组间差异的最少的OTU组合。
3. ROC曲线
接收者操作特征曲线(Receiveroperating characteristic curve,ROC 曲线)也是一种有效的有监督学习方法。ROC分析属于二元分类算法,用来处理只有两种分类的问题,可以用于选择最佳的判别模型,选择最佳的诊断界限值。
可依据专业知识,对疾病组和参照组测定结果进行分析,确定测定值的上下限、组距以及截断点(cut-offpoint),按选择的组距间隔列出累积频数分布表,分别计算出所有截断点的敏感性(Sensetivity)、特异性和假阳性率(1-特异性:Specificity)。以敏感性为纵坐标代表真阳性率,(1-特异性)为横坐标代表假阳性率,作图绘成ROC曲线。ROC曲线越靠近左上角,诊断的准确性就越高。亦可通过分别计算各个试验的ROC曲线下的面积(AUC)进行比较,哪一种试验的AUC最大,则哪一种试验的诊断价值最佳。
注:图中横坐标为假阳性率false positive rate(FPR):Specificity,纵坐标为真阳性率true positive rate(TPR):Sensetivity。最靠近左上角的ROC曲线的点是错误最少的最好阈值,其假阳性和假阴性的总数最少。ROC曲线下的面积值在1.0和0.5之间。在AUC>0.5的情况下,AUC越接近于1,说明诊断效果越好。AUC在 0.5~0.7时有较低准确性,AUC在0.7~0.9时有一定准确性,AUC在0.9以上时有较高准确性。AUC=0.5时,说明诊断方法完全不起作用,无诊断价值。AUC<0.5不符合真实情况,在实际中极少出现。
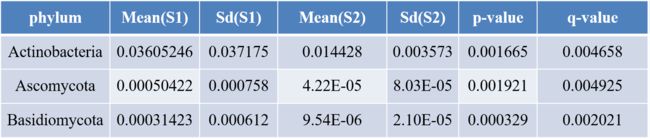
4. Wilcoxon秩和检验分析
Wilcoxonrank-sum test,也叫曼-惠特尼U检验(Mann–WhitneyU test),是两组独立样本非参数检验的一种方法。其原假设为两组独立样本来自的两总体分布无显著差异,通过对两组样本平均秩的研究来实现判断两总体的分布是否存在差异,该分析可以对两组样品的物种进行显著性差异分析,并对p值计算假发现率(FDR)q值。
注:mean分别为两组样品物种的平均相对丰度,sd分别是两组样本物种相对丰度的标准差。P值为对两组检验原假设为真的概率值,p<0.05表示存在差异,p<0.01表示差异显著,q值为假发现率。
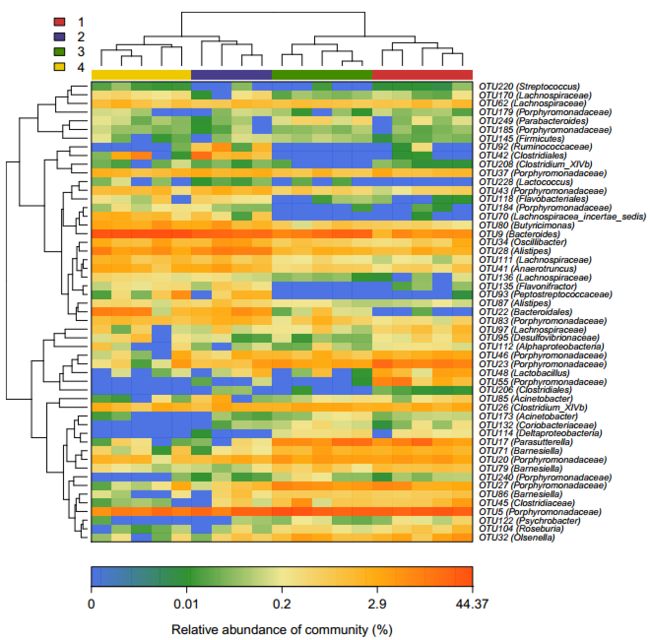
5. 差异菌群Heatmap分析
以10倍交叉验证(10-foldcross-validation)估计泛化误差(Generalizationerror)的大小,其余参数使用默认设置。建模结果同时包含“基线”误差(Baselineerror)的期望值,即数据集中属于最优势分类的样本全部被错误分类的概率。每个OTU根据其被移除后模型预报错误率增加的大小确定其重要度数值,重要度越高,该OTU对模型预报准确率的贡献越大。
根据挑选出来的差异OTU,根据其在每个样品中的丰度信息,对物种进行聚类,绘制成热图,便于观察哪些物种在哪些样品中聚集较多或含量较低。
注:图中越接近蓝色表示物种丰度越低,越接近橙红色表示丰度越高。左边的聚类树是根据各物种间的spearman相关性距离进行聚类;上边的聚类树是采用样本间距离算法中最常用的Bray-Curtis算法进行聚类。
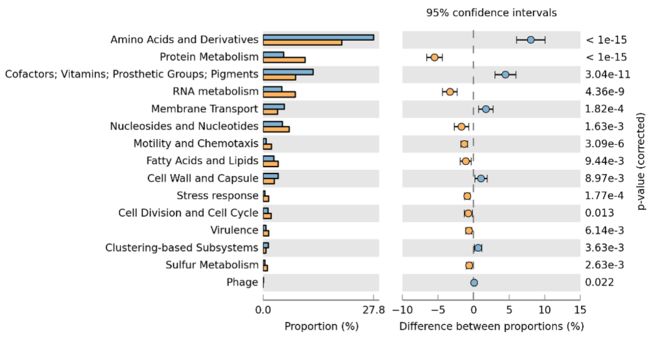
6. 两组样本Welch’s t-test分析
两组不同方差的样本可使用Welch’st-test进行差异比较分析,通过此分析可获得在两组中有显著性差异的物种[或差异基因丰度—适用于元(宏)基因组]。
注:上图所示为不同基因丰度(或物种)在两组样品中的丰度比例,中间所示为95%置信度区间内,物种丰度的差异比例,最右边的值为p值,p值<0.05,表示差异显著。
7. Shannon多样性指数比较盒状图
将不同分类或环境的多组样本的Shannon多样性指数进行四分位计算,比较不同样本组的组间Shannon指数差异。同时进行非参数Mann-Whitney判断样本组间的显著性差异。
注:横坐标表示样本分组,纵坐标表示相对应的Alpha多样性指数值;图形可以显示5个统计量(最小值,第一个四分位,中位数,第三个中位数和最大值,及由下到上5条线)。p<0.05,表示差异显著;P<0.01,表示差异极显著。
8. 基于距离的箱式图
将不同分类或环境的多组样本的距离进行四分位计算,比较不同样本组的组内和组间的距离分布差异。同时进行multipleStudent's two-sample t-tests判断样本组间差异的显著性。
箱式图的作用:识别数据异常值;粗略估计和判断数据特征;比较几批数据的形状,同一数轴上,几批数据的箱形图并行排列,几批数据的中位数、尾长、异常值、分布区间等形状信息一目了然。
箱线图(Boxplot)也称箱须图(Box-whiskerPlot),是利用数据中的五个统计量:最小值、第一四分位数、中位数、第三四分位数与最大值来描述数据的一种方法,它也可以粗略地看出数据是否具有对称性,分布的分散程度等信息,特别可以用于对几组样本的比较。简单箱线图由五部分组成,分别是最小值、中位数、最大值和两个四分位数。
注:第一四分位数 (Q1),又称“下四分位数”,等于该样本中所有数值由小到大排列后第25%的数字。第二四分位数 (Q2),又称“中位数”,等于该样本中所有数值由小到大排列后第50%的数字。 第三四分位数 (Q3),又称“上四分位数”,等于该样本中所有数值由小到大排列后第75%的数字。
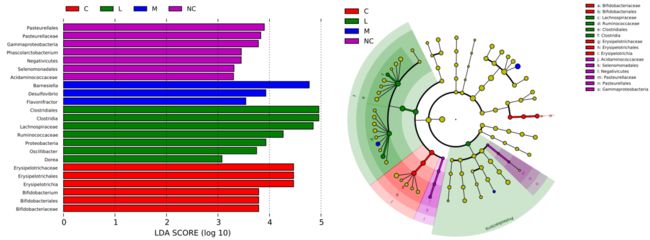
9. LEfSe分析
LEfSe是一种用于发现高维生物标识和揭示基因组特征的软件。包括基因,代谢和分类,用于区别两个或两个以上生物条件(或者是类群)。该算法强调的是统计意义和生物相关性。让研究人员能够识别不同丰度的特征以及相关联的类别。
LEfSe通过生物学统计差异使其具有强大的识别功能。然后,它执行额外的测试,以评估这些差异是否符合预期的生物学行为。
具体来说,首先使用non-parametric factorial Kruskal-Wallis (KW) sum-rank test(非参数因子克鲁斯卡尔—沃利斯和秩验检)检测具有显著丰度差异特征,并找到与丰度有显著性差异的类群。最后,LEfSe采用线性判别分析(LDA)来估算每个组分(物种)丰度对差异效果影响的大小。
说明:左边的图为统计两个组别当中有显著作用的微生物类群通过LDA分析(线性回归分析)后获得的LDA分值。右边的图为聚类树,节点大小表示丰度,默认从门到属依次向外排列。红色区域和绿色区域表示不同分组,树枝中红色节点表示在红色组别中起到重要作用的微生物类群,绿色节点表示在绿色组别中起到重要作用的微生物类群,黄色节点表示的是在两组中均没有起到重要作用的微生物类群。图中英文字母表示的物种名称在右侧图例中进行展示。
10. ANOSIM相似性分析
相似性分析(ANOSIM)是一种非参数检验,用来检验组间(两组或多组)的差异是否显著大于组内差异,从而判断分组是否有意义。首先利用Bray-Curtis算法计算两两样品间的距离,然后将所有距离从小到大进行排序,按以下公式计算R值,之后将样品进行置换,重新计算R*值,R*大于R的概率即为P值。
其中,
r ̅ _b:表示组间(Between groups)距离排名的平均值;
r ̅ _w:表示组内(Within groups)距离排名的平均值;
n:表示样品总数。
Table. Anosimanalysis
注:理论上,R值范围为-1到+1,实际中R值一般从0到1,R值接近1表示组间差异越大于组内差异,R值接近0则表示组间和组内没有明显差异。P值则反映了分析结果的统计学显著性,P值越小,表明各样本分组之间的差异显著性越高,P< 0.05表示统计具有显著性;Number of permutation表示置换次数。
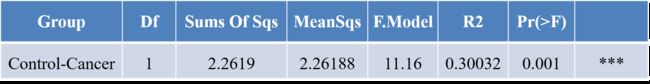
11. Adonis多因素方差分析
Adonis又称置换多因素方差分析(permutationalMANOSVA)或非参数多因素方差分析(nonparametricMANOVA)。它利用半度量(如Bray-Curtis)或度量距离矩阵(如Euclidean)对总方差进行分解,分析不同分组因素对样品差异的解释度,并使用置换检验对划分的统计学意义进行显著性分析。
Table permutational MANOVA analysis
注:
Group:表示分组;
Df:表示自由度;
SumsOfSqs:总方差,又称离差平方和;
MeanSqs:平均方差,即SumsOfSqs/Df;
F.Model:F检验值;
R2:表示不同分组对样品差异的解释度,即分组方差与总方差的比值,即分组所能解释的原始数据中差异的比例,R2越大表示分组对差异的解释度越高;
Pr(>F):通过置换检验获得的P值,P值越小,表明组间差异显著性越强。