me, knowing nothing still
P23 系统的入门 R 语言
03:01 荐书:《R语言实战(第2版》
05:07 安装 R 及编辑器 Rstudio
05:45 介绍 Rstudio,内容和 P1 基本一致
-
08:15 设置默认镜像,切换为国内镜像 ,从此速度飞起
## 清华镜像 > options(repos=structure(c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))) ## 中科大镜像 > options(repos=structure(c(CRAN="https://mirrors.ustc.edu.cn/bioc/"))) 11:00 R 与 excel 的异同,内容和 P15 基本一致
19:18 理解R 中的变量
P24 R 语言可视化基础
-
01:03 用 R Markdown 写网页
'knit' 生成 html 文件
-
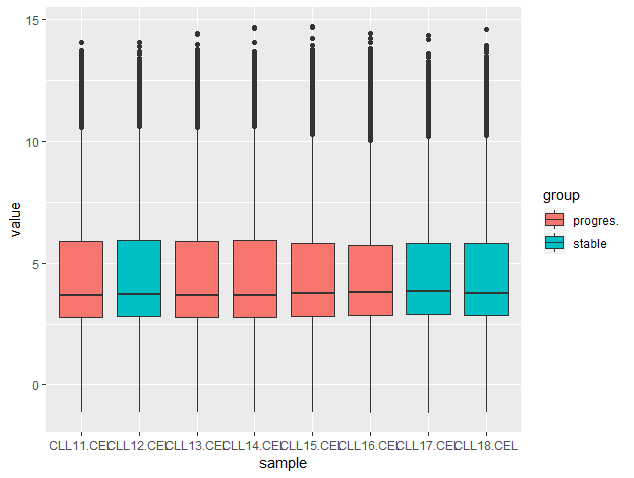
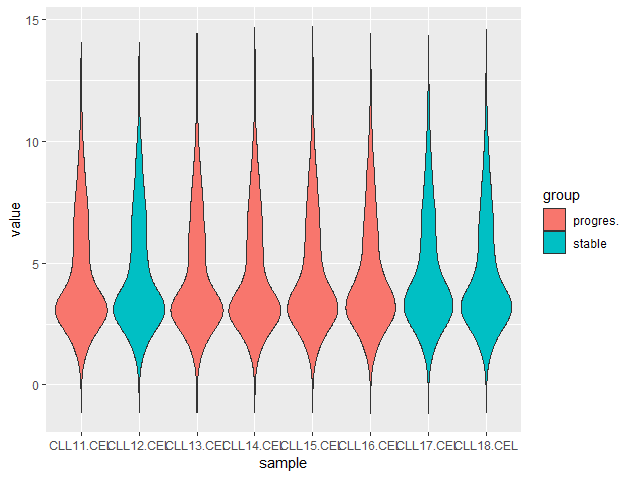
05:54 载入数据,绘图
> library(CLL) > data("sCLLex") > sCLLex=sCLLex[,1:8] > group_list=sCLLex$Disease > exprSet=exprs(sCLLex) > write.table(exprSet,'exprSet.txt') > exprSet_L$group=rep(group_list,each=nrow(exprSet))> ggplot(exprSet_L,aes(x=sample,y=value,fill=group))+ + geom_boxplot()> ggplot(exprSet_L,aes(x=sample,y=value,fill=group))+ + geom_violin()
P25 R 语言可视化进阶
-
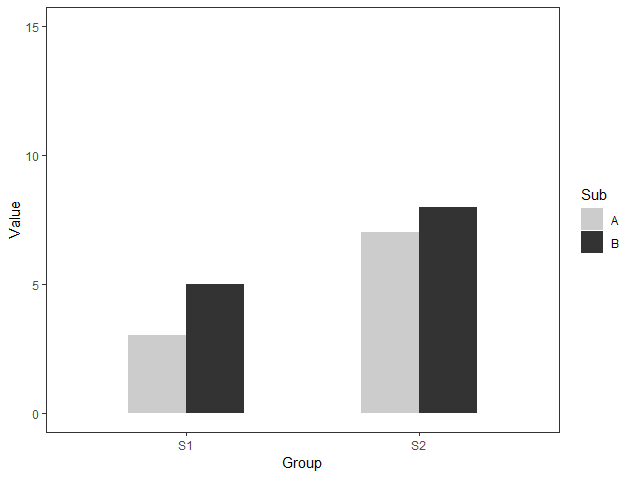
05:15 复现一个例子
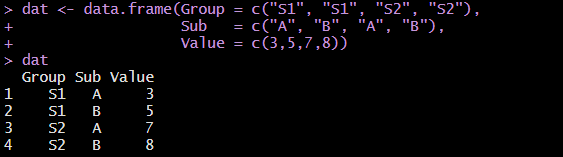
> dat <- data.frame(Group = c("S1", "S1", "S2", "S2"), + Sub = c("A", "B", "A", "B"), + Value = c(3,5,7,8))> p <- + ggplot(dat, aes(Group, Value)) + + theme_bw() + theme(panel.grid = element_blank()) + + coord_cartesian(ylim = c(0, 15)) + + scale_fill_manual(values = c("grey80", "grey20")) + + geom_bar(aes(fill = Sub), stat="identity", position="dodge", width=.5)加元素
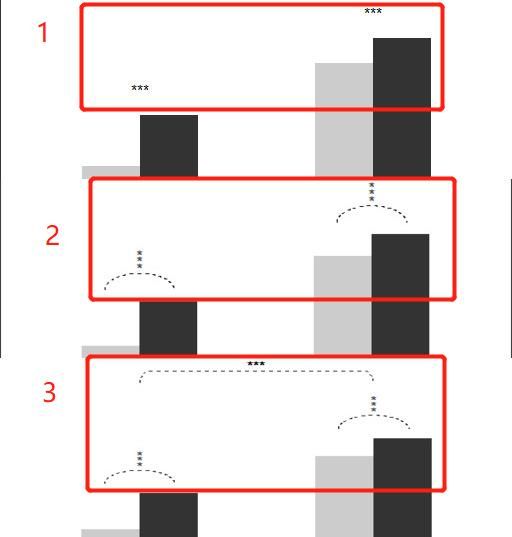
## 1 > label.df <- data.frame(Group = c("S1", "S2"), + Value = c(6, 9)) > p + geom_text(data = label.df, label = "***")## 2 > label.df <- data.frame(Group = c(1,1,1, 2,2,2), + Value = c(6.5,6.8,7.1, 9.5,9.8,10.1)) > r <- 0.15 > t <- seq(0, 180, by = 1) * pi / 180 > x <- r * cos(t) > y <- r*5 * sin(t) > arc.df <- data.frame(Group = x, Value = y) > p2 <- + p + geom_text(data = label.df, label = "*") + + geom_line(data = arc.df, aes(Group+1, Value+5.5), lty = 2) + + geom_line(data = arc.df, aes(Group+2, Value+8.5), lty = 2)## 3 > r <- .5 > x <- r * cos(t) > y <- r*4 * sin(t) > y[20:162] <- y[20] > arc.df <- data.frame(Group = x, Value = y) > p2 + geom_line(data = arc.df, aes(Group+1.5, Value+11), lty = 2) + + geom_text(x = 1.5, y = 12, label = "***")
P26 用 shiny 写一个计算器
-
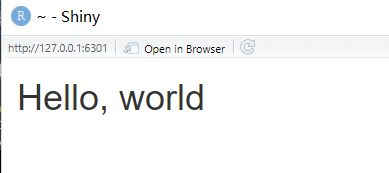
01:43 用 shiny 写网页
> library(shiny) > ui <- fluidPage(h1('Hello, world')) > server <- function(input,output){} > shinyApp(ui=ui,server = server) -
03:37 用 shiny 写计算器,test
> library(shiny) > ui <- fluidPage(textInput('a','please input the first number:'), + checkboxInput("some value","some value",FALSE), + textInput('a','please input the second number:')) > server <- function(input,output){} > shinyApp(ui=ui,server = server) -
08:22 给计算器加功能
> library(shiny) > ui <- fluidPage(textInput("a","please input the first number:"), + checkboxGroupInput("op","please choose a sign", + c("+" = "+", + "-" = "-", + "*" = "*", + "/" = "/", + "%" = "%%")), + textInput("b","please input the second number:"), + submitButton("do","calculator"), + textOutput("result") + ) > server <- function(input,output){ + output$result <- renderPrint({ + input$do + print(eval(parse(text=paste0(input$a,input$op,input$b)))) + }) + } > shinyApp(ui=ui,server = server)
P27 R 语言操作图片
-
01:05 图片合并 (merge),导出 PDF
设置边界,重复图片120次
> library("png") > img <- readPNG(system.file("img", "Rlogo.png", package="png")) > par(mar=rep(0,4)) # no margins > layout(matrix(1:120, ncol=12, byrow=TRUE))绘图
> for(i in 1:120) { + plot(NA,xlim=0:1,ylim=0:1,xaxt="n",yaxt="n",bty="n") + rasterImage(img,0,0,1,1) + }导出为 PDF
> dev.print(pdf, "output.pdf") -
03:24 合并两张图片
> img1 <- readPNG(system.file("img", "Rlogo.png", package="png")) > img2 <- readPNG(system.file("img", "Rlogo.png", package="png")) > layout(1:2) > plot(NA,xlim=0:1,ylim=0:1,xaxt="n",yaxt="n",bty="n") > rasterImage(img1,0,0,1,1) > plot(NA,xlim=0:1,ylim=0:1,xaxt="n",yaxt="n",bty="n") > rasterImage(img2,0,0,1,1) -
06:44 生成 gif
安装
ImageMagick> install.packages("installr") > require(installr) > install.ImageMagick()生成 gif
> dir.create("examples") > setwd("examples") > png(file="example%02d.png", width=200, height=200) > for (i in c(10:1, "G0!")){ + plot.new() + text(.5, .5, i, cex = 6) + } > dev.off() > system("convert -delay 80 *.png example_1.gif") > file.remove(list.files(pattern=".png"))在 examples 文件夹下生成了 “1-10” 和 “GO!” 的 png 文件,然鹅....
> system("convert -delay 80 *.png example_1.gif") 无效参数 - 0放弃
P28 R 语言写爬虫
-
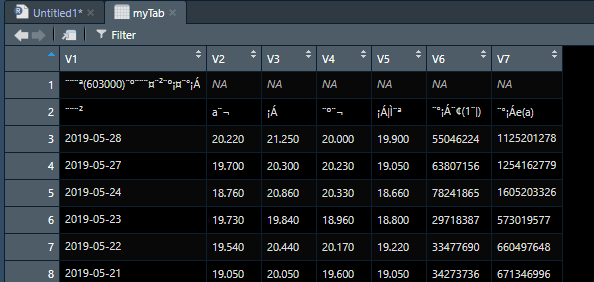
01:46 示例 (该博客已过期……:disappointed_relieved:
> library(XML) > library(RCurl) > URL = getURL("http://vip.stock.finance.sina.com.cn/corp/go.php/vMS_MarketHistory/stockid/603000.phtml?year=2019&jidu=2") > doc <- htmlParse(URL,encoding = "utf-8") > tables<-readHTMLTable(doc,header = F) > myTab=tables[[14]]
P29 用 R 语言爬取生信软件列表到思维导图
-
00:48 数据获取
> library(rvest) > url='https://omictools.com/single-cell-rna-seq-category#' > fit <- try(web <- read_html(url),silent =TRUE) ## 获取原代码 > if('try-error' %in% class(fit)){ + cat('HTTP error 404\n') + }else{ + h2 <- web %>% html_nodes('h2') %>% html_text() + h3 <- web %>% html_nodes('h3') %>% html_text() + }Jimmy大大视频里的代码是:
h2 <<- web %>% html_nodes('h2') %>% html_text() h3 <<- web %>% html_nodes('h3') %>% html_text()不造为什么报错:Error: cannot change value of locked binding for 'h2' , ’Error: cannot change value of locked binding for 'h3'‘
> library(stringr) > h2=trimws(h2) > h3=trimws(h3)
-
08:33 制作思维导图
> write.table(h2,'h2.txt',row.names = F,col.names = F,quote = F) > write.table(h3,'h3.txt',row.names = F,col.names = F,quote = F)从
h2.txt,h3.txt文件中得到分类名称,复制到幕布得到网页中 tools 的个数
> web %>% html_nodes('div.category-info span') %>% html_text()
P30 用 R 语言进行 ID 转换
-
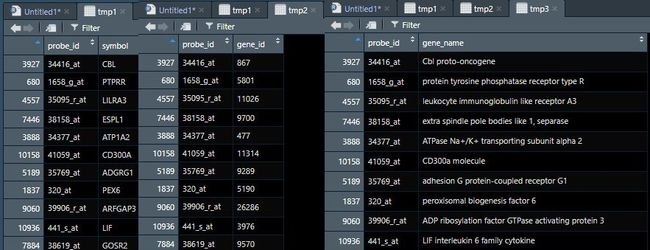
02:25 通过
hgu95av2.db包,分别将探针和 symbol, geneid, genename 匹配,获得 probe, symbol, geneID 等文件> library("hgu95av2.db") > probe2entrezID=toTable(hgu95av2ENTREZID) > probe2symbol=toTable(hgu95av2SYMBOL) > probe2genename=toTable(hgu95av2GENENAME) > my_probe = sample(unique(mappedLkeys(hgu95av2ENTREZID)),30) > tmp1 = probe2symbol[match(my_probe,probe2symbol$probe_id),] > tmp2 = probe2entrezID[match(my_probe,probe2entrezID$probe_id),] > tmp3 = probe2genename[match(my_probe,probe2genename$probe_id),] > write.table(my_probe,'my_probe.txt',quote = F,col.names = F,row.names =F) > write.table(tmp1$symbol,'my_symbol.txt',quote = F,col.names = F,row.names =F) > write.table(tmp2$gene_id,'my_geneID.txt',quote = F,col.names = F,row.names =F) -
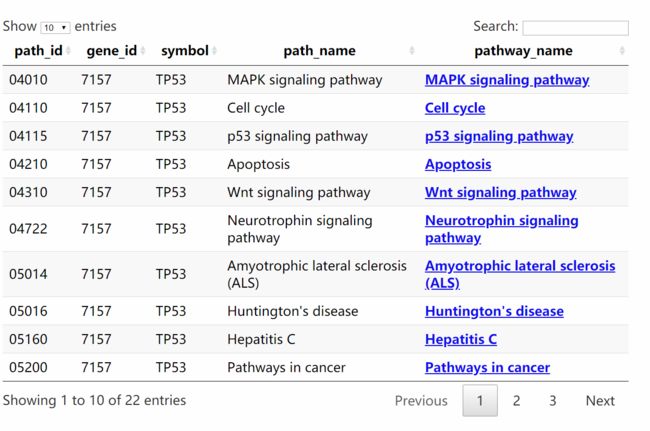
07:00
org.Hs.eg.db包可以对应到通路> keggAnnotation <- function(geneLists=c(1,2,9),update=F,dir_bin='KEGG_update',fileType='html',fileName='gene'){ + if(update){ + dir_bin <- 'YourPath' + path2gene_file=file.path(dir_bin,'kegg2geneID.txt') + path2name_file=file.path(dir_bin,'kegg_hierarchical.txt') + keggID2geneID=read.table(path2gene_file,sep="\t",colClasses=c('character')) + keggID2geneID=keggID2geneID[,c(2,1)] + names(keggID2geneID)=c('gene_id','path_id') + keggID2name<<- read.delim(path2name_file,header=F,sep="\t",colClasses=c('character'),stringsAsFactors =F)[,3:4] + colnames(keggID2name)=c('path_id','path_name') + }else{ + suppressMessages(library("org.Hs.eg.db")) + keggID2geneID=toTable(org.Hs.egPATH) + suppressMessages(library("KEGG.db")) + keggID2name=toTable(KEGGPATHID2NAME) + } + suppressMessages(library("org.Hs.eg.db")) + all_EG=mappedkeys(org.Hs.egSYMBOL) + EG2Symbol=toTable(org.Hs.egSYMBOL) + EG2Symbol$symbol=toupper(EG2Symbol$symbol) + if( all(! geneLists %in% all_EG) ){ + inputType='symbol' + geneLists=data.frame(symbol=geneLists) + results=merge(geneLists,EG2Symbol,by='symbol',all.x=T) + }else{ + inputType='entrezID' + geneLists=data.frame(gene_id=geneLists) + results=merge(geneLists,EG2Symbol,by='gene_id',all.x=T) + } + tmp=merge(results,keggID2geneID,by = 'gene_id') + tmp2=merge(tmp,keggID2name,by='path_id') + kegg_info=tmp2 + if(fileType !='html'){ + write.csv(kegg_info, paste0(fileName,'_kegg_anno.csv'),row.names = F) + } + else{ + kegg_info$pathway_name=apply(kegg_info,1,function(x) { + pathwayID=sprintf("%05.0f",as.numeric(x['path_id'])) + pathwayName=x['path_name'] + href=paste0("http://www.genome.jp/kegg-bin/show_pathway?hsa", + pathwayID,"+hsa:",x['gene_id'] + ) + tmp=paste0('',pathwayName,'') + }) + kegg_info=kegg_info[order(kegg_info[,'gene_id']),] + DT::datatable(kegg_info,escape = FALSE,rownames=F) + y <- DT::datatable(kegg_info,escape = F,rownames=F) + DT::saveWidget(y, paste0(fileName,'_kegg_anno.html')) + } + }得到函数
keggAnnotation()生成一个 html 文件
P31 多个差异分析结果直接量量取交集并集
-
03:53 用 perl 得到一个由26个基因 (A-Z) 随机取10次的列表
perl -e 'BEGIN{use List::Util qw/shuffle/;@gene=A..Z}{foreach(1..10){@this_genes=@gene[(shuffle(0..25))[0..int(rand(20))+4]];print "comparison_$_ <- ";print join(";",@this_genes);print "\n"}}' > test.txt -
04:15 用 R 操作
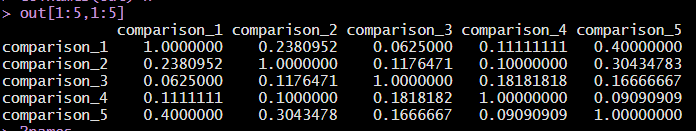
> rm(list = ls()) > a=readLines('test.txt') > n=unlist(lapply(a , function(x){ + trimws(strsplit(x,'<-')[[1]][1]) > + })) ## 按‘<-’分割,取第1个元素 > v=lapply(a , function(x){ > + trimws(strsplit(trimws(strsplit(x,'<-')[[1]][2]),';')[[1]]) > + }) ## 按‘<-’分割,取第2个元素,再按';'分割,取第1个元素 > names(v)=n > tmp=unlist(lapply(v, function(x){ > + lapply(v, function(y){ > + p1=length(intersect(x,y)) > + p2=length(union(x,y)) > + return(p1/p2) > + }) > + })) ## 每个基因循环2次 > out=matrix(tmp,nrow = length(n)) > rownames(out)=n > colnames(out)=n > out[1:5,1:5]
P32 用 R 语言进行多个同样的行列式文件合并
05:10 数据下载
GSE48213_RAW.tar-
11:37 在 shell 中合并文件
awk '{print FILENAME"\t"$0}' * |grep -v EnsEMBL_Gene_ID > tmp.txt P32.2
P32.2 -
12:20 在 R 中操作,得到表达矩阵
> a=read.table('tmp.txt',sep = '\t',stringsAsFactors = F) > library(reshape2) > fpkm <- dcast(a,formula = V2~V1) > rownames(fpkm) <- fpkm[,1] > fpkm <- fpkm[,-1]
> unlist(lapply(names(fpkm), function(x){
+ tmp <- strsplit(x,'_')[[1]][2]
+ tmp <- strsplit(tmp,'\\.')[[1]][1]
+ })
+ )
-
35:46
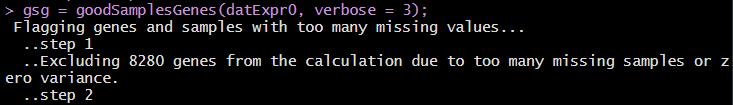
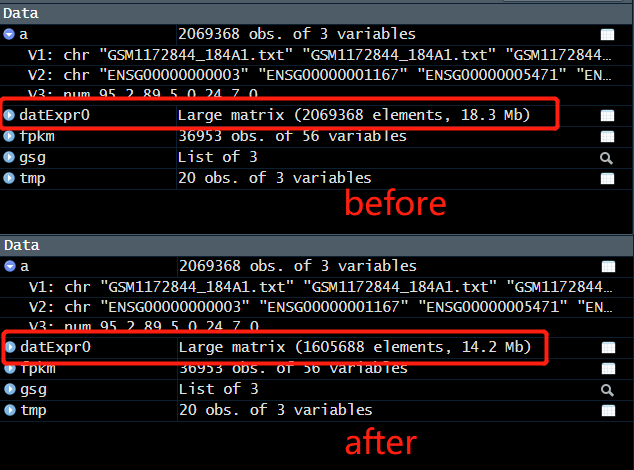
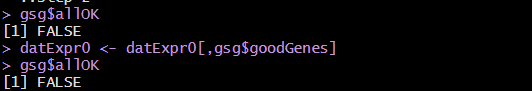
> datExpr0=t(fpkm) > library(WGCNA) > gsg = goodSamplesGenes(datExpr0, verbose = 3);> gsg$allOK > datExpr0 <- datExpr0[,gsg$goodGenes] > gsg$allOK -
42:38 聚类分析绘图
> sampleTree = hclust(dist(datExpr0), method = "average") > par(cex = 0.6) > par(mar = c(0,4,2,0)) > plot(sampleTree, main = "Sample clustering to detect outliers", sub="", xlab="", cex.lab = 1.5, + cex.axis = 1.5, cex.main = 2) > abline(h = 80000, col = "red")> clust = cutreeStatic(sampleTree, cutHeight = 80000, minSize = 10) > table(clust) clust 0 56 > keepSamples = (clust==1) > datExpr = datExpr0[keepSamples, ] > nGenes = ncol(datExpr) > nSamples = nrow(datExpr) > clust # [1] 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 # [38] 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0emmmmmm 视频结束了,这一步我当然也不会...
最后,向大家隆重推荐生信技能树的一系列干货!
- 生信技能树全球公益巡讲:https://mp.weixin.qq.com/s/E9ykuIbc-2Ja9HOY0bn_6g
- B站公益74小时生信工程师教学视频合辑:https://mp.weixin.qq.com/s/IyFK7l_WBAiUgqQi8O7Hxw
- 招学徒:https://mp.weixin.qq.com/s/KgbilzXnFjbKKunuw7NVfw