scATAC分析神器ArchR初探-简介(1)
scATAC分析神器ArchR初探-ArchR进行doublet处理(2)
scATAC分析神器ArchR初探-创建ArchRProject(3)
scATAC分析神器ArchR初探-使用ArchR降维(4)
scATAC分析神器ArchR初探--使用ArchR进行聚类(5)
scATAC分析神器ArchR初探-单细胞嵌入(6)
scATAC分析神器ArchR初探-使用ArchR计算基因活性值和标记基因(7)
scATAC分析神器ArchR初探-scRNA-seq确定细胞类型(8)
scATAC分析神器ArchR初探-ArchR中的伪批次重复处理(9)
scATAC分析神器ArchR初探-使用ArchR-peak-calling(10)
scATAC分析神器ArchR初探-使用ArchR识别标记峰(11)
scATAC分析神器ArchR初探-使用ArchR进行主题和功能丰富(12)
scATAC分析神器ArchR初探-利用ArchR丰富ChromVAR偏差(13)
scATAC分析神器ArchR初探-使用ArchR进行足迹(14)
scATAC分析神器ArchR初探-使用ArchR进行整合分析(15)
scATAC分析神器ArchR初探-使用ArchR进行轨迹分析(16)
14-使用ArchR进行足迹
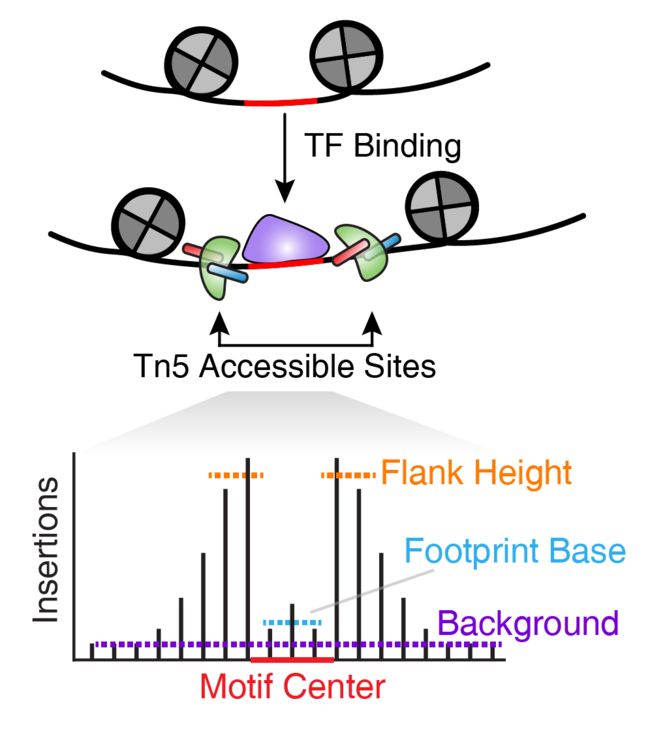
转录因子(TF)足迹可以预测TF在特定位点的精确结合位置。这是因为实际上可以保护直接与TF结合的DNA碱基不易转位,而可以接近与TF结合紧邻的DNA碱基。
理想情况下,TF足迹在单个位置执行,以确定TF的精确结合位置。但是,实际上,这需要很高的测序深度,通常比大多数用户从批量或单细胞ATAC序列获得的测序深度要高得多。为了解决这个问题,我们可以在预计的TF绑定的许多实例中组合Tn5插入位置。例如,我们可以获取所有带有CTCF基序的峰,并在整个基因组中为CTCF绘制总计的TF足迹。
该足迹的准确性取决于为感兴趣的TF生成可靠的预测结合位点清单。ArchR addMotifAnnotations()通过在峰区域中搜索与基序匹配的任何DNA序列,以一种幼稚的方式通过功能实现了此功能。取决于目的基元的简并性,这可能是足够的,也可能不是足够的。这些基元注释ArchRProject以每个峰的二进制表示形式添加到中(0 =无基元,1 =存在基元)。有了这些图案注释后,ArchR将使用将getFootprints()一个ArchRProject对象和一个GenomicRanges包含图案位置的对象作为输入的功能执行覆盖。这些位置可以ArchRProject通过getPositions()功能从中访问。然后可以使用plotFootprints() 功能。
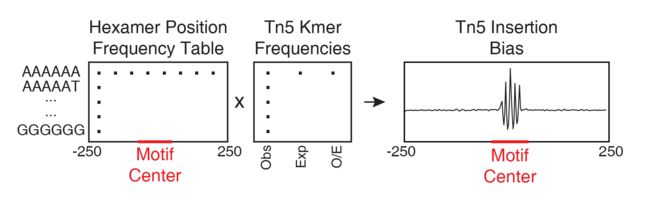
也许最重要的是,ArchR中的足迹分析分析了已知的Tn5插入序列偏差。为此,ArchR在Tn5插入位点使用六聚体位置频率矩阵和k-mer频率矩阵:
综上所述,该工作流生成的足迹图考虑了Tn5插入偏差。
ArchR支持主题足迹和用户提供的功能的自定义足迹,本章将对这两者进行讨论。
14.1主题足迹
重要的是,从本教程数据生成的足迹并不理想,这是因为教程数据集的大小很小。从较大的数据集生成的足迹将显示出更少的变化。
进行覆盖时,我们要做的第一件事就是获取相关图案的位置。为此,我们调用getPositions()函数。该函数有一个可选参数name,它可以接受peakAnnotation我们要从中获取位置的对象的名称。如果为name = NULL,则ArchR将使用peakAnnotation插槽中的第一个条目。在下面显示的示例中,我们未指定name并且ArchR使用第一个条目即CIS-BP主题。
motifPositions <- getPositions(projHeme5)
这将创建一个GRangesList对象,其中每个TF主题都由一个单独的GRanges对象表示。
motifPositions
我们可以将其子集GRangesList化为我们感兴趣的几个TF主题。由于在搜索“ EBF1”时出现了SREBF1 TF,因此我们使用%ni%提供与%in%base 相对功能的辅助函数从下面的下游分析中明确删除了它。R.
motifs <- c("GATA1", "CEBPA", "EBF1", "IRF4", "TBX21", "PAX5")
markerMotifs <- unlist(lapply(motifs, function(x) grep(x, names(motifPositions), value = TRUE)))
markerMotifs <- markerMotifs[markerMotifs %ni% "SREBF1_22"]
markerMotifs
为了准确地描绘TF足迹,需要进行大量读取。因此,将单元分组以创建伪批量ATAC-seq配置文件,然后将其用于TF足迹。这些伪批量配置文件存储为组覆盖文件,我们最初在上一章中创建了这些文件以执行高峰调用。如果您尚未将网上论坛报道添加到中ArchRProject,请立即进行。
projHeme5 <- addGroupCoverages(ArchRProj = projHeme5, groupBy = "Clusters2")
通过计算组覆盖率,我们现在可以计算先前使用该getFootprints()功能选择的标记模体子集的足迹。即使ArchR实现了高度优化的足迹工作流程,还是建议对子集而不是所有主题执行足迹。因此,我们通过positions参数将图案的子集提供给足迹。
seFoot <- getFootprints(
ArchRProj = projHeme5,
positions = motifPositions[markerMotifs],
groupBy = "Clusters2"
)
一旦检索到这些足迹,就可以使用plotFootprints()函数将其绘制出来。此功能可以同时以各种方式对足迹进行归一化。下一部分将讨论足迹的标准化和实际绘制。
14.2 Tn5偏差的足迹归一化
使用ATAC-seq数据进行TF足迹的一个主要挑战是Tn5转座酶的插入序列偏倚,这可能导致TF足迹的错误分类。为了解决Tn5插入偏差,ArchR会识别每个Tn5插入位点周围的k-mer(用户定义的长度,默认长度6)序列。为了进行此分析,ArchR会为每个假体识别单碱基分辨率Tn5插入位点,将这些1 bp位点的大小调整为k-bp窗口(插入后为-k / 2和+(k / 2-1)bp),然后使用包中的oligonucleotidefrequency(w=k, simplify.as="collapse")函数创建一个k-mer频率表Biostrings。然后,ArchR使用与BSgenome相关的基因组文件。为了计算伪大量足迹的插入偏差,ArchR创建了一个k-mer频率矩阵,该矩阵表示为距图案中心+/- N bp(用户定义,默认为250 bp)的窗口上所有可能的k-mer。 。然后,在每个主题位点上迭代,ArchR将定位的k-mers填充到k-mer频率矩阵中。然后针对全基因组范围内的每个基序位置进行计算。使用样本的k-mer频率表,ArchR可以通过将k-mer位置频率表乘以观察到/预期的Tn5 k-mer频率来计算预期的Tn5插入。
所有这些都发生在plotFootprints()功能内部。
14.2.1减去Tn5偏置
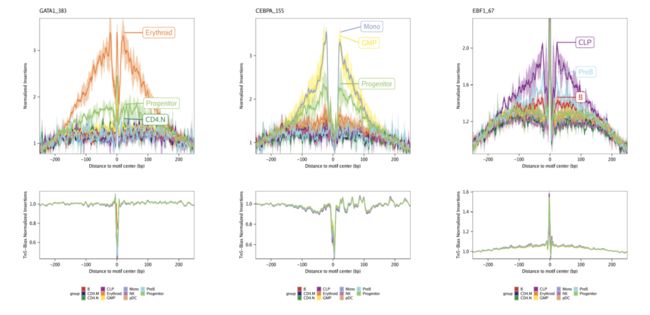
一种归一化方法是从覆盖信号中减去Tn5偏置。通过normMethod = "Subtract"调用时的设置来执行此规范化plotFootprints()。
plotFootprints(
seFoot = seFoot,
ArchRProj = projHeme5,
normMethod = "Subtract",
plotName = "Footprints-Subtract-Bias",
addDOC = FALSE,
smoothWindow = 5
)
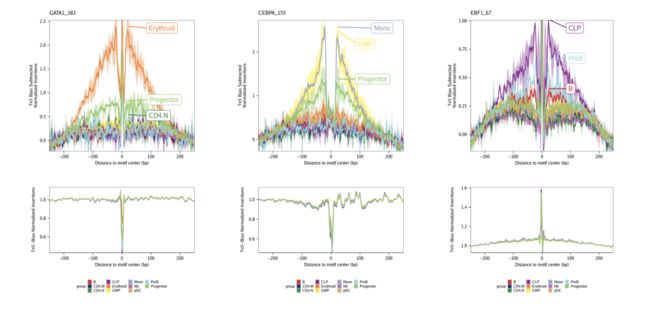
默认情况下,这些图将保存在outputDirectory中ArchRProject。如果您要求绘制所有图案并将其作为ggplot对象返回,则该ggplot对象将非常大。偏差减去分析后的图案足迹示例如下所示。
14.2.2除以Tn5偏差
归一化的第二种策略是将足迹信号除以Tn5偏置信号。通过normMethod = "Divide"调用时的设置来执行此规范化plotFootprints()。
plotFootprints(
seFoot = seFoot,
ArchRProj = projHeme5,
normMethod = "Divide",
plotName = "Footprints-Divide-Bias",
addDOC = FALSE,
smoothWindow = 5
)
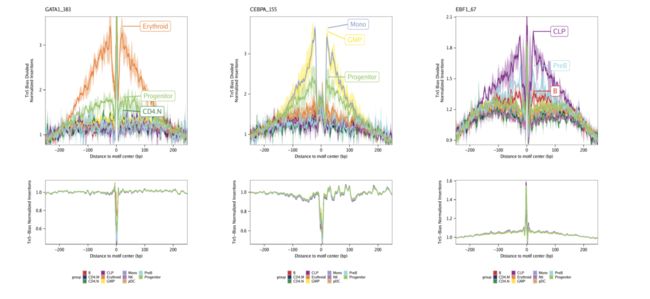
下面显示了来自偏差划分的分析的图案足迹示例。
14.2.3 Tn5偏差未归一化的封装
虽然我们强烈推荐的Tn5序列插入偏置正火的脚印,就可以不进行归通过设置执行足迹normMethod = "None"的plotFootprints()功能。
plotFootprints(
seFoot = seFoot,
ArchRProj = projHeme5,
normMethod = "None",
plotName = "Footprints-No-Normalization",
addDOC = FALSE,
smoothWindow = 5
)
没有归一化的图案足迹示例如下所示。
14.3功能足迹
除了覆盖图案之外,ArchR还可以覆盖任何用户定义的功能集。为了说明这种功能,我们将使用该plotFootprints()功能来创建TSS插入配置文件(先前在数据质量控制部分中介绍过)。TSS插入配置文件只是占位的特殊子情况。
如上一部分所述,使用从伪批量复制中导出的组覆盖文件来执行覆盖。我们最初在上一章中创建了这些函数以执行峰值调用。如果您尚未将网上论坛报道添加到中ArchRProject,请立即进行。
projHeme5 <- addGroupCoverages(ArchRProj = projHeme5, groupBy = "Clusters2")
我们创建的TSS插入配置文件没有对Tn5偏差进行归一化。与我们之前的分析的主要区别在于,我们指定flank = 2000在每个TSS的每一侧将这些足迹扩展2000 bp。
seTSS <- getFootprints(
ArchRProj = projHeme5,
positions = GRangesList(TSS = getTSS(projHeme5)),
groupBy = "Clusters2",
flank = 2000
)
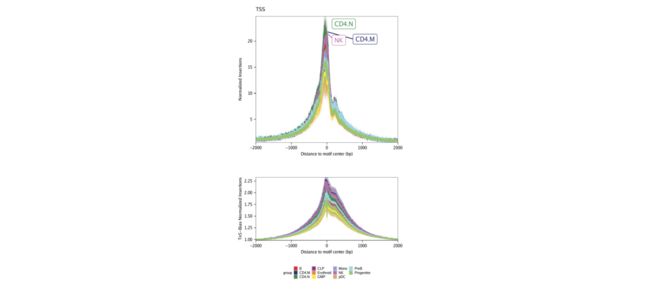
然后,我们可以使用绘制每个细胞组的TSS插入图plotFootprints()。
plotFootprints(
seFoot = seTSS,
ArchRProj = projHeme5,
normMethod = "None",
plotName = "TSS-No-Normalization",
addDOC = FALSE,
flank = 2000,
flankNorm = 100
)
参考材料:
https://www.archrproject.com/