R| brainconn功能连接作图工具
brainconn是2020年新出的一个基于R的工具,实现在R中制作功能连接图,这样就无需导出数据用其他软件作图了。和BrainNet相比还是有一些不完善的地方,不过不影响基本的使用。和其他作图软件一样,比如BrainNet/Nilearn,作功能连接图的主要步骤就是:
定义node
定义edge
调整参数作图
软件很容易上手,只做简介。Vignettes在此:
https://sidchop.github.io/brainconn/articles/brainconn.html
node
软件自带模版有
aal116
aal90
craddock200
dk68
dk82_aspree
dkt62,
gordon_333
shen_268
shen_368
schaefer1000_n7
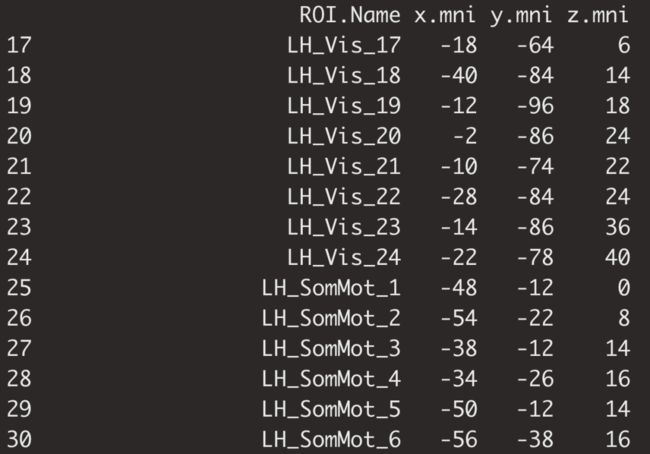
当然也支持自定义node,加载工具包后,可以查看自定义node的例子,并且可以使用check_atlas来检查自定义node是否可用。
custom_atlas <- custom_atlas_example
check_atlas(custom_atlas)自定义node的格式如下:
可以看到,与BrainNet的文件相比少了两列,所以设置不同node的颜色和大小有一点麻烦。注意⚠️使用自定义模板,必须设置node颜色为同一个颜色,否则会报错。
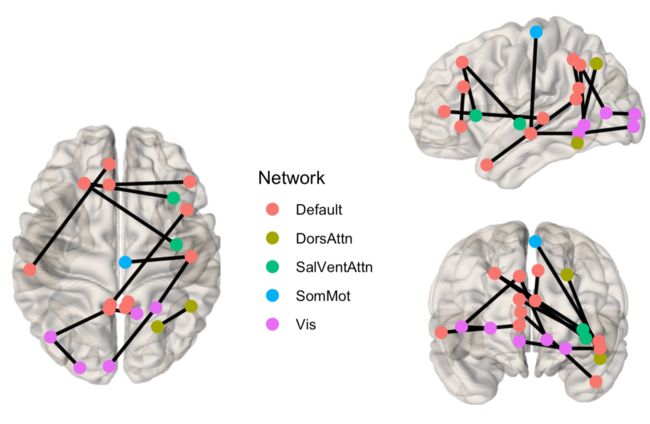
使用软件自带模板,不同network的node默认用不同的颜色
brainconn(atlas ="schaefer300_n7",
conmat=example_unweighted_undirected,
node.size = 3,
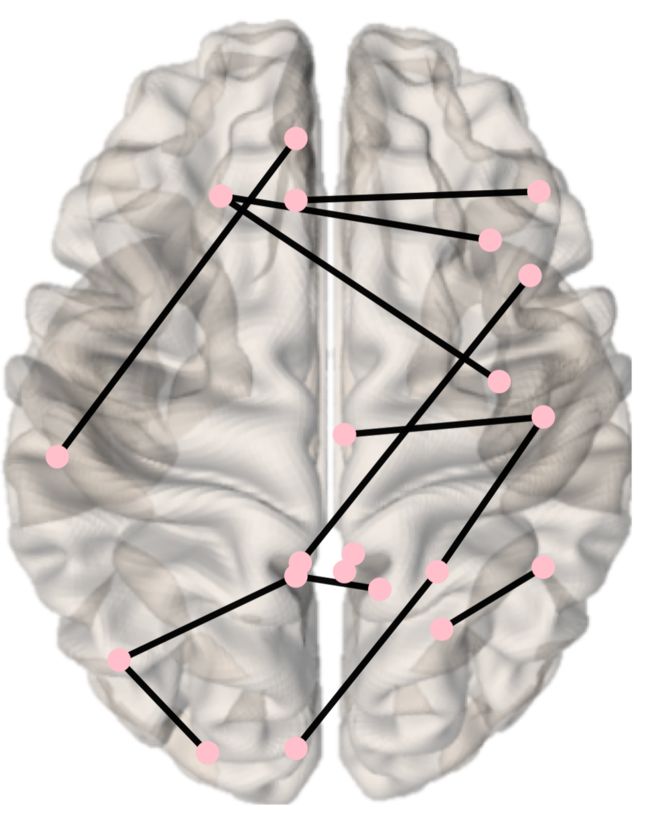
view="ortho")也可以设置node.color为统一的颜色。
brainconn(atlas ="schaefer300_n7",
conmat=example_unweighted_undirected,
node.color = 'pink',
node.size = 3,
view="top")默认设置下,只显示有连接的node。如果需要显示所有node的话,设置all.nodes
brainconn(atlas ="schaefer300_n7",
conmat=example_unweighted_undirected,
view="left",
node.size = 3,
node.color = "hotpink",
edge.width = 2,
edge.color="darkblue",
edge.alpha = 0.8,
all.nodes = T,
show.legend = F)如果要设置每个node的大小,node.size使用vector即可。注意⚠️node.size输入的长度需要和显示的node数量对应。
d=rowSums(example_unweighted_undirected)
d=(d+1)*1.5
brainconn(atlas ="schaefer300_n7",
conmat=example_unweighted_undirected,
node.size = d,
all.nodes = T)对于只显示有连接的node的情况
d=rowSums(example_unweighted_undirected)
d <- d[d != 0] #remove nodes with no edges
d=(d+1)*1.5
brainconn(atlas ="schaefer300_n7",
conmat=example_unweighted_undirected,
node.size = d)edge
无向的edge,看一眼example:
example_unweighted_undirected它是一个300x300的binary对称矩阵,300是网络中node的数量,1代表连接的边。如果有weight,则代表连接的强度。可以根据连接的强度设置edge的大小。
brainconn(atlas ="schaefer300_n7",
conmat=example_weighted_undirected,
node.size = 5,view="bottom",
scale.edge.width = c(1,3),
background.alpha = 0.4,
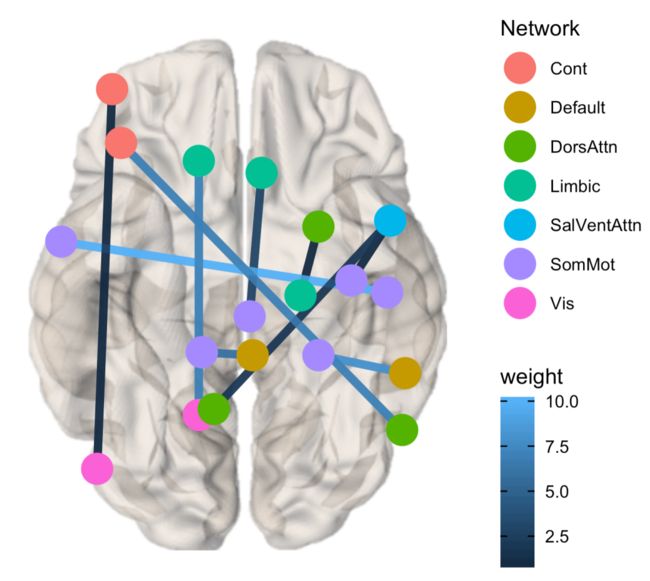
show.legend = F)也可以根据连接的强度设置edge的颜色。
brainconn(atlas ="schaefer300_n7",
conmat=example_weighted_undirected,
node.size = 7,
view="bottom",
edge.width = 2,
edge.color.weighted = T,
show.legend = T)如果连接矩阵是非对称的说明是有向连接,作图方法和无向连接相同。
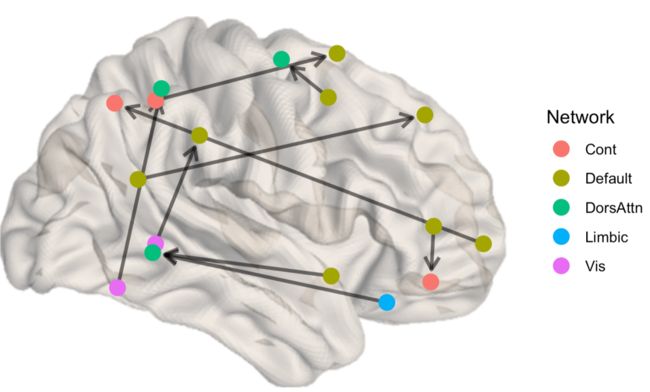
brainconn(atlas ="schaefer300_n7",
conmat=example_unweighted_directed,
node.size = 4,
view="right",
edge.alpha = 0.6)注意⚠️,这里如果设置node.color的话,会显示所有的node,应该是一个小bug。
brainconn(atlas ="schaefer300_n7",
conmat=example_unweighted_directed,
node.size = 4,
node.color = 'pink',
view="right",
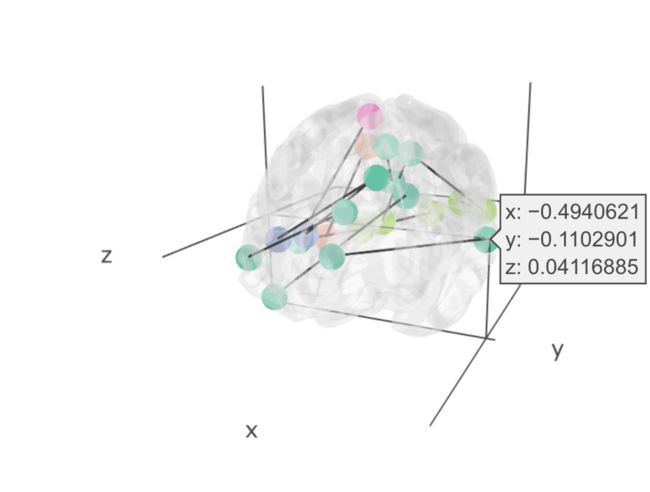
edge.alpha = 0.6)最后它还支持3d的输出和交互。
brainconn3D(atlas ="schaefer300_n7",
conmat=example_unweighted_undirected, show.legend = F)—END—