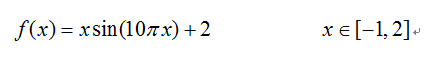遗传算法详解及代码实现
遗传算法
-
- 定义
- 相关术语
-
- 交叉
- 变异
- 产生子代完整过程
- 遗传算法应用
- 问题的提出与解决方案
-
- “袋鼠跳”问题
-
- 爬山法(最速上升爬山法)
- 模拟退火
- 遗传算法
- 遗传算法实现过程
-
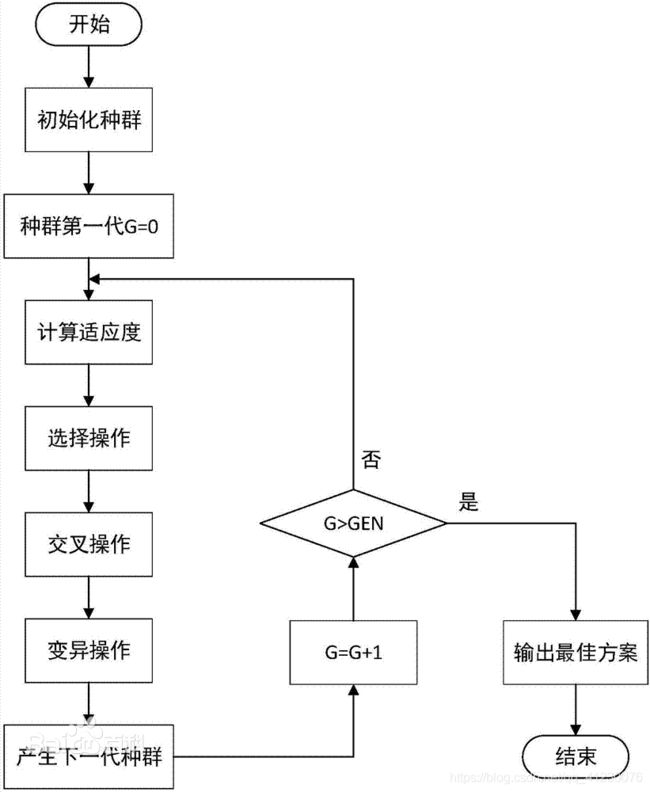
- 遗传算法的一般步骤
- 遗传算法图解
- 进化细节
-
- 种群和个体
- 编码方法
-
- 二进制编码
- 浮点编码法
- 符号编码法
- 袋鼠染色体编码
- 评价个体的适应度--适应度函数(fitness function)
- 射杀一些袋鼠--选择函数(selection)
- 遗传--染色体交叉(crossover)
- 变异--基因突变(Mutation)
- 代码实现
-
- 编码---生成种群
- 解码
- 适应度计算
- 选择
- 交叉、变异
- 完整代码
定义
遗传算法(Genetic Algorithm, GA)起源于对生物系统所进行的计算机模拟研究。它是模仿自然界生物进化机制发展起来的随机全局搜索和优化方法,借鉴了达尔文的进化论和孟德尔的遗传学说。其本质是一种高效、并行、全局搜索的方法,能在搜索过程中自动获取和积累有关搜索空间的知识,并自适应地控制搜索过程以求得最佳解。
相关术语
- 基因型(genotype):性状染色体的内部表现。
- 表现型(phenotype):染色体决定的性状的外部表现,或者说,根据基因型形成的个体的外部表现。
- 进化(evolution):种群逐渐适应生存环境,品质不断得到改良。生物的进化是以种群的形式进行的。
- 适应度(fitness):度量某个物种对于生存环境的适应程度。
- 选择(selection):以一定的概率从种群中选择若干个个体。一般,选择过程是一种基于适应度的优胜劣汰的过程。
- 复制(reproduction):细胞分裂时,遗传物质DNA通过复制而转移到新产生的细胞中,新细胞就继承了旧细胞的基因。
- 交叉(crossover):两个染色体的某一相同位置处DNA被切断,前后两串分别交叉组合形成两个新的染色体。也称基因重组或杂交。
- 变异(mutation):复制时可能(很小的概率)产生某些复制差错,变异产生新的染色体,表现出新的性状。
- 编码(coding):DNA中遗传信息在一个长链上按一定的模式排列。遗传编码可看作从表现型到基因型的映射。
- 解码(decoding):基因型到表现型的映射。
- 个体(individual):指染色体带有特征的实体。
- 种群(population):个体的集合,该集合内个体数称为种群的大小。
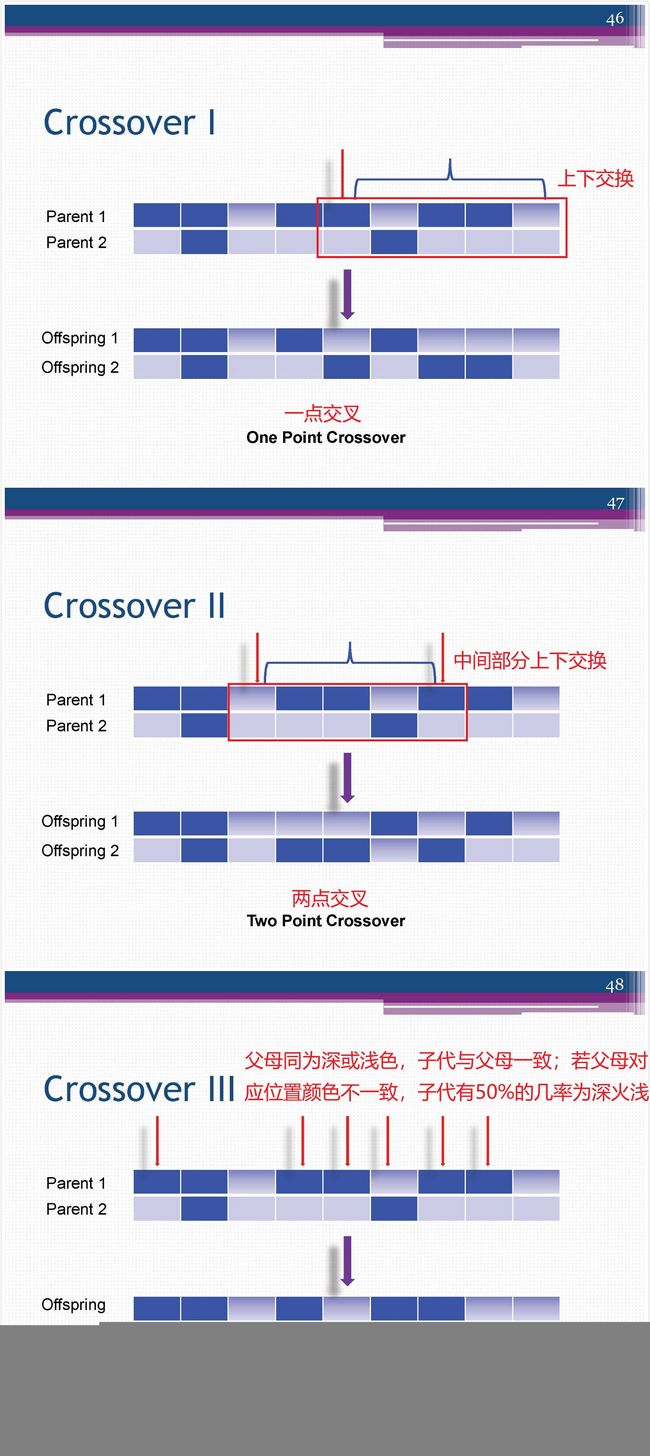
交叉
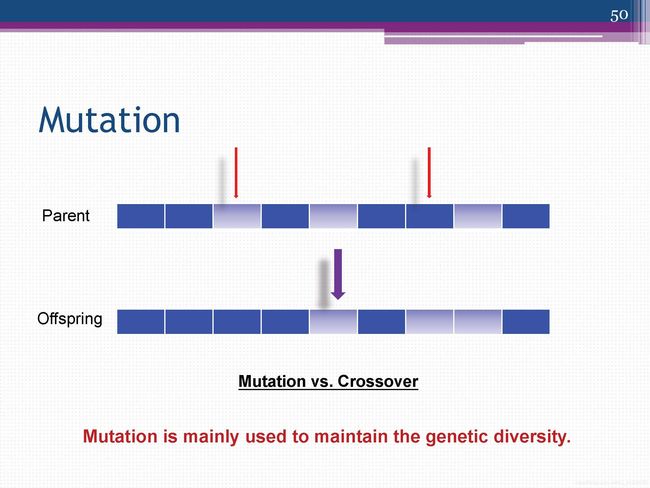
变异
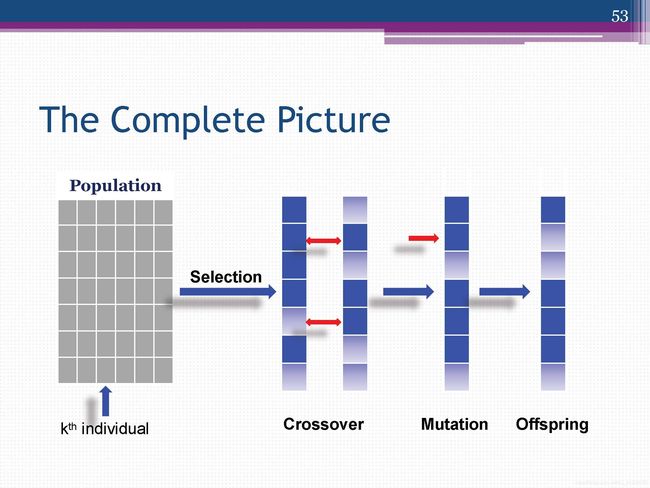
产生子代完整过程
遗传算法应用
遗传算法的有趣应用很多,诸如寻路问题,8数码问题,囚犯困境,动作控制,找圆心问题(在一个不规则的多边形中,寻找一个包含在该多边形内的最大圆圈的圆心),TSP问题,生产调度问题,人工生命模拟等。下面以袋鼠为例子讲讲遗传算法。
遗传算法中每一条染色体,对应着遗传算法的一个解决方案,一般我们用适应性函数(fitness function)来衡量这个解决方案的优劣。所以从一个基因组到其解的适应度形成一个映射。可以把遗传算法的过程看作是一个在多元函数里面求最优解的过程。可以这样想象,这个多维曲面里面有数不清的“山峰”,而这些山峰所对应的就是局部最优解。而其中也会有一个“山峰”的海拔最高的,那么这个就是全局最优解。而遗传算法的任务就是尽量爬到最高峰,而不是陷落在一些小山峰。(另外,值得注意的是遗传算法不一定要找“最高的山峰”,如果问题的适应度评价越小越好的话,那么全局最优解就是函数的最小值,对应的,遗传算法所要找的就是“最深的谷底”)
问题的提出与解决方案
“袋鼠跳”问题
既然我们把函数曲线理解成一个一个山峰和山谷组成的山脉。那么我们可以设想所得到的每一个解就是一只袋鼠,我们希望它们不断的向着更高处跳去,直到跳到最高的山峰(尽管袋鼠本身不见得愿意那么做)。所以求最大值的过程就转化成一个“袋鼠跳”的过程。
作为对比下面简单介绍“袋鼠跳”的几种方式。
爬山法(最速上升爬山法)
从搜索空间中随机产生邻近的点,从中选择对应解最优的个体,替换原来的个体,不断重复上述过程。因为爬山法只对“邻近”的点作比较,所以目光比较“短浅”,常常只能收敛到离开初始位置比较近的局部最优解上面。对于存在很多局部最优点的问题,通过一个简单的迭代找出全局最优解的机会非常渺茫。(在爬山法中,袋鼠最有希望到达最靠近它出发点的山顶,但不能保证该山顶是珠穆朗玛峰,或者是一个非常高的山峰。因为一路上它只顾上坡,没有下坡。)
模拟退火
这个方法来自金属热加工过程的启发。在金属热加工过程中,当金属的温度超过它的熔点(Melting Point)时,原子就会激烈地随机运动。与所有的其它的物理系统相类似,原子的这种运动趋向于寻找其能量的极小状态。在这个能量的变迁过程中,开始时,温度非常高, 使得原子具有很高的能量。随着温度不断降低,金属逐渐冷却,金属中的原子的能量就越来越小,最后达到所有可能的最低点。利用模拟退火的时候,让算法从较大的跳跃开始,使到它有足够的“能量”逃离可能“路过”的局部最优解而不至于限制在其中,当它停在全局最优解附近的时候,逐渐的减小跳跃量,以便使其“落脚 ”到全局最优解上。(在模拟退火中,袋鼠喝醉了,而且随机地大跳跃了很长时间。运气好的话,它从一个山峰跳过山谷,到了另外一个更高的山峰上。但最后,它渐渐清醒了并朝着它所在的峰顶跳去。)
遗传算法
模拟物竞天择的生物进化过程,通过维护一个潜在解的群体执行了多方向的搜索,并支持这些方向上的信息构成和交换。是以面为单位的搜索,比以点为单位的搜索,更能发现全局最优解。(在遗传算法中,有很多袋鼠,它们降落到喜玛拉雅山脉的任意地方。这些袋鼠并不知道它们的任务是寻找珠穆朗玛峰。但每过几年,就在一些海拔高度较低的地方射杀一些袋鼠,并希望存活下来的袋鼠是多产的,在它们所处的地方生儿育女。)(或者换个说法。从前,有一大群袋鼠,它们被莫名其妙的零散地遗弃于喜马拉雅山脉。于是只好在那里艰苦的生活。海拔低的地方弥漫着一种无色无味的毒气,海拔越高毒气越稀薄。可是可怜的袋鼠们对此全然不觉,还是习惯于活蹦乱跳。于是,不断有袋鼠死于海拔较低的地方,而越是在海拔高的袋鼠越是能活得更久,也越有机会生儿育女。就这样经过许多年,这些袋鼠们竟然都不自觉地聚拢到了一个个的山峰上,可是在所有的袋鼠中,只有聚拢到珠穆朗玛峰的袋鼠被带回了美丽的澳洲。)
遗传算法的实现过程
遗传算法实现过程
遗传算法的实现过程实际上就像自然界的进化过程那样。首先寻找一种对问题潜在解进行“数字化”编码的方案。(建立表现型和基因型的映射关系)然后用随机数初始化一个种群(那么第一批袋鼠就被随意地分散在山脉上),种群里面的个体就是这些数字化的编码。接下来,通过适当的解码过程之后(得到袋鼠的位置坐标),用适应性函数对每一个基因个体作一次适应度评估(袋鼠爬得越高,越是受我们的喜爱,所以适应度相应越高)。用选择函数按照某种规定择优选择(我们要每隔一段时间,在山上射杀一些所在海拔较低的袋鼠,以保证袋鼠总体数目持平。)。让个体基因变异(让袋鼠随机地跳一跳)。然后产生子代(希望存活下来的袋鼠是多产的,并在那里生儿育女)。遗传算法并不保证你能获得问题的最优解,但是使用遗传算法的最大优点在于你不必去了解和操心如何去“找”最优解。(你不必去指导袋鼠向那边跳,跳多远。)而只要简单的“否定”一些表现不好的个体就行了。(把那些总是爱走下坡路的袋鼠射杀,这就是遗传算法的精粹!)
遗传算法的一般步骤
遗传算法中每一条染色体,对应着遗传算法的一个解决方案,一般我们用适应性函数(fitness function)来衡量这个解决方案的优劣。所以从一个基因组到其解的适应度形成一个映射。遗传算法的实现过程实际上就像自然界的进化过程那样。
下面我们用袋鼠跳中的步骤一一对应解释,以方便大家理解:
1.首先寻找一种对问题潜在解进行“数字化”编码的方案。(建立表现型和基因型的映射关系)
2.随机初始化一个种群(那么第一批袋鼠就被随意地分散在山脉上),种群里面的个体就是这些数字化的编码
3.接下来,通过适当的解码过程之后(得到袋鼠的位置坐标)
4.用适应性函数对每一个基因个体作一次适应度评估(袋鼠爬得越高当然就越好,所以适应度相应越高)
5.用选择函数按照某种规定择优选择(每隔一段时间,射杀一些所在海拔较低的袋鼠,以保证袋鼠总体数目持平)
让个体基因变异(让袋鼠随机地跳一跳)
6.然后产生子代(希望存活下来的袋鼠是多产的,并在那里生儿育女)
由此我们可以得出遗传算法的一般步骤:
1.随机产生种群
2.根据策略判断个体的适应度,是否符合优化准则,若符合,输出最佳个体及其最优解,结束。否则,进行下一步
3.依据适应度选择父母,适应度高的个体被选中的概率高,适应度低的个体被淘汰
4.用父母的染色体按照一定的方法进行交叉,生成子代
5.对子代染色体进行变异
由交叉和变异产生新一代种群,返回步骤2,直到最优解产生
遗传算法图解
进化细节
种群和个体
遗传算法启发自进化理论,而我们知道进化是由种群为单位的,种群是什么呢?维基百科上解释为:在生物学上,是在一定空间范围内同时生活着的同种生物的全部个体。显然要想理解种群的概念,又先得理解个体的概念,在遗传算法里,个体通常为某个问题的一个解,并且该解在计算机中被编码为一个向量表示! 我们的例子中要求最大值,所以该问题的解为一组可能的(x,y) (x, y)(x,y)的取值。比如(x=2.1,y=0.8),(x=−1.5,y=2.3)… 就是求最大值问题的一个可能解,也就是遗传算法里的个体,把这样的一组一组的可能解的集合就叫做种群 ,比如在这个问题中设置100个这样的x,y 的可能的取值对,这100个个体就构成了种群。
编码方法
在上面个体概念里提到个体(也就是一组可能解)在计算机程序中被编码为一个向量表示,而在我们这个问题中,个体是x,y 的取值,是两个实数,所以问题就可以转化为如何将实数编码为一个向量表示,可能有些朋友有疑惑,实数在计算机里不是可以直接存储吗,为什么需要编码呢?这里编码是为了后续操作(交叉和变异)的方便。
生物的DNA有四种碱基对,分别是ACGT,DNA的编码可以看作是DNA上碱基对的不同排列,不同的排列使得基因的表现出来的性状也不同(如单眼皮双眼皮)。在计算机中,我们可以模仿这种编码,但是碱基对的种类只有两种,分别是0,1。只要我们能够将不同的实数表示成不同的0,1二进制串表示就完成了编码,也就是说其实我们并不需要去了解一个实数对应的二进制具体是多少,我们只需要保证有一个映射
y=f(x),x is decimal system,y is binary system
能够将十进制的数编码为二进制即可,至于这个映射是什么,其实可以不必关心。将个体(可能解)编码后的二进制串叫做染色体,染色体(或者有人叫DNA)就是个体(可能解)的二进制编码表示。为什么可以不必关心映射f(x)呢?因为其实我们在程序中操纵的都是二进制串,而二进制串生成时可以随机生成。
编码是应用遗传算法时要解决的首要问题,也是设计遗传算法时的一个关键步骤。编码方法影响到交叉算子、变异算子等遗传算子的运算方法,大很大程度上决定了遗传进化的效率。
迄今为止人们已经提出了许多种不同的编码方法。总的来说,这些编码方法可以分为三大类:二进制编码法、浮点编码法、符号编码法。下面分别进行介绍:
二进制编码
就像人类的基因有AGCT 4种碱基序列一样。不过在这里我们只用了0和1两种碱基,然后将他们串成一条链形成染色体。一个位能表示出2种状态的信息量,因此足够长的二进制染色体便能表示所有的特征。这便是二进制编码。如下:
1110001010111
它由二进制符号0和1所组成的二值符号集。有以下一些优点:
1.编码、解码操作简单易行
2.交叉、变异等遗传操作便于实现
3.符合最小字符集编码原则
4.利用模式定理对算法进行理论分析
缺点:对于一些连续函数的优化问题,由于其随机性使得其局部搜索能力较差,如对于一些高精度的问题,当解迫近于最优解后,由于其变异后表现型变化很大(比如00000变异成10000等),不连续,所以会远离最优解,达不到稳定。
浮点编码法
二进制编码虽然简单直观,但明显地存在着连续函数离散化时的映射误差。个体长度较短时,可能达不到精度要求,而个体编码长度较长时,虽然能提高精度,但增加了解码的难度,使遗传算法的搜索空间急剧扩大。
所谓浮点法,是指个体的每个基因值用某一范围内的一个浮点数来表示。在浮点数编码方法中,必须保证基因值在给定的区间限制范围内,遗传算法中所使用的交叉、变异等遗传算子也必须保证其运算结果所产生的新个体的基因值也在这个区间限制范围内。如下所示:
1.2-3.2-5.3-7.2-1.4-9.7
浮点数编码方法有下面几个优点:
1.适用于在遗传算法中表示范围较大的数
2.适用于精度要求较高的遗传算法
3.便于较大空间的遗传搜索
4.改善了遗传算法的计算复杂性,提高了运算交率
5.便于遗传算法与经典优化方法的混合使用
6.便于设计针对问题的专门知识的知识型遗传算子
7.便于处理复杂的决策变量约束条件
符号编码法
符号编码法是指个体染色体编码串中的基因值取自一个无数值含义、而只有代码含义的符号集如{A,B,C…}。
符号编码的主要优点是:
1.符合有意义积术块编码原则
2.便于在遗传算法中利用所求解问题的专门知识
3.便于遗传算法与相关近似算法之间的混合使用。
袋鼠染色体编码
在上面介绍了一系列编码方式以后,那么,如何利用上面的编码来为我们的袋鼠染色体编码呢?首先我们要明确一点:编码无非就是建立从基因型到表现型的映射关系。这里的表现型可以理解为个体特征(比如身高、体重、毛色等等)。那么,在此问题下,我们关心的个体特征就是:袋鼠的位置坐标(因为我们要把海拔低的袋鼠给杀掉)。无论袋鼠长什么样,爱吃什么。我们关心的始终是袋鼠在哪里,并且只要知道了袋鼠的位置坐标(位置坐标就是相应的染色体编码,可以通过解码得出),我们就可以:
1.在喜马拉雅山脉的地图上找到相应的位置坐标,算出海拔高度。(相当于通过自变量求得适应函数的值)然后判读该不该射杀该袋鼠。
2.可以知道染色体交叉和变异后袋鼠新的位置坐标。
上文所提的求一元函数最大值的问题。在上面我们把极大值比喻为山峰,那么,袋鼠的位置坐标可以比喻为区间[-1, 2]的某一个x坐标(有了x坐标,再通过函数表达式可以算出函数值 <==> 得到了袋鼠染色体编码,解码得到位置坐标,在喜马拉雅山脉地图查询位置坐标算出海拔高度)。这个x坐标是一个实数,现在,说白了就是怎么对这个x坐标进行编码。下面以二进制编码为例,不过这种情况下以二进制编码比较复杂。
一定长度的二进制编码序列,只能表示一定精度的浮点数。在这里假如我们要求解精确到六位小数,由于区间为[-1,2],所以长度为3 ,为了保证精度要求,至少把区间[-1,2]分为3 × 10^6等份。又因为
![]()
所以编码的二进制串至少需要22位。
把一个二进制串(b0,b1,…bn)转化位区间里面对应的实数值通过下面两个步骤。
1.将一个二进制串代表的二进制数转化为10进制数:
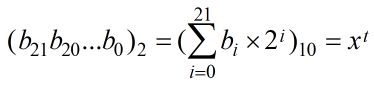
2.对应区间内的实数:
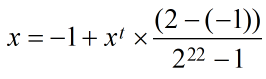
例如一个二进制串<1000101110110101000111>表示实数值0.637197
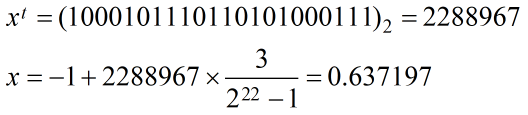
以上编码方式只是举个例子便于理解,编码的方式千奇百怪,层出不穷,每个问题可能采用的编码方式都不一样。在这一点上大家要注意。
评价个体的适应度–适应度函数(fitness function)
适应度函数主要是通过个体特征从而判断个体的适应度。在本例的袋鼠跳中,我们只关心袋鼠的海拔高度,以此来判断是否该射杀该袋鼠。这样一来,该函数就非常简单了。只要输入袋鼠的位置坐标,在通过相应查找运算,返回袋鼠当前位置的海拔高度就行。
适应度函数也称评价函数,是根据目标函数确定的用于区分群体中个体好坏的标准。适应度函数总是非负的,而目标函数可能有正有负,故需要在目标函数与适应度函数之间进行变换。
评价个体适应度的一般过程为:
1.对个体编码串进行解码处理后,可得到个体的表现型。
2.由个体的表现型可计算出对应个体的目标函数值
根据最优化问题的类型,由目标函数值按一定的转换规则求出个体的适应度
射杀一些袋鼠–选择函数(selection)
遗传算法中的选择操作就是用来确定如何从父代群体中按某种方法选取那些个体,以便遗传到下一代群体。选择操作用来确定重组或交叉个体,以及被选个体将产生多少个子代个体。前面说了,我们希望海拔高的袋鼠存活下来,并尽可能繁衍更多的后代。但我们都知道,在自然界中,适应度高的袋鼠越能繁衍后代,但这也是从概率上说的而已。毕竟有些适应度低的袋鼠也可能逃过我们的眼睛。
那么,怎么建立这种概率关系呢?
下面介绍几种常用的选择算子:
轮盘赌选择(Roulette Wheel Selection):是一种回放式随机采样方法。每个个体进入下一代的概率等于它的适应度值与整个种群中个体适应度值和的比例。选择误差较大。
随机竞争选择(Stochastic Tournament):每次按轮盘赌选择一对个体,然后让这两个个体进行竞争,适应度高的被选中,如此反复,直到选满为止。
最佳保留选择:首先按轮盘赌选择方法执行遗传算法的选择操作,然后将当前群体中适应度最高的个体结构完整地复制到下一代群体中。
无回放随机选择(也叫期望值选择Excepted Value Selection):根据每个个体在下一代群体中的生存期望来进行随机选择运算。方法如下:
(1) 计算群体中每个个体在下一代群体中的生存期望数目N。
(2) 若某一个体被选中参与交叉运算,则它在下一代中的生存期望数目减去0.5,若某一个体未 被选中参与交叉运算,则它在下一代中的生存期望数目减去1.0。
(3) 随着选择过程的进行,若某一个体的生存期望数目小于0时,则该个体就不再有机会被选中。
确定式选择:按照一种确定的方式来进行选择操作。具体操作过程如下:
(1) 计算群体中各个个体在下一代群体中的期望生存数目N。
(2) 用N的整数部分确定各个对应个体在下一代群体中的生存数目。
(3) 用N的小数部分对个体进行降序排列,顺序取前M个个体加入到下一代群体中。至此可完全确定出下一代群体中M个个体。
无回放余数随机选择:可确保适应度比平均适应度大的一些个体能够被遗传到下一代群体中,因而选择误差比较小。
均匀排序:对群体中的所有个体按期适应度大小进行排序,基于这个排序来分配各个个体被选中的概率。
最佳保存策略:当前群体中适应度最高的个体不参与交叉运算和变异运算,而是用它来代替掉本代群体中经过交叉、变异等操作后所产生的适应度最低的个体。
随机联赛选择:每次选取几个个体中适应度最高的一个个体遗传到下一代群体中。
排挤选择:新生成的子代将代替或排挤相似的旧父代个体,提高群体的多样性。
以轮盘赌选择为例:
假如有5条染色体,他们的适应度分别为5、8、3、7、2。
那么总的适应度为:F = 5 + 8 + 3 + 7 + 2 = 25。
那么各个个体的被选中的概率为:
α1 = ( 5 / 25 ) * 100% = 20%
α2 = ( 8 / 25 ) * 100% = 32%
α3 = ( 3 / 25 ) * 100% = 12%
α4 = ( 7 / 25 ) * 100% = 28%
α5 = ( 2 / 25 ) * 100% = 8%
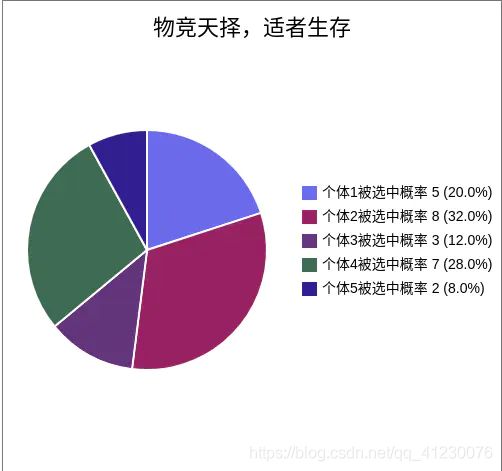
当指针在这个转盘上转动,停止下来时指向的个体就是天选之人啦。可以看出,适应性越高的个体被选中的概率就越大。
遗传–染色体交叉(crossover)
遗传算法的交叉操作,是指对两个相互配对的染色体按某种方式相互交换其部分基因,从而形成两个新的个体。
适用于二进制编码个体或浮点数编码个体的交叉算子:
1.单点交叉(One-point Crossover):指在个体编码串中只随机设置一个交叉点,然后再该点相互交换两个配对个体的部分染色体。
2.两点交叉与多点交叉:
(1) 两点交叉(Two-point Crossover):在个体编码串中随机设置了两个交叉点,然后再进行部分基因交换。
(2) 多点交叉(Multi-point Crossover)
3.均匀交叉(也称一致交叉,Uniform Crossover):两个配对个体的每个基因座上的基因都以相同的交叉概率进行交换,从而形成两个新个体。
4.算术交叉(Arithmetic Crossover):由两个个体的线性组合而产生出两个新的个体。该操作对象一般是由浮点数编码表示的个体。
变异–基因突变(Mutation)
遗传算法中的变异运算,是指将个体染色体编码串中的某些基因座上的基因值用该基因座上的其它等位基因来替换,从而形成新的个体。
例如下面这串二进制编码:
101101001011001
经过基因突变后,可能变成以下这串新的编码:
001101011011001
以下变异算子适用于二进制编码和浮点数编码的个体:
基本位变异(Simple Mutation):对个体编码串中以变异概率、随机指定的某一位或某几位仅因座上的值做变异运算。
均匀变异(Uniform Mutation):分别用符合某一范围内均匀分布的随机数,以某一较小的概率来替换个体编码串中各个基因座上的原有基因值。(特别适用于在算法的初级运行阶段)
边界变异(Boundary Mutation):随机的取基因座上的两个对应边界基因值之一去替代原有基因值。特别适用于最优点位于或接近于可行解的边界时的一类问题。
非均匀变异:对原有的基因值做一随机扰动,以扰动后的结果作为变异后的新基因值。对每个基因座都以相同的概率进行变异运算之后,相当于整个解向量在解空间中作了一次轻微的变动。
高斯近似变异:进行变异操作时用符号均值为P的平均值,方差为P**2的正态分布的一个随机数来替换原有的基因值。
代码实现
问题引入
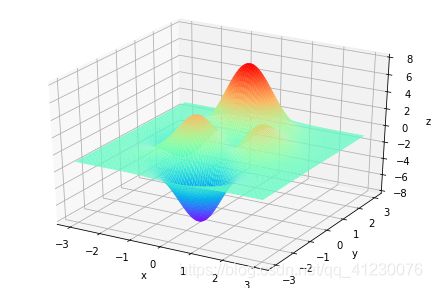
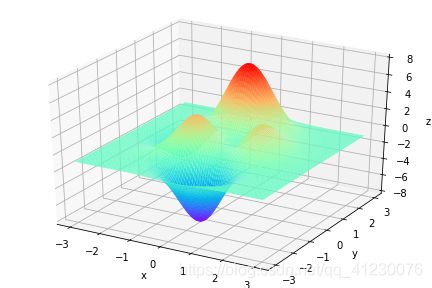
上面提到遗传算法是用来解决最优化问题的,下面在x∈[−3,3],y∈[−3,3] 范围里的最大值为例子来详细讲解遗传算法的每一步。
def F(x, y):
return 3*(1-x)**2*np.exp(-(x**2)-(y+1)**2)- 10*(x/5 - x**3 - y**5)*np.exp(-x**2-y**2)- 1/3**np.exp(-(x+1)**2 - y**2)
编码—生成种群
#pop表示种群矩阵,一行表示一个二进制编码表示的DNA,矩阵的行数为种群数目,DNA_SIZE为编码长度,函数涉及到两个变量,总长度为DNA_SIZE*2
pop = np.random.randint(2, size=(POP_SIZE, DNA_SIZE*2)) #matrix (POP_SIZE, DNA_SIZE*2)
解码
目标是求一个逆映射将这个二进制串映射到x,y∈[−3,3]
首先设定X,Y的取值范围
X_BOUND = [-3, 3] #x取值范围
Y_BOUND = [-3, 3] #y取值范围
1.首先获取X,Y,将二进制串按权展开,将二进制数转化为十进制数
x_pop = pop[:,1::2]#奇数列表示X,从索引列1开始,加入了步长2
y_pop = pop[:,::2] #偶数列表示y,从索引列1开始,加入了步长2
x_pop= x_pop.dot(2**np.arange(DNA_SIZE)[::-1])
y_pop= y_pop.dot(2**np.arange(DNA_SIZE)[::-1])
其中,np.arange(DNA_SIZE)[::-1]类似于倒序输出,比如[1,2,3,4]–>[4,3,2,1],然后将二进制串按权展开。
2.将转换后的实数压缩到[0,1]之间的一个小数
x_pop= x_pop/float(2**DNA_SIZE-1)
y_pop= y_pop/float(2**DNA_SIZE-1)
3.将[0,1] 区间内的数映射到我们要的区间[-3,3]即可
#X_BOUND,Y_BOUND是x,y的取值范围 X_BOUND = [-3, 3], Y_BOUND = [-3, 3],
x= x_pop* (X_BOUND[1] - X_BOUND[0]) + X_BOUND[0] #映射为x范围内的数
y= y_pop* (Y_BOUND[1] - Y_BOUND[0]) + Y_BOUND[0] #映射为y范围内的数
以下为解码过程的python代码:
DNA_SIZE=24(一个实数DNA长度),两个实数x,y一共用48位二进制编码,同时将x和y编码到同一个48位的二进制串里,每一个变量用24位表示,奇数24列为x的编码表示,偶数24列为y的编码表示。
def translateDNA(pop):#pop表示种群矩阵,一行表示一个二进制编码表示的DNA,矩阵的行数为种群数目
x_pop = pop[:,1::2]#奇数列表示X
y_pop = pop[:,::2] #偶数列表示y
#pop:(POP_SIZE,DNA_SIZE)*(DNA_SIZE,1) --> (POP_SIZE,1)完成解码
x = x_pop.dot(2**np.arange(DNA_SIZE)[::-1])/float(2**DNA_SIZE-1)*(X_BOUND[1]-X_BOUND[0])+X_BOUND[0]
y = y_pop.dot(2**np.arange(DNA_SIZE)[::-1])/float(2**DNA_SIZE-1)*(Y_BOUND[1]-Y_BOUND[0])+Y_BOUND[0]
return x,y
适应度计算
我们已经得到了一个种群,现在要根据适者生存规则把优秀的个体保存下来,同时淘汰掉那些不适应环境的个体。现在摆在我们面前的问题是如何评价一个个体对环境的适应度?在我们的求最大值的问题中可以直接用可能解(个体)对应的函数的函数值的大小来评估,这样可能解对应的函数值越大越有可能被保留下来,以求解上面定义的函数F的最大值为例,python代码如下:
def get_fitness(pop):
x,y = translateDNA(pop)
pred = F(x, y)
return (pred - np.min(pred)) + 1e-3 #减去最小的适应度是为了防止适应度出现负数,通过这一步fitness的范围为[0, np.max(pred)-np.min(pred)],最后在加上一个很小的数防止出现为0的适应度
pred是将可能解带入函数F中得到的预测值,因为后面的选择过程需要根据个体适应度确定每个个体被保留下来的概率,而概率不能是负值,所以减去预测中的最小值把适应度值的最小区间提升到从0开始,但是如果适应度为0,其对应的概率也为0,表示该个体不可能在选择中保留下来,这不符合算法思想,遗传算法不绝对否定谁也不绝对肯定谁,所以最后加上了一个很小的正数。
有了求最大值的适应度函数求最小值适应度函数也就容易了,python代码如下:
def get_fitness(pop):
x,y = translateDNA(pop)
pred = F(x, y)
return -(pred - np.max(pred)) + 1e-3
根据适者生存规则在求最小值问题上,函数值越小的可能解对应的适应度应该越大,同时适应度也不能为负值,先将适应度减去最大预测值,将适应度可能取值区间压缩为[np.min(pred−np.max(pred),0],然后添加个负号将适应度变为正数,同理为了不出现0,最后在加上一个很小的正数。
选择
有了评估的适应度函数,下面可以根据适者生存法则将优秀者保留下来了。选择则是根据新个体的适应度进行,但同时不意味着完全以适应度高低为导向(选择top k个适应度最高的个体,容易陷入局部最优解),因为单纯选择适应度高的个体将可能导致算法快速收敛到局部最优解而非全局最优解,我们称之为早熟。作为折中,遗传算法依据原则:适应度越高,被选择的机会越高,而适应度低的,被选择的机会就低。 在python中可以写做:
def select(pop, fitness): # nature selection wrt pop's fitness
idx = np.random.choice(np.arange(POP_SIZE), size=POP_SIZE, replace=True,
p=(fitness)/(fitness.sum()) )
return pop[idx]
np.random.choice()函数的参数p描述了从np.arange(POP_SIZE)里选择每一个元素的概率,概率越高约有可能被选中,最后返回被选中的个体即可。
交叉、变异
通过选择得到了当前看来“还不错的基因”,但是这并不是最好的基因,我们需要通过繁殖后代(包含有交叉+变异过程)来产生比当前更好的基因,但是繁殖后代并不能保证每个后代个体的基因都比上一代优秀,这时需要继续通过选择过程来让试应环境的个体保留下来,从而完成进化,不断迭代上面这个过程种群中的个体就会一步一步地进化。
具体地繁殖后代过程包括交叉和变异两步。交叉是指每一个个体是由父亲和母亲两个个体繁殖产生,子代个体的DNA(二进制串)获得了一半父亲的DNA,一半母亲的DNA,但是这里的一半并不是真正的一半,这个位置叫做交配点,是随机产生的,可以是染色体的任意位置。通过交叉子代获得了一半来自父亲一半来自母亲的DNA,但是子代自身可能发生变异,使得其DNA即不来自父亲,也不来自母亲,在某个位置上发生随机改变,通常就是改变DNA的一个二进制位(0变到1,或者1变到0)。
交叉和变异不是必然发生,而是有一定概率发生。先考虑交叉,最坏情况,交叉产生的子代的DNA都比父代要差(这样算法有可能朝着优化的反方向进行,不收敛),如果交叉是有一定概率不发生,那么就能保证子代有一部分基因和当前这一代基因水平一样;而变异本质上是让算法跳出局部最优解,如果变异时常发生,或发生概率太大,那么算法到了最优解时还会不稳定。交叉概率,范围一般是0.6~1,突变常数(又称为变异概率),通常是0.1或者更小。
def crossover_and_mutation(pop, CROSSOVER_RATE = 0.8):
new_pop = []
for father in pop: #遍历种群中的每一个个体,将该个体作为父亲
child = father #孩子先得到父亲的全部基因(这里我把一串二进制串的那些0,1称为基因)
if np.random.rand() < CROSSOVER_RATE: #产生子代时不是必然发生交叉,而是以一定的概率发生交叉
mother = pop[np.random.randint(POP_SIZE)] #再种群中选择另一个个体,并将该个体作为母亲
cross_points = np.random.randint(low=0, high=DNA_SIZE*2) #随机产生交叉的点
child[cross_points:] = mother[cross_points:] #孩子得到位于交叉点后的母亲的基因
mutation(child) #每个后代有一定的机率发生变异
new_pop.append(child)
return new_pop
#变异
def mutation(child, MUTATION_RATE=0.003):
if np.random.rand() < MUTATION_RATE: #以MUTATION_RATE的概率进行变异
mutate_point = np.random.randint(0, DNA_SIZE*2) #随机产生一个实数,代表要变异基因的位置
child[mutate_point] = child[mutate_point]^1 #将变异点的二进制为反转
完整代码
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import cm
from mpl_toolkits.mplot3d import Axes3D
DNA_SIZE = 24
POP_SIZE = 200
CROSSOVER_RATE = 0.8
MUTATION_RATE = 0.005
N_GENERATIONS = 50
X_BOUND = [-3, 3]
Y_BOUND = [-3, 3]
def F(x, y):
return 3*(1-x)**2*np.exp(-(x**2)-(y+1)**2)- 10*(x/5 - x**3 - y**5)*np.exp(-x**2-y**2)- 1/3**np.exp(-(x+1)**2 - y**2)
def plot_3d(ax):
X = np.linspace(*X_BOUND, 100)
Y = np.linspace(*Y_BOUND, 100)
X,Y = np.meshgrid(X, Y)
Z = F(X, Y)
ax.plot_surface(X,Y,Z,rstride=1,cstride=1,cmap=cm.coolwarm)
ax.set_zlim(-10,10)
ax.set_xlabel('x')
ax.set_ylabel('y')
ax.set_zlabel('z')
plt.pause(3)
plt.show()
def get_fitness(pop):
x,y = translateDNA(pop)
pred = F(x, y)
return (pred - np.min(pred)) #减去最小的适应度是为了防止适应度出现负数,通过这一步fitness的范围为[0, np.max(pred)-np.min(pred)]
def translateDNA(pop): #pop表示种群矩阵,一行表示一个二进制编码表示的DNA,矩阵的行数为种群数目
x_pop = pop[:,1::2]#奇数列表示X,从索引列1开始,加入了步长2
y_pop = pop[:,::2] #偶数列表示y,从索引列1开始,加入了步长2
#pop:(POP_SIZE,DNA_SIZE)*(DNA_SIZE,1) --> (POP_SIZE,1)
x = x_pop.dot(2**np.arange(DNA_SIZE)[::-1])/float(2**DNA_SIZE-1)*(X_BOUND[1]-X_BOUND[0])+X_BOUND[0]
y = y_pop.dot(2**np.arange(DNA_SIZE)[::-1])/float(2**DNA_SIZE-1)*(Y_BOUND[1]-Y_BOUND[0])+Y_BOUND[0]
return x,y
def crossover_and_mutation(pop, CROSSOVER_RATE = 0.8):
new_pop = []
for father in pop: #遍历种群中的每一个个体,将该个体作为父亲
child = father #孩子先得到父亲的全部基因(这里我把一串二进制串的那些0,1称为基因)
if np.random.rand() < CROSSOVER_RATE: #产生子代时不是必然发生交叉,而是以一定的概率发生交叉
mother = pop[np.random.randint(POP_SIZE)] #再种群中选择另一个个体,并将该个体作为母亲
cross_points = np.random.randint(low=0, high=DNA_SIZE*2) #随机产生交叉的点
child[cross_points:] = mother[cross_points:] #孩子得到位于交叉点后的母亲的基因
mutation(child) #每个后代有一定的机率发生变异
new_pop.append(child)
return new_pop
def mutation(child, MUTATION_RATE=0.003):
if np.random.rand() < MUTATION_RATE: #以MUTATION_RATE的概率进行变异
mutate_point = np.random.randint(0, DNA_SIZE*2) #随机产生一个实数,代表要变异基因的位置
child[mutate_point] = child[mutate_point]^1 #将变异点的二进制为反转
def select(pop, fitness): # nature selection wrt pop's fitness
idx = np.random.choice(np.arange(POP_SIZE), size=POP_SIZE, replace=True,
p=(fitness)/(fitness.sum()) )
return pop[idx]
def print_info(pop):
fitness = get_fitness(pop)
max_fitness_index = np.argmax(fitness)
print("max_fitness:", fitness[max_fitness_index])
x,y = translateDNA(pop)
print("最优的基因型:", pop[max_fitness_index])
print("(x, y):", (x[max_fitness_index], y[max_fitness_index]))
if __name__ == "__main__":
fig = plt.figure()
ax = Axes3D(fig)
plt.ion()#将画图模式改为交互模式,程序遇到plt.show不会暂停,而是继续执行
plot_3d(ax)
pop = np.random.randint(2, size=(POP_SIZE, DNA_SIZE*2)) #matrix (POP_SIZE, DNA_SIZE)
for _ in range(N_GENERATIONS):#迭代N代
x,y = translateDNA(pop)
if 'sca' in locals():
sca.remove()
sca = ax.scatter(x, y, F(x,y), c='black', marker='o')
plt.show()
plt.pause(0.1)
pop = np.array(crossover_and_mutation(pop, CROSSOVER_RATE))
#F_values = F(translateDNA(pop)[0], translateDNA(pop)[1])#x, y --> Z matrix
fitness = get_fitness(pop)
pop = select(pop, fitness) #选择生成新的种群
print_info(pop)
plt.ioff()
plot_3d(ax)
max_fitness: 0.10333042920383484
最优的基因型: [1 1 1 0 0 0 0 0 0 0 0 0 1 1 1 0 0 0 0 0 0 0 0 0 1 1 0 1 1 1 0 0 1 0 1 1 0
1 1 1 0 1 1 0 1 0 0 1]
(x, y): (0.04820019294024647, 1.571304832178642)
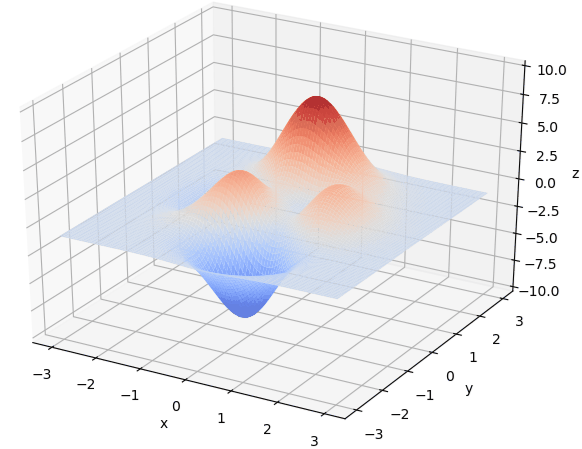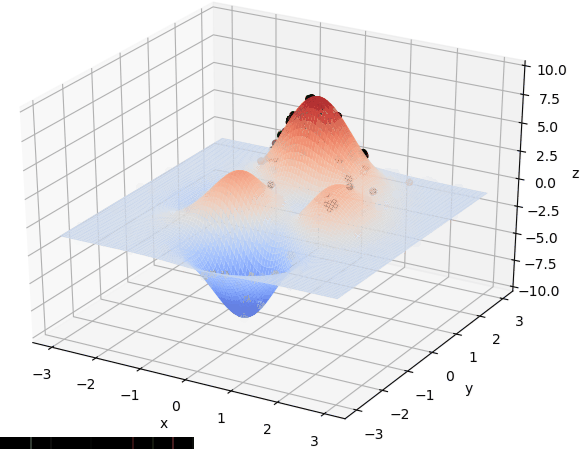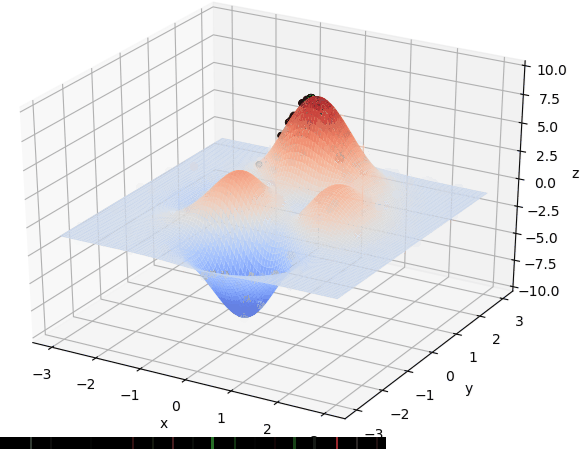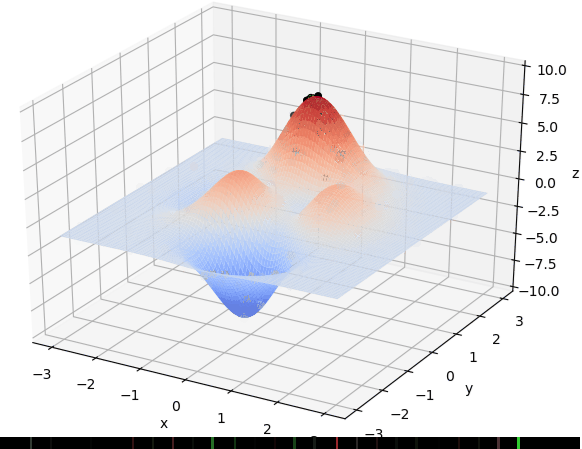
图源:遗传算法详解 附python代码实现
原文:【算法】超详细的遗传算法(Genetic Algorithm)解析
遗传算法详解 附python代码实现