论文解读:《功能基因组学transformer模型的可解释性》
·@[TOC](论文解读:《Explainability in transformer models for functional genomics》)
文章地址:https://academic.oup.com/bib/advance-article/doi/10.1093/bib/bbab060/6214646?login=true
DOI:https://doi.org/10.1093/bib/bbab060
期刊:Briefings in Bioinformatics(2区)
影响因子:8.99
发布时间:2021年4月8日
代码:https://github.com/jdcla/DNA-transformer
测试集的注释和模型输出:https://kermit.ugent.be/files/UCSC/UCSC_browser.html
1.文章概括
深度学习方法的有效性很大程度上归功于从原始数据中自动提取相关特征。在功能基因组学领域,这通常涉及从DNA序列中自动选择相关的核苷酸基序。为了从自动化学习方法中获益,需要新的策略来揭示训练模型的决策过程。在这篇论文中,作者提出了一种新的方法,它已经成功地收集了关于大肠杆菌转录过程的见解。这项工作建立在一个基于transformer 的神经网络框架设计的原核基因组注释的目的。作者发现,该模型的大多数亚基(注意头)专门用于识别转录因子,并能够成功地表征它们的结合位点和一致序列,揭示了参与转录过程启动的已知和潜在新元素。随着注意力头的专业化自动出现,transformer 模型对在这一领域创建可解释的神经网络具有很高的兴趣。
2.背景
深度学习技术在基因组学的众多预测任务中越来越多地获得最先进的性能。这些技术能够在训练期间自动化特征提取过程,与机器学习研究的传统方法相反;不需要有意义的描述符作为模型的输入,而是使用原始输入数据。因此,识别由模型学习的具有生物意义的描述符及其与目标标签的关系需要一组新的方法。
深度学习为目前对生物过程理解不足的功能基因组学提供了解决方案。因此,可解释的模型可以提供对模型所针对的生物过程的洞察力,受过培训的人可能会在发现新的监管机制方面发挥作用。
对解剖神经网络工作的主要贡献是通过将输入特征的重要性映射到模型输出来实现的,或者是通过研究输入的梯度,或者是通过研究输入的排列策略。这些技术仅限于对模型输出上的输入特征进行评估,因此在试图捕捉生物过程中涉及的多个调节元件之间的复杂机制时可能会受到限制。
在先前的研究中,作者介绍了一种在原核生物基因组序列上执行注释任务的模型,在识别转录起始点(Tsss)、翻译起始点、翻译起始点和甲基化位点方面取得了最先进的结果。该模型基于Transform-XL架构,首次引入该架构用于自然语言处理。Vaswani等人研究发现,作为神经网络核心机制的专门单位–注意力头部,可以映射出传达语义的关系。基于这一发现,作者的目标是研究从接受过基因组注释训练的变压器网络中提取生物学意义的新方法。
这项工作提供了几个贡献:(i)作者评估和比较了各种最新的注释,主要是基于测序的,在活体方法学中用于检测大肠杆菌中的TSS。基于四个独立的数据集,挑选了一个改进的注释集;(ii)提出了作者的方法来表征注意头部的功能,其中许多是专门用于根据序列和位置信息检测调控元件的存在;(iii)利用这些信息,作者提供了与现有的关于转录过程中涉及的机制的知识的比较,将现有的发现与文献联系起来,并讨论它们的相关性;(iv)使用全基因组序列上的模型输出预测,可以更好地理解模型的特点和潜在的缺陷。基于这些结果,作者得出结论:对所讨论的基因组注释任务模型的分析为提取生物学意义提供了独特的潜力,其中所讨论的技术适用于任何基因组注释任务。
3.相关工作
促进核苷酸序列的解释和作用的方法学的创建始于共识序列、权重矩阵和n阶比对的数学公式。人们对分子生物学的理解日益加深,产生了核苷酸序列的有意义的描述符,如GC含量、可弯曲性、灵活性和自由能。最近出版的Seq2Feature(计算了 252 种蛋白质和 41 种基于 DNA 序列的描述符。这些特征包括蛋白质的物理化学、能量和构象特性、突变矩阵和接触潜力以及核苷酸成分、DNA的物理化学和构象特性。)使用41个描述符来表征核苷酸序列,这表明有意义的序列描述符在今天仍然有意义。
尽管努力创建核苷酸序列的有意义的描述符,但对这些如何影响生物过程的理解仍然缺乏。即使在早期机器学习方法的帮助下,DNA在许多生物过程中的作用也没有得到正确的理解。今天,深度学习的分支越来越多地为功能基因组学领域的各种任务返回最先进的表演。通过在训练过程中集成特征提取过程,模型能够从原始DNA序列自动映射关系,而不需要高级描述符。然而,与早期的应用不同的是,为了理解核苷酸序列和生物过程之间的关系,需要研究训练后的网络的决策过程。
目前,有两种流行的策略旨在通过评估输入特征对输出预测的影响来深入了解神经网络的工作原理。第一组通过对输入特征的定向排列来实现这一点,Zeiler等人首先描述了这一点,还描述了许多其他策略,包括Fisher等人的工作和Zintgraf等人的工作。另一组方法试图调查模型输出类相对于输入特征的偏导数。该技术最早由Simonyan等人在2014年讨论,此后被用于构建各种工具。在结合了基因组学和深度学习的领域,这些想法已经被借用来绘制重要的位点和核苷酸基序。例如,Alipanahi等人进行敏感性分析以指导DNA上蛋白质结合位点模体的构建。Angermueller等人评估了核苷酸对甲基化状态的敏感性。Hill等人每静音输入序列,以调查对翻译起始和终止位点的影响。通过应用成对变异图,显示了两个位置特征之间的相关性。另一项研究利用部分衍生物的分析来构建真核生物剪接位点周围的基序。
4.方法
4.1 数据收集
为了减少噪声的潜在影响,比较了最近几种高精度的活体实验方法对原核生物中tsss的检测和注释:Etwiller等人的Cappable-Seq方法检索16348个TSS,SMRT-Cappableseq(SMRT-Cappableseq),严氏等人检索2311个TSS和同步5-端和3-端测序(SEND-SEQ),Ju等人检索4026个TSS。
考虑到它对社区的重要性,RegulonDB列出的TSS也被包括在内。RegulonDB提供了最新的手工管理和自动(差异RNA-seq)TSS的集合,这些TSS来自很多独立来源,它已经成为最近几个机器学习任务的选定数据集,这些任务旨在识别TSS。正数集由标有“有力证据”的仓位组成,总计6487个注解仓位。表一列出了有关数据的概览。

4.2 模型体系结构
该模型是从Transformer-XL架构构建的。一般而言,Transformer模型正越来越多地取代递归神经网络,因为这些体系结构已被证明更适合于序列数据的优化,从而改善了训练时间和性能。作者的模型还包括卷积层,通常用于检测局部模式(补充表A2),从而增强了核苷酸基序的检测。应用模型已在以前的工作中提出,其中包括对应用于基因组注释任务的现有深度学习方法的深入比较。

注意力头部为计算每个隐藏状态h的注意力z而执行的数学运算的概述。对于l个隐藏状态(H→Z)并行执行计算注意力的运算。通过H与Wq、Wk和Wv的矩阵相乘得到q、k和v向量,使用与dheads一样多核的单个卷积层导致每个输入的单个q、k和v矢量表示的变换,所述各个输入将从dconv相邻核苷酸的q、k和v矢量导出,然后计算注意力Z。该模式保持简单以便更好地理解,并且不包括添加到Q和K矩阵的相对位置编码,也不包括递归机制。
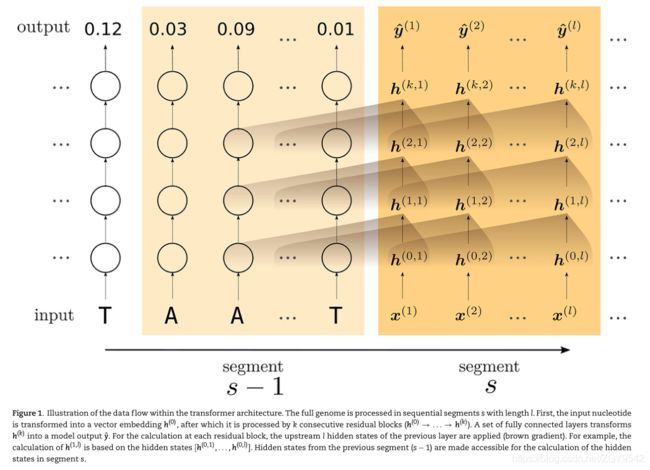
在长度为l的连续片段中处理全基因组序列。每个输入核苷酸x∈{A,C,G,T}首先被变换成嵌入h(0)的载体,通过与另一载体相加(剩余连接)变换k次,所述另一载体由每层中存在的多头注意函数(h(0)→…→h(K))获得。一组完全连接的层将h(K)转换为模型输出y(K)。对于每个残差块,使用L个上游位置的隐藏状态来计算与输入相加的向量(以获得h(1),…,h(K))。图1说明了网络内中间值之间的关系,如果需要可以访问来自前一段(s−1)的隐藏状态,以便计算段s中的新隐藏状态。
在每个残差区块中应用的多头注意在方法上是相同的。根据每个输入隐藏状态h,计算相等形状的查询query(q)、键key(k)和值value(v)向量。注意头的输出z,应用于位置n处的隐藏状态计算如下:

其中K,V∈RL×d heads是由L个上游隐藏状态组成的矩阵(例如K=[k(n−L),…,k(n)])。分母用于基于q、k和v(Dhead)的维度来稳定分数。查询向量与所有键向量相乘得到一个分数向量,该向量使用Softmax函数对所有输入值进行归一化。这些分数乘以v向量以计算z(即线性组合)。本质上,注意力分数表示存在于两个位置之间的信息的相关性,其中q和k向量的乘法起锁定和密钥编码的作用,其返回嵌入在两个隐藏状态(由v定义)中的信息的拟合优度分数。在每个剩余块中,存在多个注意力头部(因此,多头部注意力),每个注意力头部具有其自己的独特的模型权重集合,以计算Q、k和V。因此,可以从输入的隐藏状态中提取多种类型的信息。同一层内的不同注意力头部的结果被进一步处理成单个向量,该向量与h相加以获得下一层的隐藏状态(例如h(1)→h(2))。
在先前的研究中,作者发现嵌入在由单核苷酸产生的隐藏状态中的上下文信息是有限的。由多个相邻核苷酸形成的基序通常被认为对生物过程更重要。卷积层的添加允许从多个相邻的隐藏状态导出Q、K和V矢量,而不影响输入/输出分辨率。因此,改进了使用注意力检索相关信息,从而提高了对各种任务的预测性能。
在矢量q、k和v内需要位置信息,因为它们不是通过网络的架构隐含的。这是通过将位置编码矢量叠加(即,通过求和)到h来实现的。添加的信号是矢量索引和相对于其他输入隐藏状态的相对定位的函数。结果表明,Transformer模型能够很好地提取这些信息,并利用这些信息来获得更好的性能。
在先前的研究中,模型架构针对TSS的检测进行了优化,得到的模型片段长度为512个核苷酸,6层(即残留块),每个残留块内有6个注意头。为了包括带注释的TSS的下游朝向其注释的信息,标签可以在处理时间期间向下游移动,以便包括该信息(即位于上游)。根据最近关于检测TSS的文献,下游结合位于TSS下游的20个核苷酸。补充文件1中给出了模型体系结构的详细概述。

4.3 训练和评估
基于Transformer的模型使用全基因组序列进行训练,总样本量为9283204。正样本和负样本的大小如表1所示。该模型依次迭代基因组,通过在分别占基因组70%、20%和10%的三个位置分割基因组来创建训练、测试和验证集。训练、测试和验证集合具有相等的输入和输出类别分布。为了比较同一基因组上的所有数据集,最初定位在‘U00096.2’参考基因组上的Cappableseq实验的注释被重新映射到‘NC_000913.3’基因组上。相同的基因组区域被用来训练和评估所有的模型,位于位置2738785、3667115和4131280(根据先前的研究),由‘RefSeq’基因组索引(增加:‘NC_000913.3’)。因此,这些链的正义和反义成分都包括在相同的集合中,以保证不同集合之间没有不可预见的信息传输。验证集上的最小损失用于确定停止网络训练的点。假设预测任务是一个二进制分类问题,则使用交叉熵损失。通过对测试集的评估,得到了模型的性能度量。
4.4 模型分析
对于位置n处的模型输出的计算,每个注意力头部基于n的隐藏状态和该层中的511(L)上游隐藏状态来计算注意力分数。具体地说,如之前所述,这是通过将q和k矢量相乘并使用Softmax函数进行连续归一化来实现的。对于一个有36个注意力头部和1856660个核苷酸位置(样本)的测试集,总共计算了大约3.4·1013个注意力分数。为了减少处理的值的数量,只处理少量样本的上游隐藏状态的分数。具体地说,除了测试集约1%的随机抽样位置(18295个样本)外,还包括根据核苷酸位置对注意力得分进行分组,这会导致最高和最低的模型输出概率(各500),从而可以查看某些注意力头部对模型输出的影响以及平均注意力得分曲线之间的差异。
5.结果
5.1 评估的批注集有许多不同的批注起点
为了优化大肠杆菌中的TSS注释集,处理了各种数据集。严等人考虑在5个核苷酸的距离内的两组注释,如果他们定位在五个核苷酸的距离共享。根据该标准,图2A中给出了四个数据集之间共享TSS的分布。所有四个数据集之间仅共享1012个位置。由至少三个和两个数据集共享的TSS总数分别为2224和5104。在RegulonDB(38.17%)和cappable-seq(28.33%)中,任何其他数据集列出的TSS所占比例最小。SMRT-Cappableseq(89.78%)和Send-seq(73.17%)在共享TSS中所占比例最高(表1)。然而,由于数据集的大小不同,应谨慎考虑百分比。例如,使用16348个注释,cappable-seq具有的TSS数量是任何其他数据集的两倍多。图2B图2A中使用的五个核苷酸窗口内的TSS之间距离的分布。


5.2 改进的模型性能表明精选定制集的质量更好
为了提供对注释质量和创建精选集合所遵循的各种步骤的验证手段,对不同模型在各种集合上的性能进行了训练和评估。验证和测试集跨越基因组的相同区域。因此,对于不同的标注,可以容易地评估训练模型在测试集上的性能。表2给出了所有模型在每个测试集上的性能的完整概述。

5.3 大多数注意力集中的人对某些启动子区域有很高的选择性
该模型使用注意力机制从大量数据中选择性地收集信息。为了确定在给定位置是否存在TSS,每个注意力头部为512个上游位置分配分数,确定由这些隐藏状态表示的信息的重要性。每个位置的平均和最高分数由下图表明,大多数注意力集中的头部都是高度选择性的w.r.t 使用信息的位置。换句话说,大多数注意力集中在特定的启动子区域。

为每个注意头的每个上游位置计算的平均注意力分数。注意头按层分组(第一个索引)。每个位置的注意分数是计算位于位置0的核苷酸的模型输出所需的中间值。已经分离了三组:完整测试集的子样本(随机),占18295个样本,500个模型输出的最高和最低。
针对每个注意头的每个上游位置计算的最高注意力分数。注意头按层分组(第一个索引)。每个位置的注意分数是计算位于位置0的核苷酸的模型输出所需的中间值。已经分离了三组:完整测试集的子样本(随机),占18295个样本,500个模型输出的最高和最低。
5.4 注意力集中在检测转录因子结合位点上
对于每个注意力头部,基于平均得分最高位置的50个得分最高的样本创建序列主题。这些序列模体除了反映位置信息外,还反映了导致高注意力分数的序列特征。

次采样测试集(18295个样本)的平均注意力分数的一种可视化。每个位置的注意分数是计算位于位置0的核苷酸的模型输出所需的中间值。颜色图将低值表示为深蓝色,较高值表示为黄色,并为每个注意头归一化为(AH)的0到1。
次采样测试集(18,295个样本)的平均注意力分数的一种可视化方法,以最后120个隐藏状态为中心。每个位置的注意分数是计算位于位置0的核苷酸的模型输出所需的中间值。颜色图将低值表示为深蓝色,较高值表示为黄色,并标准化为每个注意头的0到1(AH)。
从经过训练的变压器网络中提取的信息的示例。(I)用于计算模型体系结构内的中间隐藏状态h的注意力头部(正方形)的图示。每一层都有六个注意力头部。绿色方块显示了这里描述的注意力头部的位置。(Ii)对于所讨论的6个注意头部,显示了512个上游隐藏状态中的每一个的注意分数,平均为基因组上的18 295个随机位置(占测试集的1%)。根据位置信息,几个注意力集中的头部具有很高的针对性,黄色显示高分,蓝显示低分。对每个注意力头部的分数进行归一化处理。(Iii)对于每个注意力头部,根据平均得分最高的上游位置的50个最高得分的核苷酸序列制作序列基序,说明每个注意力头部返回高分数的序列信息。(Iv)基于来自(Ii)和(Iii)的序列和位置信息,存在将注意力焦点头部与参与转录过程的已知转录因子的工作方式连接起来的匹配。
5.5 注意头对模型的影响可以映射
对于每个注意力头部,显示分配给每个(上游)位置的注意力分数和模型输出之间的Spearman等级相关性。每个相关系数都乘以该位置的平均权重。注意头按层(第一个索引)分组。

5.6 模型的输出概率反映了正义和反义之间的相关性
增加的模型输出通常在这两方面都是一致的向前和向后的股线。图4A所示为TSS周围每个位置的值。这显示了增加了正反义的输出概率股,其中后者仅表现出整体增长活动,但也高峰上游的TSS。分析概率分布揭示了总体上增加的输出概率对于任何一条链上都没有编码序列的区域。整个测试区域的平均输出概率设为0.0052。所有核苷酸的中位概率任何一条链上存在编码序列的位置都是0.0049,与整个测试集的中值一致。核苷酸的中值是原来位于编码序列之间的两倍多(0.0105)。反义的可能性更高,不仅存在于边界和向外的编码序列中,而且因为两条链上的较高模型输出普遍存在于操作子内的边界编码序列中。使用RegulonDB上的操纵子映射,操纵子非编码序列中正义的概率中值为0.0079,反义的概率中值为0.0071。这些短序列有大量的峰,其中每个区域最大值的中位数大约是基线的10倍,正义和反义的中位数分别为0.0415和0.0480。

概述了描述模型预测特性的几个曲线图。数据是通过对精选注释的测试进行训练和评估的模型来获得的。测试集(0.053)上的中值模型输出被表示为(a,b,c)中的水平黑线。每个曲线图给出了以感兴趣部位为中心的1201个核苷酸窗口内每个位置的中位模型输出。显示的是所有(A)转录起始位点(TSS),(B)标记为假基因的基因的TSS和©所有转录终止位点周围位置的中值模型输出,从Ju等人获得。
下图给出了假基因区的示例,显示了UCSC浏览器(补充数据)上显示的经过管理的TSS注释的模型输出。该图显示了模型输出的正义和反义之间紧密交互的示例。还显示了smrt-cappable-seq实验的映射读数。

6.讨论
功能基因组学和深度学习领域的进步为理解生物过程提供了新的机会。目前,有几种方法可以分析输入特征与模型输出的敏感度,并且适用于不同的模型体系结构。然而,当驱动注释任务的生物过程涉及复杂机制时,现有方法可能是有限的。例如,这些可能涉及重叠的结合区。为了对经过训练的模型的决策过程有更多层次的理解,可以创建绑定到模型体系结构本身的方法。因此,要洞察深度学习模型,可能需要模型设计和寻求解释其功能的专门技术的协调努力。
在功能基因组学的情况下,机器学习模型在基因组注释任务上的训练通常会带来几个挑战:(i)输出类别之间的不平衡,负集,它比正集大几个数量级;(ii)缺乏高质量的注释,其中存在大量的噪声;(iii)输入特征空间的维度,数百万长度的核苷酸序列;(iv)注释景观的生物变异,这除其他因素外,通常取决于(亚)物种。
关键点:
- 作者专注于探索变压器网络可以为基因组注释任务提供可解释的机器学习的优势。
- 对于TSS的检测,作者能够表征模型中注意头部的功能,并发现它们专门用于检测转录因子结合位点,不需要任何人工输入,就可以精确定位几个重要的启动子元件的位置、它们的最佳序列基序以及它们的重要性。
- 虽然作者专门为TSS提供了这种分析,但概述的方法是通用的,适用于任何基因组注释任务。


