- OmicsTools除b站教学视频外已整理的零代码生信全流程分析文档
邢博士谈科教
医学科研生信分析r语言数据可视化数据挖掘数据分析生信医学生信分析
OmicsTools软件介绍和下载安装配置软件简介我开发了一款本地电脑无限使用的零代码生信数据分析作软图神器电脑软件OmicsTools,欢迎大家使用OmicsTools进行生物医学科研数据分析和作图,该软件件能让大家在不需要任何编程和代码编写的基础上,分析次数没有限制,可以无限使用,让您在自己电脑上快速进行大量的生信分析和加速大家的科研。OmicsTools生信分析电脑软件可以做医学生物生信各个
- 学生管理系统
wu1113_
java
文章目录1.案例需求2.编程思路3.运行效果4.案例源码5.小结1.案例需求上次我们完成了一个酒店管理系统,这次我们使用面向对象思维,完成一个学生管理系统。实现一个简单的学生管理系统,它具备5个功能,分别为显示所有的学生信息添加学生修改学生信息删除学生退出本系统功能2.编程思路首先我们要额外定义一个Studnet类,包括学生的学号、姓名、年龄、性别等属性。其次我们定义一个测试类Test,在测试类的
- 100个基本,明确自己的目标
石小沫_
最近在读日本作家松浦弥太郎写的《100个基本》这本书,其中作者解释“100个基本”是为了让自己明白的事,是对自己展开的思考,是为了整理自己的思路而存在的。这是为了个人成长、学习,为了更像自己,而应该恪守的人生信条。重要的事情,想要遵守的规则,我想每个人都有,但这些内容其实是很模糊的,难以用言语表述。如果能一条条地认真思考如何用言语表达它们,也能借此更好地认识自身。“基本”可以关涉任何事――计划、目
- EasyExcel实现复杂Excel的导入
小卡车555
java处理excelexcel
最近项目中遇到一个复杂的Excel的导入,并且数据量较大。因为数据不规则,所以只能使用POI进行自定义读取,但是发现数据量大之后,读取数据非常耗时。后面换成EasyExcel,性能起飞。1.Excel样板如上图,需要导入学校学生信息。前面三列,固定为学校班级信息,可以理解为主表。从第四列开始,为学生信息,其中一列为一条子表信息。2.具体实现引入EasyExcel的依赖com.alibabaeasy
- 读书笔记
归根曰静
无信仰的结局都是一样的,都是空虚与绝望。雅斯贝尔斯再进一步来看,他说:“真正使人产生信仰的,既不是宇宙万物,也不是人生的特别遭遇,而是人的自由。一个人意识到自己是自由的,就等于确信了上帝的存在。”理由是:作为自由的存在者,当我真正成为我自己的时候,我知道我并不是靠我自己而成为自由的。图片发自App只要谈到自由,就有一个观念,就是能够自制、能够自我约束的才能够享有自由,因为它跟命令不能分开。但有的时
- 高情商沟通(9)迅速破冰建立信任,持续获得好感
叁叁二二叁磊设计
高情商,两人相遇无话可说,实质是彼此没有安全感,而让对方有安全感的基本是信任感,破冰就是要让对方产生信任感,建立信任的目的:在沟通中,建立彼此之间的信任感,其最重要的目的,是建立起良好的人脉关系,建立信任的需求:没有安全感,就不会链接到其他伙伴,也不会有彼此之间的资源互换,建立信任的核心:破冰沟通不成功的核心原因是彼此没有找到共同认可的观点和话题建立信任的结果:信任产生人脉,人脉产生资源沟通破冰模
- javaweb基于ssm框架学生信息管理(成绩)系统设计与实现
ancen_73bd
开发平台、开发工具、应用服务器的介绍开发平台:Windows开发工具:idea+mySql应用服务器:ApacheTomcat8.0学生成绩管理系统主要用于学校学生成绩信息管理,能实现学生、老师、院系、班级、课程的增删改查操作,同时学生能进行选课和退课操作,老师能对学生的成绩录入和修改操作。系统流程图功能结构图部分截图免费源码获得:扫码关注微信公众号:ancenok,然后回复:013
- 纯生信很难发表?只是你没有及时抓住研究热点
SCI狂人团队
当你还做meta分析的时候,你会发现meta分析很难发或者单位已经不承认了,而聪明的人已经开始做常规的生信GEO、TCGA数据挖掘这些(这个时候生信比较好发)。当你开始做常规的生信GEO、TCGA数据挖掘的时候,你会发现这些一样也是比较难发了,而聪明的人已经开始抓免疫评分这个热点进行生信数据挖掘(这个时候免疫评分比较好发)。当你开始对免疫评分这个热点进行生信数据挖掘的时候,你会发现自己的研究方向差
- 本周回顾2021-02-28
曾有卓
目标生信、交易系统、健身、写作、音乐本周完成节食坚持了11天,周末最终还是中断了,明天继续。工作较忙,加上沟通问题,对于生信有些动摇。音乐上还是要加强练习。反思管住嘴,坚持住。克服情感上的小波动对心态的冲击,对于父母、夫妻、兄弟姐妹之间的关系,永远保持赤诚之心,这是最后的依靠。投资上也是一样的道理,分清自己做的到底是理财、还是投资还是交易。下周计划做好日常工作。不管是否投稿,都要完成科普文章。践行
- 本周回顾2021-10-31
曾有卓
目标生信、交易系统、子女教育、健身、写作、音乐本周完成健身一次,节食3天,效果不好,下周继续健身其实孩子的老师原来愿意SL,有喜有忧吧孩子玩乐高非常投入,可以说忘我,这还真是天生的。近距离参与了一次WQ,能成功非常之复杂,多方面的因素都要考虑,像一场以少胜多的战役,方方面面的,个人虽然有一些想法,奈何表达能力不足,不能挺身而出。反思抓紧做好手头工作,别把事情拖到最后。下周计划把现实工作做好。
- Java 知识框架主要包括面向对象、集合框架、异常处理、泛型和多线程等知识
爱摄影的程序猿
JAVA面试java开发工具windowsjavaspringboot
Java知识框架主要包括面向对象、集合框架、异常处理、泛型和多线程等知识。下面将对每个知识点进行详解,并提供相关的案例和面试题解答。面向对象:面向对象是Java的核心思想,它将程序中的数据和操作封装成对象,并通过对象之间的交互实现程序的功能。在面向对象的思想中,包括类、对象、继承、多态等概念。案例:创建一个学生类,包含学生的姓名和年龄属性,以及获取学生信息和修改学生信息的方法。publicclas
- 2019-06-19暑期补习,你怎么看?
M曼君
2019/6/195/17周三多云眼下,已经开始有一些教育培训机构在已经在争先恐后的开始发放起来了,琳琅满目的招生信息一下子席卷而来。话说,这离放假还早着呢,你们就这么着急开始收学生了吗?好不容易放个暑假吧,又得安排孩子学这学那的,要是不学吧,很多家长都说了,别的孩子在暑假的时候补课了,你不给孩子补课,到时候补课的时候孩子跟不上呀,孩子着急呀,孩子压力会更大呀,怎么办?哎呀,要我说这种中国的这种教
- sql 外键约束
Tanjarh
MySQL笔记sql
创建有外键约束的数据库表首先创建父表这里用的学生信心录入为例子--创建父表:班级表createtablet_class(cnoint(4)primarykeyauto_increment,cnamevarchar(10)notnull,roomchar(4))再创建子表createtablet_student(snoint(6)primarykeyauto_increment,snamevarch
- 生信人应该这样来装软件(未完待续)
dandanwu90
P1优先选择二进制可执行软件来安装不同组学任务模块比对重要全基因组测序/外显子测序找差异和变异转录组相关计数软件安装规律:二进制,下载解压,全路径调用#C语言查看在还是不在whichmakewhichcmakewhichjavawhichperlwhichpython#查看版本python--versionperl--version查看版本,版本问题导致错误如何判断所有软件是否是二进制,进入软件官
- 生信学习Day-1
GJJDr
1.如何学习2.怎样解决学习中遇到的问题?a.第一步:搜索:首选-谷歌,其次-必应,大神级的搜索引擎:虫部落快搜。专业教程-搜狗微信、搜狗知乎、、githubb.第二步:如果你的问题不知该如何搜索,可在微信群中与小组成员讨论c.第三步:正确的提问3.如何搭建高效的学习平台a.效率软件:(1)浏览器-chrome浏览器简洁高效无广告,可以添加插件,比如”沙拉查词”(自行搜索),可以即时翻译。(2)电
- 7+纯生信,单细胞识别细胞marker+100种机器学习组合建模,机器学习组合建模取代单独lasso回归势在必行!
生信小课堂
影响因子:7.3研究概述:皮肤黑色素瘤(SKCM)是所有皮肤恶性肿瘤中最具侵袭性的类型。本研究从GEO数据库下载单细胞RNA测序(scRNA-seq)数据集,根据原始研究中定义的细胞标记重新注释各种免疫细胞,以确定其特异性标志。接着通过计算免疫细胞通信网络,结合对通信网络的大量分析和通信模式的识别,对所有网络进行了定量表征和比较。最后基于bulkRNA测序数据,使用机器学习训练了枢纽通讯细胞的特定
- 在TCGA上下载数据并且进行处理
Red Red
生信小技巧r语言数据库
浏览器搜索TCGAGDC进入网站在TCGA数据库主页选择“Repository”模式根据所需要的选项在侧边栏选择数据清空购物车!!第一次登陆可忽略将刚刚选择好的数据加入购物车,并且在购物车里下载Metadata和Cart数据,下载到同一个文件夹下。使用R语言脚本对数据进行处理,将其提取为genesymbol和样本的数据,推荐看一下该博主处理数据!!真的非常详细!他R语言脚本在这个链接里
- 学习小组Day3笔记--安之若素
安之若素_9e53
Conda的安装与初步使用Conda是Linux环境下的软件安装与管理工具相当于应用商店,日常生信使用miniconda即可miniconda的下载与安装从清华镜像下载miniconda3安装包wgethttps://mirrors.tuna.tsinghua.edu.cn/anaconda/miniconda/Miniconda3-latest-Linux-x86_64.sh安装minicond
- tkinter+sqlite3学生信息管理系统_python图形界面数据库tkinter+sqlite3
2301_82244392
程序员python学习面试
网上学习资料一大堆,但如果学到的知识不成体系,遇到问题时只是浅尝辄止,不再深入研究,那么很难做到真正的技术提升。需要这份系统化学习资料的朋友,可以戳这里获取一个人可以走的很快,但一群人才能走的更远!不论你是正从事IT行业的老鸟或是对IT行业感兴趣的新人,都欢迎加入我们的的圈子(技术交流、学习资源、职场吐槽、大厂内推、面试辅导),让我们一起学习成长!用到了sqlite数据库,std_data.py文
- 0基础跟德姆(dom)一起学AI Python进阶03-面向对象版: 学生管理系统
跟德姆(dom)一起学AI
python开发语言人工智能
*面向对象版:学生管理系统*Student学生类*StudentCMS学生管理类*main.py程序的主入口*加入文件:student.txt存储学生信息*深拷贝和浅拷贝*可变类型*不可变类型---1.学生管理系统-分析*涉及到的知识点*面向对象*列表,字典*for,if,input...*文件操作*......*需求分析*结论:分模块(.py文件)进行开发***main.py**程序的主入口**
- 第十四节:学习Springboot 的restful接口风格(自学Spring boot 3.x的第三天)
Mero技术博客
从零开始学习SpringBoot3.x学习springbootrestful
这节记录下自己学习restful的记录。增(PostMapping)/***保存学生*@return*/@PostMappingpublicStudentsave(@RequestBodyStudentstudent){studentService.save(student);returnstudent;}注意:传参使用RequestBody删(DeleteMapping)/***删除学生信息*@
- seurat提取表达矩阵_10X scRNA免疫治疗学习笔记-3-走Seurat标准流程
幸福的小酒瓶
seurat提取表达矩阵
刘小泽写于19.10.15笔记目的:根据生信技能树的单细胞转录组课程探索10XGenomics技术相关的分析课程链接在:http://jm.grazy.cn/index/mulitcourse/detail.html?cid=55第二单元第7讲:走Seurat标准流程【文章结构总-分-总,结尾有完整的代码,熟悉者前面可以跳过,去看后面8min完成的代码】前言前面介绍了自己利用cellrangerc
- 自己公司业务逻辑,仅做个记录备份方便查阅
风中凌乱的男子
后端数据表结构学生表:ID、学生名字、家长名字、购买课时、购买金额、date老师表:ID、老师名字、老师手机号(唯一的)、学生ID、date余额表:ID、剩余课时、剩余金额、学生ID、date消耗明细表:ID、学生名字、家长名字、单次消耗课时、单次消耗金额、当前剩余课时、当前剩余金额、上课老师后台管理逻辑思维:超级管理员登录后台,首先输入学生信息(学生名字家长名字购买课时购买金额)。点击提交,跳转
- 使用集合操作思路,查询选修了CS305,但没有选修CS315的所有学生信息
naughty610
sql
--23.2使用集合操作思路,查询选修了CS305,但没有选修CS315的所有学生信息。selectstudidfromtranscriptt1wherecrscode='cs305'andnotexists--查询条件为选修了cs305而且要求子查询返回的结果必须是空(即:没有选修cs315)(select*fromtranscriptt2wheret1.studid=t2.studidandt
- Linux编程--信号基础
玮玮豆豆
linuxlinux
一、概念和概述1.信号是事件发生时对进程的通知机制。有时也称为软件中断2.引发内核为进程产生信号的各类事件如下:硬件发生异常,即硬件检测到一个错误条件并通知内核,随即再由内核发送相应信号给相关进程。硬件异常的例子包括执行一条异常的机器语言指令,诸如,被0除,或者引用了无法访问的内存区域。用户键入了能够产生信号的终端特殊字符。其中包括中断字符(通常是Control-C)、暂停字符(通常是Contro
- mysql 复杂 语句_Mysql复杂查询语句汇总(仅供学习)
铁扇公子
mysql复杂语句
1、mysql查询今天、昨天、上周今天select*from表名whereto_days(时间字段名)=to_days(now());昨天SELECT*FROM表名WHERETO_DAYS(NOW())-TO_DAYS(时间字段名)34查询学员朱欣磊选修的课程信息SELECTs.*FROMsJOINscONs.id=sc.sidWHEREs.sn='朱欣磊'5查询没有选择数学课的学生信息SELEC
- 零基础入门生信数据分析——导读
呆猪儿
生信之转录组——上游分析生信之转录组——下游分析学习方法r语言数据分析数据库数据挖掘需求分析大数据
零基础入门生信数据分析——导读生信数据分析,即生物信息学数据分析,是一个涵盖了生物学、计算机科学、数学和统计学等多个领域的交叉学科。它主要利用计算机算法和统计方法对生物学数据进行处理、分析和解释,以揭示生物分子、细胞、组织和生物体等各个层次的生物学规律和机制。本帖主要是为生信数据分析的各个分析点提供跳转链接(简单说就是提供了一个目录供大家选择自己想要的知识点可以直接跳转)关联的生信数据分析的分析点
- Python程序设计之 —— 简易学生信息管理系统_用python做信息管理系统
2401_84556837
程序员python开发语言
前言Python简介Python是一个高层次的结合了解释性、编译性、互动性和面向对象的脚本语言。Python的设计具有很强的可读性,相比其他语言经常使用英文关键字,其他语言的一些标点符号,它具有比其他语言更有特色语法结构。Python是一种解释型语言:这意味着开发过程中没有了编译这个环节。类似于PHP和Perl语言。Python是交互式语言:这意味着,您可以在一个Python提示符>>>后直接执行
- 铁死亡、细胞焦亡都发了一篇纯生信,就差这个热点还没有发
SCI狂人团队
有粉丝说自己发了一篇铁死亡的纯生信,细胞焦亡的纯生信也发了一篇,现在还想发一篇纯生信。好想找一个少人做的热点来继续搞一篇纯生信,但是不知道有没有这样的热点?肯定是有的,例如你可以搞一下坏死性凋亡,这个热点现在很少人做。“有人问为什么要找少人做的热点来做纯生信,我之前写了一篇铁死亡的纯生信,同一种癌症的同一种套路的文章已经发了三篇,一样将文章发表在非预警期刊上。“那你发的是什么期刊呢?答:Bioen
- c语言结构体存储图形示例,C语言 结构体(Struct)详解及示例代码
龙猫美术的世界
c语言结构体存储图形示例
前面的教程中我们讲解了数组(Array),它是一组具有相同类型的数据的集合。但在实际的编程过程中,我们往往还需要一组类型不同的数据,例如对于学生信息登记表,姓名为字符串,学号为整数,年龄为整数,所在的学习小组为字符,成绩为小数,因为数据类型不同,显然不能用一个数组来存放。在C语言中,可以使用结构体(Struct)来存放一组不同类型的数据。结构体的定义形式为:struct结构体名{结构体所包含的变量
- JAVA中的Enum
周凡杨
javaenum枚举
Enum是计算机编程语言中的一种数据类型---枚举类型。 在实际问题中,有些变量的取值被限定在一个有限的范围内。 例如,一个星期内只有七天 我们通常这样实现上面的定义:
public String monday;
public String tuesday;
public String wensday;
public String thursday
- 赶集网mysql开发36条军规
Bill_chen
mysql业务架构设计mysql调优mysql性能优化
(一)核心军规 (1)不在数据库做运算 cpu计算务必移至业务层; (2)控制单表数据量 int型不超过1000w,含char则不超过500w; 合理分表; 限制单库表数量在300以内; (3)控制列数量 字段少而精,字段数建议在20以内
- Shell test命令
daizj
shell字符串test数字文件比较
Shell test命令
Shell中的 test 命令用于检查某个条件是否成立,它可以进行数值、字符和文件三个方面的测试。 数值测试 参数 说明 -eq 等于则为真 -ne 不等于则为真 -gt 大于则为真 -ge 大于等于则为真 -lt 小于则为真 -le 小于等于则为真
实例演示:
num1=100
num2=100if test $[num1]
- XFire框架实现WebService(二)
周凡杨
javawebservice
有了XFire框架实现WebService(一),就可以继续开发WebService的简单应用。
Webservice的服务端(WEB工程):
两个java bean类:
Course.java
package cn.com.bean;
public class Course {
private
- 重绘之画图板
朱辉辉33
画图板
上次博客讲的五子棋重绘比较简单,因为只要在重写系统重绘方法paint()时加入棋盘和棋子的绘制。这次我想说说画图板的重绘。
画图板重绘难在需要重绘的类型很多,比如说里面有矩形,园,直线之类的,所以我们要想办法将里面的图形加入一个队列中,这样在重绘时就
- Java的IO流
西蜀石兰
java
刚学Java的IO流时,被各种inputStream流弄的很迷糊,看老罗视频时说想象成插在文件上的一根管道,当初听时觉得自己很明白,可到自己用时,有不知道怎么代码了。。。
每当遇到这种问题时,我习惯性的从头开始理逻辑,会问自己一些很简单的问题,把这些简单的问题想明白了,再看代码时才不会迷糊。
IO流作用是什么?
答:实现对文件的读写,这里的文件是广义的;
Java如何实现程序到文件
- No matching PlatformTransactionManager bean found for qualifier 'add' - neither
林鹤霄
java.lang.IllegalStateException: No matching PlatformTransactionManager bean found for qualifier 'add' - neither qualifier match nor bean name match!
网上找了好多的资料没能解决,后来发现:项目中使用的是xml配置的方式配置事务,但是
- Row size too large (> 8126). Changing some columns to TEXT or BLOB
aigo
column
原文:http://stackoverflow.com/questions/15585602/change-limit-for-mysql-row-size-too-large
异常信息:
Row size too large (> 8126). Changing some columns to TEXT or BLOB or using ROW_FORMAT=DYNAM
- JS 格式化时间
alxw4616
JavaScript
/**
* 格式化时间 2013/6/13 by 半仙
[email protected]
* 需要 pad 函数
* 接收可用的时间值.
* 返回替换时间占位符后的字符串
*
* 时间占位符:年 Y 月 M 日 D 小时 h 分 m 秒 s 重复次数表示占位数
* 如 YYYY 4占4位 YY 占2位<p></p>
* MM DD hh mm
- 队列中数据的移除问题
百合不是茶
队列移除
队列的移除一般都是使用的remov();都可以移除的,但是在昨天做线程移除的时候出现了点问题,没有将遍历出来的全部移除, 代码如下;
//
package com.Thread0715.com;
import java.util.ArrayList;
public class Threa
- Runnable接口使用实例
bijian1013
javathreadRunnablejava多线程
Runnable接口
a. 该接口只有一个方法:public void run();
b. 实现该接口的类必须覆盖该run方法
c. 实现了Runnable接口的类并不具有任何天
- oracle里的extend详解
bijian1013
oracle数据库extend
扩展已知的数组空间,例:
DECLARE
TYPE CourseList IS TABLE OF VARCHAR2(10);
courses CourseList;
BEGIN
-- 初始化数组元素,大小为3
courses := CourseList('Biol 4412 ', 'Psyc 3112 ', 'Anth 3001 ');
--
- 【httpclient】httpclient发送表单POST请求
bit1129
httpclient
浏览器Form Post请求
浏览器可以通过提交表单的方式向服务器发起POST请求,这种形式的POST请求不同于一般的POST请求
1. 一般的POST请求,将请求数据放置于请求体中,服务器端以二进制流的方式读取数据,HttpServletRequest.getInputStream()。这种方式的请求可以处理任意数据形式的POST请求,比如请求数据是字符串或者是二进制数据
2. Form
- 【Hive十三】Hive读写Avro格式的数据
bit1129
hive
1. 原始数据
hive> select * from word;
OK
1 MSN
10 QQ
100 Gtalk
1000 Skype
2. 创建avro格式的数据表
hive> CREATE TABLE avro_table(age INT, name STRING)STORE
- nginx+lua+redis自动识别封解禁频繁访问IP
ronin47
在站点遇到攻击且无明显攻击特征,造成站点访问慢,nginx不断返回502等错误时,可利用nginx+lua+redis实现在指定的时间段 内,若单IP的请求量达到指定的数量后对该IP进行封禁,nginx返回403禁止访问。利用redis的expire命令设置封禁IP的过期时间达到在 指定的封禁时间后实行自动解封的目的。
一、安装环境:
CentOS x64 release 6.4(Fin
- java-二叉树的遍历-先序、中序、后序(递归和非递归)、层次遍历
bylijinnan
java
import java.util.LinkedList;
import java.util.List;
import java.util.Stack;
public class BinTreeTraverse {
//private int[] array={ 1, 2, 3, 4, 5, 6, 7, 8, 9 };
private int[] array={ 10,6,
- Spring源码学习-XML 配置方式的IoC容器启动过程分析
bylijinnan
javaspringIOC
以FileSystemXmlApplicationContext为例,把Spring IoC容器的初始化流程走一遍:
ApplicationContext context = new FileSystemXmlApplicationContext
("C:/Users/ZARA/workspace/HelloSpring/src/Beans.xml&q
- [科研与项目]民营企业请慎重参与军事科技工程
comsci
企业
军事科研工程和项目 并非要用最先进,最时髦的技术,而是要做到“万无一失”
而民营科技企业在搞科技创新工程的时候,往往考虑的是技术的先进性,而对先进技术带来的风险考虑得不够,在今天提倡军民融合发展的大环境下,这种“万无一失”和“时髦性”的矛盾会日益凸显。。。。。。所以请大家在参与任何重大的军事和政府项目之前,对
- spring 定时器-两种方式
cuityang
springquartz定时器
方式一:
间隔一定时间 运行
<bean id="updateSessionIdTask" class="com.yang.iprms.common.UpdateSessionTask" autowire="byName" />
<bean id="updateSessionIdSchedule
- 简述一下关于BroadView站点的相关设计
damoqiongqiu
view
终于弄上线了,累趴,戳这里http://www.broadview.com.cn
简述一下相关的技术点
前端:jQuery+BootStrap3.2+HandleBars,全站Ajax(貌似对SEO的影响很大啊!怎么破?),用Grunt对全部JS做了压缩处理,对部分JS和CSS做了合并(模块间存在很多依赖,全部合并比较繁琐,待完善)。
后端:U
- 运维 PHP问题汇总
dcj3sjt126com
windows2003
1、Dede(织梦)发表文章时,内容自动添加关键字显示空白页
解决方法:
后台>系统>系统基本参数>核心设置>关键字替换(是/否),这里选择“是”。
后台>系统>系统基本参数>其他选项>自动提取关键字,这里选择“是”。
2、解决PHP168超级管理员上传图片提示你的空间不足
网站是用PHP168做的,反映使用管理员在后台无法
- mac 下 安装php扩展 - mcrypt
dcj3sjt126com
PHP
MCrypt是一个功能强大的加密算法扩展库,它包括有22种算法,phpMyAdmin依赖这个PHP扩展,具体如下:
下载并解压libmcrypt-2.5.8.tar.gz。
在终端执行如下命令: tar zxvf libmcrypt-2.5.8.tar.gz cd libmcrypt-2.5.8/ ./configure --disable-posix-threads --
- MongoDB更新文档 [四]
eksliang
mongodbMongodb更新文档
MongoDB更新文档
转载请出自出处:http://eksliang.iteye.com/blog/2174104
MongoDB对文档的CURD,前面的博客简单介绍了,但是对文档更新篇幅比较大,所以这里单独拿出来。
语法结构如下:
db.collection.update( criteria, objNew, upsert, multi)
参数含义 参数
- Linux下的解压,移除,复制,查看tomcat命令
y806839048
tomcat
重复myeclipse生成webservice有问题删除以前的,干净
1、先切换到:cd usr/local/tomcat5/logs
2、tail -f catalina.out
3、这样运行时就可以实时查看运行日志了
Ctrl+c 是退出tail命令。
有问题不明的先注掉
cp /opt/tomcat-6.0.44/webapps/g
- Spring之使用事务缘由(3-XML实现)
ihuning
spring
用事务通知声明式地管理事务
事务管理是一种横切关注点。为了在 Spring 2.x 中启用声明式事务管理,可以通过 tx Schema 中定义的 <tx:advice> 元素声明事务通知,为此必须事先将这个 Schema 定义添加到 <beans> 根元素中去。声明了事务通知后,就需要将它与切入点关联起来。由于事务通知是在 <aop:
- GCD使用经验与技巧浅谈
啸笑天
GC
前言
GCD(Grand Central Dispatch)可以说是Mac、iOS开发中的一大“利器”,本文就总结一些有关使用GCD的经验与技巧。
dispatch_once_t必须是全局或static变量
这一条算是“老生常谈”了,但我认为还是有必要强调一次,毕竟非全局或非static的dispatch_once_t变量在使用时会导致非常不好排查的bug,正确的如下: 1
- linux(Ubuntu)下常用命令备忘录1
macroli
linux工作ubuntu
在使用下面的命令是可以通过--help来获取更多的信息1,查询当前目录文件列表:ls
ls命令默认状态下将按首字母升序列出你当前文件夹下面的所有内容,但这样直接运行所得到的信息也是比较少的,通常它可以结合以下这些参数运行以查询更多的信息:
ls / 显示/.下的所有文件和目录
ls -l 给出文件或者文件夹的详细信息
ls -a 显示所有文件,包括隐藏文
- nodejs同步操作mysql
qiaolevip
学习永无止境每天进步一点点mysqlnodejs
// db-util.js
var mysql = require('mysql');
var pool = mysql.createPool({
connectionLimit : 10,
host: 'localhost',
user: 'root',
password: '',
database: 'test',
port: 3306
});
- 一起学Hive系列文章
superlxw1234
hiveHive入门
[一起学Hive]系列文章 目录贴,入门Hive,持续更新中。
[一起学Hive]之一—Hive概述,Hive是什么
[一起学Hive]之二—Hive函数大全-完整版
[一起学Hive]之三—Hive中的数据库(Database)和表(Table)
[一起学Hive]之四-Hive的安装配置
[一起学Hive]之五-Hive的视图和分区
[一起学Hive
- Spring开发利器:Spring Tool Suite 3.7.0 发布
wiselyman
spring
Spring Tool Suite(简称STS)是基于Eclipse,专门针对Spring开发者提供大量的便捷功能的优秀开发工具。
在3.7.0版本主要做了如下的更新:
将eclipse版本更新至Eclipse Mars 4.5 GA
Spring Boot(JavaEE开发的颠覆者集大成者,推荐大家学习)的配置语言YAML编辑器的支持(包含自动提示,
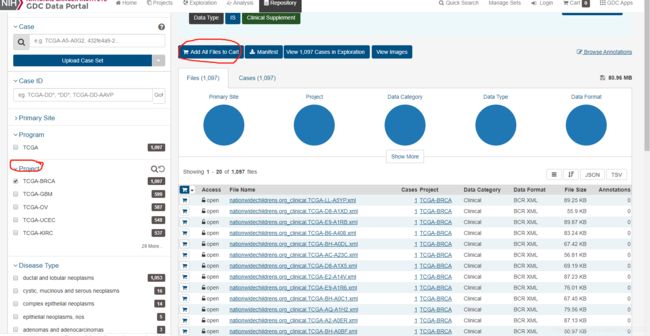
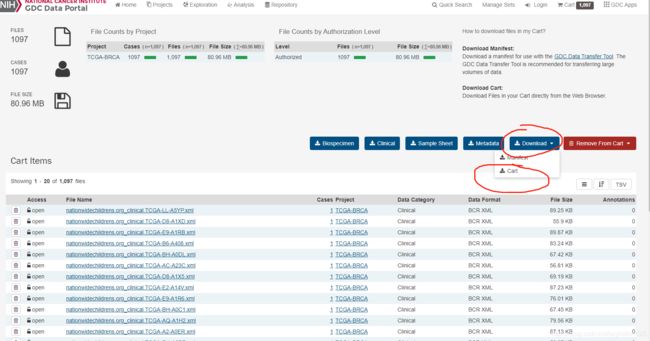
 我们选择临床数据在Data category 中选择clinical 最重要的在Data format 中一定要选择XML的]格式
我们选择临床数据在Data category 中选择clinical 最重要的在Data format 中一定要选择XML的]格式