[深度学习论文笔记]Efficient embedding network for 3D brain tumor Segmentation
Efficient embedding network for 3D brain tumor Segmentation
一种高效的脑肿瘤三维分割嵌入网络
英国皇家医科大学
Nov 2020
Multimodal Brain Tumor Segmentation Challenge 2020 (BRATS) BrainLes 2020
论文:https://arxiv.org/abs/2107.09842
摘要:
基于深度学习的三维医学图像处理存在数据匮乏的问题。因此,与二维自然图像分析相关的工作相比,该领域的研究是有限的,因为二维自然图像分析的数据集非常大。因此,强大而高效的二维卷积神经网络被开发和训练。在本文中,作者研究了一种转移二维分类网络性能的方法,以实现对脑肿瘤的三维语义分割。作者提出了一个不对称的U-Net网络,将EfficientNet模型作为编码分支的一部分。由于输入数据是三维的,编码器的第一层致力于缩小第三维,以适应EfficientNet网络的输入。对BraTS 2020挑战的验证和测试数据的实验结果表明,所提出的方法具有良好的性能。
问题动机:
胶质瘤是中枢神经系统最常见的原发性脑肿瘤。它们可以是低级的,也可以是高级的。高级别胶质瘤(HGG)是一种侵袭性的恶性脑肿瘤,生长迅速。此外,低级别胶质瘤(LGG)分为I级和II级。这些肿瘤占神经胶质肿瘤的不到50%,被认为是神经实质内孤立的肿瘤细胞,最初生长缓慢。胶质瘤的特点是浸润性,边界模糊。
然而,由于肿瘤大小、位置、组织学和生物学行为的差异,LGG的治疗仍然困难。此外,由于肿瘤显示出的压力,正常组织会变形,这使得区分正常组织和肿瘤区域更加困难。这些肿瘤的诊断和早期治疗对患者的生存至关重要。事实上,在大多数情况下,LGG患者在最初诊断后的10年内死亡。由于这些原因,精确和可重复的胶质瘤分割是研究脑MRI数据的先决步骤。
磁共振成像(MRI)已迅速成为一种基本的医学成像方式的疾病诊断。MRI在脑肿瘤诊断、患者随访、治疗评估和脑成像方面尤为有用。与使用MRI相关的主要优势是它能够获得非侵入性和非辐照的医学图像。它对对比度也非常敏感,并提供了一个极好的空间分辨率,完全适合探索大脑组织的性质。此外,成像很容易根据脑组织获得三维体积。
多模态脑肿瘤分割(BraTS)挑战旨在通过提供一个大型3D MRI注释LGG和HGG数据集,鼓励开发最先进的脑肿瘤分割方法。BraTS 2020训练数据集包括369例病例(293 HGG和76 LGG),每例均有4种模态:原生(T1)、对比后T1加权(T1Gd)、T2加权(T2)和T2流体衰减反转恢复(T2FLAIR)体积,这些数据来自多个(n=19)机构的不同临床方案和各种MRI扫描仪。每个肿瘤被分割为水肿、坏死和非强化肿瘤和活性/强化肿瘤。注释合并为3个嵌套子区域:Whole Tumor (WT)、Tumor Core (TC)和enhanced Tumor (ET)。
思路来源:
在过去的十年中,卷积神经网络(cnn)在生物医学图像分割中的表现超过了所有其他传统方法。特别是U-Net架构,因为最近医学图像分割挑战的大多数获奖贡献都是围绕U-Net构建的。
这项工作的目的是研究如何在二维图像分析中重用文献中存在的强大的深度卷积网络。事实上,许多强大的2D分类网络都是在非常大的数据集上训练好的。通过迁移学习,这些预先训练好的网络可以很容易地用于其他分类问题。然而,将分类网络的学习能力和特征检测能力转移到其他类型的问题上并不明显。特别是在使用卷积编码器-解码器体系结构(如U-Net)的问题中,这包括将预先训练的模型集成为编码器分支的一部分。当分类器和被处理网络的维度相同时(即当它们处理的数据具有相同的维度时),分类器的集成几乎是立即的。否则,需要一个自适应过程来适应分类器来处理所研究的图像的尺寸。
在文献中可以找到相关的工作,其中3D医学图像的分割是基于二维网络,其编码器在ImageNet上进行了预训练。尽管利用了预先训练的编码器,这些网络并没有集成3D的空间相干性,因为它们逐片处理数据。在作者的例子中,目的是研究如何将2D图像上强大且预先训练的网络的技能转移到处理3D图像的卷积编码器解码器中,而不丢失整个三维的一致性。众所周知,二维图像分类领域已经得到了很好的研究和发展,这通常是因为有大量标注的二维自然图像数据集。这种大型3D数据库的公众可用性是不存在的,在医疗领域更是如此。通过在3D问题中重新使用这些强大且经过充分研究的网络来利用它们是一个值得研究的想法。
小结:
提出了一种有效的方法,在不丢失三维一致性的前提下,将任何二维分类体系结构转换为三维分割目的。
提出的思想是可推广的,以便将任何低维分类体系结构集成到另一个高维体系结构,而不失去空间一致性。
主要方法:
提出的分割方法遵循卷积编码器-解码器体系结构。它是由一个非对称的大编码器提取图像特征和一个较小的解码器重建分割掩码。作者在编码器分支中嵌入了提出的称为EfficientNet的网络。
数据预处理:
由于GPU内存的限制和耗时的计算,作者不得不采取一些预防措施。作者分别处理每个模态,并通过使用最大的尺寸(192 × 160 × 108)减少背景来调整图像的尺寸,并将batchsize设置为1。作者不使用任何额外的训练数据,只使用提供的训练集。将所有输入图像归一化,使其均值和单位方差均为零。
由于EfficientNet模型的范围从0到7,作者仅通过测试基线EfficientNet- b0来限制在这个初步工作中的实验。实际上,这个模型在性能和复杂性之间做出了妥协。在这个阶段使用EfficientNet-B7需要更多的资源,这个选择可能会使模型变得非常复杂。在任何情况下,如果所提议的体系结构与这个基线模型工作得很好,那么它对EfficientNet-B7的推广将是直接的,并且肯定会提高性能。(即用简单的EfficientNet)
编码器分支:
编码过程有两个步骤。首先,作者将三维数据编码为二维数据,同时保持高度和宽度的原始大小,仅将深度压缩为3个通道。其次,数据准备好开始第二步编码,这就是没有完全连接层的EfficientNet网络。
![[深度学习论文笔记]Efficient embedding network for 3D brain tumor Segmentation_第1张图片](http://img.e-com-net.com/image/info8/883a3f0380f642a68ba57aaeb447c71a.jpg)
图1:拟议的网络架构示意图。输入是一个单通道裁剪的3D MRI。片间编码器和解码器都由一系列具有群范数规格化的残块组成。解码器的输出有三个通道,与输入通道的空间大小相同。在每个EfficientNet块下显示了相应的输出特征维度。
如图1所示,EfficientNet用块表示,与原始版本相同。但是,只表示跳跃连接层中涉及的块。片间编码部分使用卷积块,卷积块由两个卷积层组成,分别带有归一化和ReLU,然后是跳跃连接。在[基于自动编码器正则化的三维MRI脑肿瘤分割]的工作之后,作者选择使用组归一化,它将通道分成组,然后使用每组的均值和方差进行归一化。它似乎比传统的批处理规范化性能更好,特别是当批处理大小很小的时候。
作者假设输入体积的宽度为W,高度为H,深度为D,有C个通道。数据经过3D-2D压缩步骤。因此,深度通过因子3、3和4减小,最终达到3的深度大小,这与EfficientNet所需的通道数量相对应。在这个收缩过程中,单个3D批的宽度和高度没有修改。根据三维数据,可以改变和调整不同的深度还原因子。在作者的研究中,维度的变化如表1所示。在此过程后,通过二维收缩过程得到的缩减数据作为EfficientNet模型的输入。
 )
)
表1:尺寸缩小
解码器分支:
与编码部分不对称,解码器完全由同质块组成,如图1所示。显然,连接到EfficientNet的解码部分是一个2D解码器,而片间解码是一个3D解码器。解码器的每一层首先对空间维度进行上采样,将特征数量乘以2倍,然后跳过连接。一个sigmoid函数被用来激活解码器的输出,解码器有三个通道,对应于与输入具有相同空间大小的类的数量。
损失函数:
许多网络都是用交叉熵损失函数训练的,然而结果的描绘可能不是理想的dice得分。作为一种替代方案,可以使用一个soft-dice损失函数来训练所提出的网络。虽然在文献中存在几种dice损失的公式,作者更喜欢使用软dice损失,它在过去的[基于自动编码器正则化的三维MRI脑肿瘤分割]分割挑战中给出了良好的结果。软dice损失函数是可微的,给出如下:
![]()
Ptrue和Ppred分别代表ground truth和预测标签。脑MRI分割是一项具有挑战性的任务,部分原因是由于严重的失衡。在整个训练过程中,仅使用固定损失函数、交叉熵或dice来解决这个问题并不是最佳策略。因此,两个损失函数的线性组合通常被认为是最佳实践,并生成更稳健和最优的分割模型。在实际应用中,最终损失函数为:
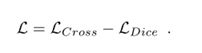
训练:
采用192 × 160 × 108体素的集中裁剪数据对该网络结构进行训练,确保每个切片的有用内容保持在裁剪区域的边界内,并在BraTS2020数据集上进行训练。由于材料性能不佳,作者将批量大小设置为1。训练是使用Adam优化器进行的,以内存要求很少而闻名,初始学习率为10 - 4,当损失50个epoch没有改善时,学习率降低了10倍。
实验与结果:
在Tensorflow上设计了拟议的网络,并在BraTS 2020的368个训练案例上运行。作者分别处理这四种模式,并将其平均输出。验证数据集用于测试模型在未见数据上的性能。共125例,分4种模式,无相应的分割。整体肿瘤(WT)、肿瘤核心(TC)和增强肿瘤(ET)的分割结果见表2。所有报告值均由在线评估平台(https://ipp.cbica.upenn.edu/)计算。图2显示了从验证数据集提取的典型分割结果。
![[深度学习论文笔记]Efficient embedding network for 3D brain tumor Segmentation_第3张图片](http://img.e-com-net.com/image/info8/5e03de4dc3804f1eb46386ea660e3501.jpg)
表2:BraTS 2020验证数据的结果。指标由在线评估平台计算。
![[深度学习论文笔记]Efficient embedding network for 3D brain tumor Segmentation_第4张图片](http://img.e-com-net.com/image/info8/bd7e7a57ca164243ba0fa7b0b679fa6c.jpg)
图2:BraTS验证集的典型结果(2020)。从左到右:T1ce轴位、冠状面末矢状面。黄色为肿瘤强化,红色为坏死,绿色为水肿。
WT类的平均Dice得分为84.13,在验证集上,所提出的模型似乎足够高效和准确地处理3D数据集上的训练和推理。另一方面,增强和核心肿瘤的结果效率较低。这可能是由于分别处理每一种核磁共振成像模式。
经过深度压缩后,网络保留了大脑的形状和结构,即使它们看起来有点模糊和退化。网络学会在或多或少的规则水平上提取三个轴向切片。作者注意到在三个已知的切片上有肿瘤的存在,通常涉及3通道压缩的轴向切片是表示肿瘤信息存在的切片。对肿瘤部位的高强度表明该网络的特征集中在对肿瘤的检测上。这些细节见图3和图4。
![[深度学习论文笔记]Efficient embedding network for 3D brain tumor Segmentation_第5张图片](http://img.e-com-net.com/image/info8/bf982916e4e84f8f8e7aac361b791d25.jpg)
图3:BraTS训练集的结果可视化(2020)。从上到下:Flair中一个独特样本的三个轴向切片,3D编码器对应的通道输出,3D解码器对应的通道输入,对应的标签。坏死和非增强的肿瘤中心为红色,增强的肿瘤为黄色,瘤周水肿为绿色
![[深度学习论文笔记]Efficient embedding network for 3D brain tumor Segmentation_第6张图片](http://img.e-com-net.com/image/info8/c8ba2a2b6d0e488aaddd3e7f682f2426.jpg)
图4:BraTS训练集的结果可视化(2020)。T1ce中一个独特样品的三个轴向切片,对应的3D编码器输出通道,对应的3D解码器输入通道和对应的标签。坏死和无强化的肿瘤中心为红色,增强的肿瘤为黄色,瘤周水肿为绿色
测试集的结果报告在表3。可以注意到,与验证集相比,ET和TC评分有了很大的改善,WT略有下降。从图3可以看出,轴向切片的三维压缩得到的图像在学习整个肿瘤特征的基础上具有很好的定向性,因此在FLAIR上可见区域。在图4中,发现压缩数据在学习增强和坏死肿瘤成分的结构特征时更加定向,这是T1CE中可见的区域。
![[深度学习论文笔记]Efficient embedding network for 3D brain tumor Segmentation_第7张图片](http://img.e-com-net.com/image/info8/fd0d96697ce84f54a40c53963a666a37.jpg)
表3:BraTS 2020测试数据的结果。指标由在线评估平台计算。
总结:
在本文中,作者介绍了一个通用的3D U-Net架构,通过重用和嵌入任何2D分类器网络来实现性能传输,编码器和解码器由两个阶段组成,3D输入数据经过深度收缩的过程,将3D数据转换为2D数据,这个过程是一个连续的三维卷积块和最大池化只减少第三维,转换后的输出数据可以通过任何二维分类网络进行编码。此外,解码还经过2D解码阶段,然后是3D解码过程。由于有限的计算资源,作者调整了图像的大小,并使用四种特定模式的网络分别训练第四种模式。尽管如此,初步结果似乎是有希望的。