简单分析一下RSNNS包中的一个案例demo。这个文件的源码可以在RSNNS/demo中找到。
library(RSNNS)
set.seed(2)
data(iris)
#shuffle the vector
iris <- iris[sample(nrow(iris)),]
irisValues <- iris[,1:4]
#irisTargets <- decodeClassLabels(iris[,5])
irisTargets <- decodeClassLabels(iris[,5], valTrue=0.9, valFalse=0.1)
iris <- splitForTrainingAndTest(irisValues, irisTargets, ratio=0.15)
#normalize data
iris <- normTrainingAndTestSet(iris)
#parameterGrid <- expand.grid(c(3,5,9,15), c(0.00316, 0.0147, 0.1))
parameterGrid <- expand.grid(c(3,5,9,15), c(0.00316, 0.0147, 0.1))
colnames(parameterGrid) <- c("nHidden", "learnRate")
rownames(parameterGrid) <- paste("nnet-", apply(parameterGrid, 1, function(x) {paste(x,sep="", collapse="-")}), sep="")
models <- apply(parameterGrid, 1, function(p) {
mlp(iris$inputsTrain, iris$targetsTrain, size=p[1], learnFunc="Std_Backpropagation",
learnFuncParams=c(p[2], 0.1), maxit=200, inputsTest=iris$inputsTest,
targetsTest=iris$targetsTest)
})
par(mfrow=c(4,3))
for(modInd in 1:length(models)) {
plotIterativeError(models[[modInd]], main=names(models)[modInd])
}
trainErrors <- data.frame(lapply(models, function(mod) {
error <- sqrt(sum((mod$fitted.values - iris$targetsTrain)^2))
error
}))
testErrors <- data.frame(lapply(models, function(mod) {
pred <- predict(mod,iris$inputsTest)
error <- sqrt(sum((pred - iris$targetsTest)^2))
error
}))
t(trainErrors)
t(testErrors)
trainErrors[which(min(trainErrors) == trainErrors)]
testErrors[which(min(testErrors) == testErrors)]
model <- models[[which(min(testErrors) == testErrors)]]
model
summary(model)
这个过程还是比较简洁明了的。首先导入数据并且对数据进行打乱,因为原始iris的数据是根据类型排序的,洗牌还是有必要的。
irisTargets <- decodeClassLabels(iris[,5], valTrue=0.9, valFalse=0.1)
这个地方对标签进行了处理,先看一下这个处理的结果是什么。
> head(irisTargets)
setosa versicolor virginica
[1,] 0.9 0.1 0.1
[2,] 0.9 0.1 0.1
[3,] 0.9 0.1 0.1
[4,] 0.9 0.1 0.1
[5,] 0.9 0.1 0.1
[6,] 0.9 0.1 0.1
也就是说,如果是对应类别就置0.9,不是的话就置0.1,这个数值根据valTrue和valFalse来设定。
iris <- splitForTrainingAndTest(irisValues, irisTargets, ratio=0.15)
这个函数将数据集合划分成训练集和测试集,其中测试集大小根据ratio参数确定。此处就是取数据集的15%。训练集合又分为inputs(特征值)和targets(目标分类结果),测试集也同样分为这两个集合。
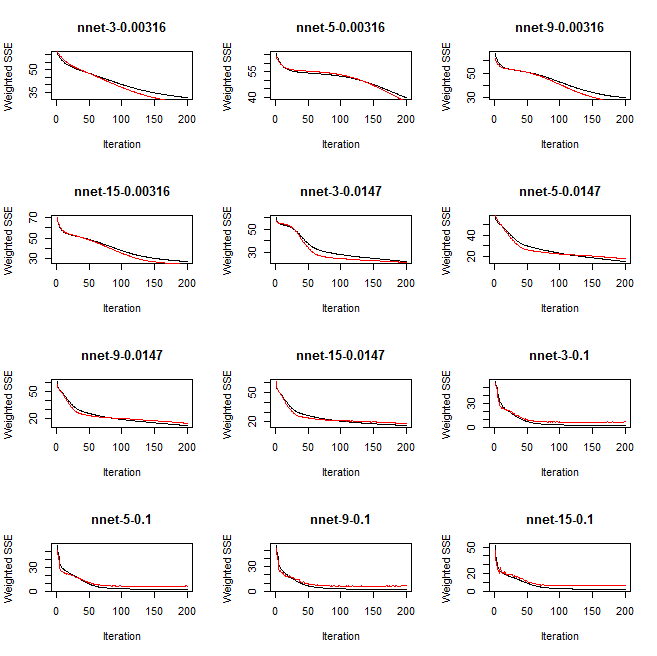
为了展示在不同的参数下,运行的效果,案例设置了12组参数。
parameterGrid <- expand.grid(c(3,5,9,15), c(0.00316, 0.0147, 0.1))
expand.grid()函数让两个向量间组合。看一下这个组合的结果
> expand.grid(c(3,5,9,15), c(0.00316, 0.0147, 0.1)) Var1 Var2 1 3 0.00316 2 5 0.00316 3 9 0.00316 4 15 0.00316 5 3 0.01470 6 5 0.01470 7 9 0.01470 8 15 0.01470 9 3 0.10000 10 5 0.10000 11 9 0.10000 12 15 0.10000
于是这就得到了12组不同的参数。其中第一列是隐藏神经元个数,第二列是学习率。
models <- apply(parameterGrid, 1, function(p) {
mlp(iris$inputsTrain, iris$targetsTrain, size=p[1], learnFunc="Std_Backpropagation",
learnFuncParams=c(p[2], 0.1), maxit=200, inputsTest=iris$inputsTest,
targetsTest=iris$targetsTest)
})
此时根据不同的参数进行测试。
for(modInd in 1:length(models)) {
plotIterativeError(models[[modInd]], main=names(models)[modInd])
}
然后利用plotIterativeError()函数绘制迭代误差。其中IterativeFitError (as black line),
IterativeTestError (as red line)。
最后看一下绘制的图