GCN项目的笔录
项目流程:
总体概括为几大步:1.数据集的准备工作-->2.构建生物分子网络-->3.构建图神经网络模型-->4.数据预测与模型训练
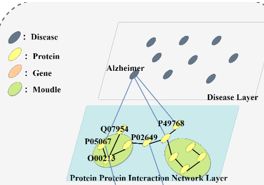
1)在列表中,选取了110种疾病类型;16156种蛋白质节点;
在创建的异构图bghg中,存在307156条蛋白质和蛋白质相互作用PPI的边;882条蛋白质和疾病关联(pdr)的边。这些边均是已经被证实的。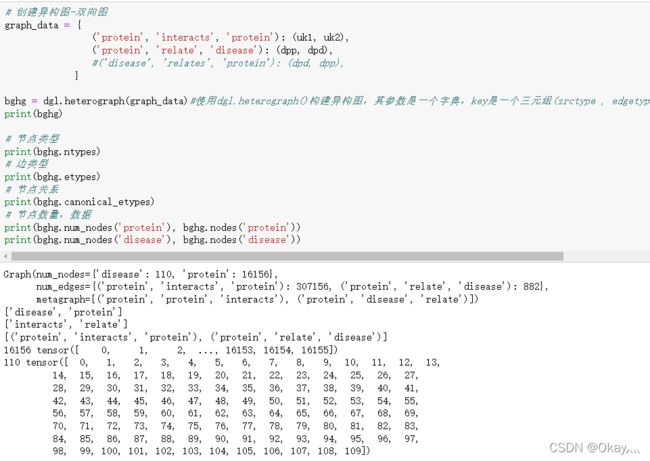
2)构造特征矩阵
①matrix:共16156行128列;对应蛋白质;每一行代表一个蛋白质的128维特征值。
②mm:共110行,128列; 对应疾病;每一行代表一个疾病的128维特征值。
这两个矩阵用于为创建的异构图中的每个节点添加它们应有的特征。
3)构建图神经网络模型--利用DGL构建GCN
4)对模型进行训练
一、首先,我们需要构建正负样本图,在进行数据集的分类之前,有一些准备工作要做。我们已经选取了882条蛋白质与疾病存在关联(pdr)的边,标签均设为1;作为正样本图。然后,经过设计,从二层网络(蛋白质与疾病,如下图)中,随机的从1773418条未连边中选择882条pdr,标签均设为0;作为负样本图。(这是我们进行模型训练时的分类思想!)
二、然后,设置训练集和测试集。
问题:上面这段负采样的方法是否有一点问题,那就是在全局图上进行了负采样,这样就将测试集中的数据也进行了负采样,理论上讲应该在训练集上进行负采样,对测试集不可见。
三、接下来,我们对数据集进行划分。
在机器学习建模中,通常需要将数据集划分为两部分:
①训练集:已经有标签的数据。
②测试集:无标签的数据。(二者相互独立)
为了防止“拟合”--模型在训练过程中表现良好,但在实测中却不如训练结果。因此,人们将训练集一分为二,再次划分:
①训练集 ②验证集;目的是在实际进行测试之前就进行一次自我评估。
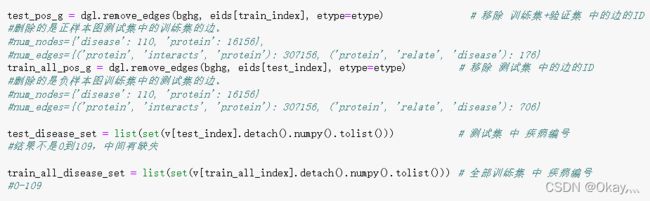
在这里,使用的是“不放回--K折交叉抽样验证”---KFold,将训练集划分为5份,训练集为4份,测试集为1份。五次划分结果如下:①eids_train:705;eids_test:177 ②eids_train:705;eids_test:177
③eids_train:706;eids_test:176④eids_train:706;eids_test:176⑤eids_train:706;eids_test:176
四、接下来,我们设计正负样本图。
主要采用的就是正负样本图对比来训练节点特征,以此来获得一个让正负样本图区别最大的网络模型。先选取生物分子网络中已被证实的疾病与蛋白质之间的连边作为正样本图,选取(疾病与蛋白质)不存在连边的某些疾病与蛋白质作为负样本图。在训练过程中,需要从graph中删除测试集的边,从graph中删除训练集的边,使用dgl.remove_edges。
各个变量的含义:
训练集中:设计了正负样本图,其中train_all_pos_g代表正样本图,它包括110个疾病节点和16156种蛋白质节点,pdr=706;train_all_neg_g代表负样本图,pdr=1757291。
测试集中:设计了正负样本图,其中test_pos_g代表正样本图,它包括110个疾病节点和16156种蛋白质节点,pdr=176;train_all_neg_g代表负样本图,pdr=1402516。
五、训练。
在定义了节点表示以及边的计算方法之后,就可以进行损失函数的计算,以及评价模型了(所有的函数定义代码前部分已经设计好了)。