R语言实现随机森林
R语言实现随机森林
- 数据介绍
- 一、相关R包的下载
- 二、实现过程
-
- 1.数据读取与数据集划分
- 2.构建随机森林模型
- 3.模型验证
- 总结
数据介绍
本问使用的数据集与R语言实现决策树的数据集相同,详情可参考这篇文章.
一、相关R包的下载
本文实现随机森林及相关图形绘制的R包如下:
library(randomForest)
library("pROC")
二、实现过程
1.数据读取与数据集划分
read.table("D:\\Rprojects\\tree.csv",header=TRUE,sep=",")->mydata #读取数据
mydata$group<-factor(mydata$group)#数据处理
sub<-sample(1:392,260)#260个样例作为训练集,其余作为测试集
train<-mydata[sub,]
test<-mydata[-sub,]
2.构建随机森林模型
利用randomForest包实现随机森林:
mydata.rf<-randomForest(group~.,data=train,importance=TRUE,proximity=TRUE)
计算得到各指标的平均最小基尼指数:
importance(mydata.rf,type=2)
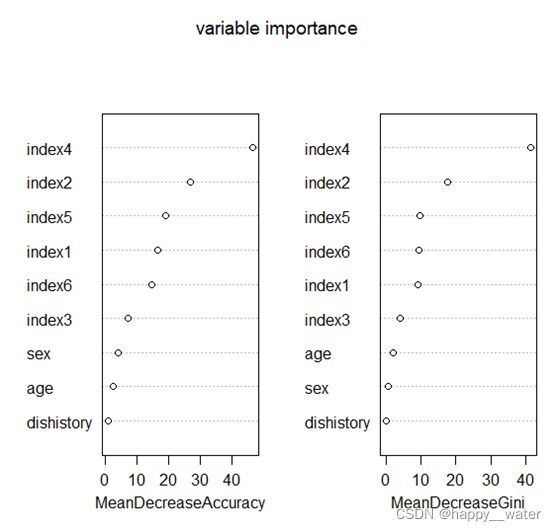
可使用图形的方式对指标的重要性进行可视化:
varImpPlot(mydata.rf, main = "variable importance")#变量的重要性判断
3.模型验证
对测试集数据进行预测,并输出混淆矩阵
#对测试集进行预测
pre_ran <- predict(mydata.rf,newdata=test)
#将真实值和预测值整合到一起
obs_p_ran = data.frame(prob=pre_ran,obs=test$group)
#输出混淆矩阵
table(test$group,pre_ran,dnn=c("真实值","预测值"))
| 混淆矩阵 | case | control |
|---|---|---|
| case | 36 | 3 |
| control | 0 | 93 |
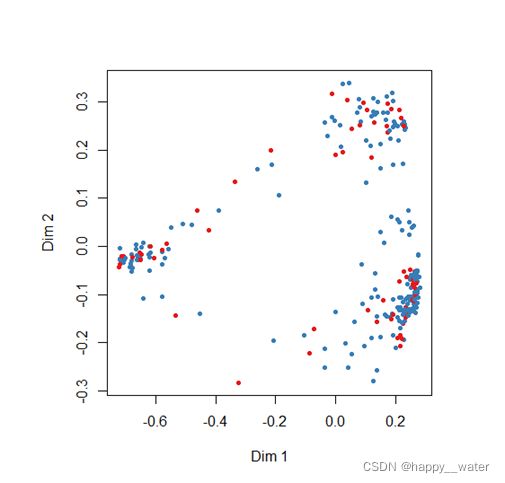
绘制ROC曲线图及多维尺度图:
#绘制ROC曲线
ran_roc <- roc(test$group,as.numeric(pre_ran))
plot(ran_roc, print.auc=TRUE, auc.polygon=TRUE, grid=c(0.1, 0.2),grid.col=c("green", "red"), max.auc.polygon=TRUE,auc.polygon.col="skyblue", print.thres=TRUE,main='随机森林模型ROC曲线')
MDSplot(mydata.rf,test$group)
完整代码如下:
library(randomForest)
library("pROC")
read.table("D:\\Rprojects\\tree.csv",header=TRUE,sep=",")->mydata #读取数据
mydata$group<-factor(mydata$group)
sub<-sample(1:392,260)#260个样例作为训练集,其余作为测试集
train<-mydata[sub,]
test<-mydata[-sub,]
mydata.rf<-randomForest(group~.,data=train,importance=TRUE,proximity=TRUE)
importance(mydata.rf,type=2)
varImpPlot(mydata.rf, main = "variable importance")#变量的重要性判断
#对测试集进行预测
pre_ran <- predict(mydata.rf,newdata=test)
#将真实值和预测值整合到一起
obs_p_ran = data.frame(prob=pre_ran,obs=test$group)
#输出混淆矩阵
table(test$group,pre_ran,dnn=c("真实值","预测值"))
#绘制ROC曲线
ran_roc <- roc(test$group,as.numeric(pre_ran))
plot(ran_roc, print.auc=TRUE, auc.polygon=TRUE, grid=c(0.1, 0.2),grid.col=c("green", "red"), max.auc.polygon=TRUE,auc.polygon.col="skyblue", print.thres=TRUE,main='随机森林模型ROC曲线')
MDSplot(mydata.rf,test$group)
总结
随机森林具有很高的预测准确率,对异常值和噪声具有良好的容忍度,且不会随着构建的决策树的增加而出现过拟合现象。但在引用随机森林方法时,也会产生一定限度内的泛化误差。随即特征(特征变量)的数目对泛化误差会产生一定影响。