2020-12-18 论文阅读
Deep learning-enabled breast cancer hormonal receptor status determination from base-level H&E stains
2020年发表于Nature Communications,论文传送门
摘要
对于初诊的乳腺癌患者,雌激素受体状态(ERS)是他们预后和治疗的重要分子标志物。在临床中,雌激素受体状态是通过免疫组化染色确定的。免疫组化昂贵又耗时,而且免疫组化过程的差异以及医生主观因素的影响都会导致结果不一致。相反,HE染色能够显示细胞形态,而且快速又廉价,制备过程变异少。本研究表明机器学习能够直接从细胞形态决定分子标志物的状态,即评估雌激素受体的状态。我们开发了一种基于多示例学习的深度神经网络,能够直接从HE染色数字病理图像决定雌激素受体状态。只利用WSI水平的标签就能够训练我们的模型,我们使用来自多个国家的3474名患者的数据对模型进行了训练,AUC达到了0.92。我们的方法有望利用人眼无法察觉到的生物信号来增强医生在肿瘤预后和治疗方面的判断能力。
引言
2018年全世界有200万女性被诊断为乳腺癌,其中有60万人因此死亡。多数浸润性乳腺癌患者都为雌激素受体阳性(即肿瘤细胞在雌激素或者孕酮存在下生长)。这些雌激素受体阳性的患者通常能够受益于激素疗法,该疗法通过作用于雌激素信号通路发挥作用。美国国家综合癌症中心指南规定确定每个新诊断为乳腺癌患者的激素受体状态,包括雌激素受体状态,因为这对于临床决策至关重要。
在目前的诊断流程中,病理工作人员将患者的组织样本切片并染色,然后在显微镜下进行观察。通常使用HE染色进行初步诊断,然后针对分子标志物进行特定的染色以进行确诊和分型。对于乳腺癌患者,临床会对其雌激素受体状态进行检测,因为雌激素受体状态对于预后以及对于激素疗法的反应都至关重要。病理工作人员通过对病理标本进行免疫组化染色并对其进行观察,以确定雌激素受体状态。这一过程有几点局限性:首先,免疫组化染色昂贵又耗时;另外,检测的结果可能受到多方面因素的干扰,如染色的深浅,染色深浅达到可检测标准的细胞的比例等,由于工作人员操作和制片固定等存在的差异,或者染色时抗体的来源不同,以及工作人员操作熟练度的不同,这些都会造成检测结果的不同;最后,病理工作人员的决策过程易受主观因素的影响,因此会导致人为误差。以上的种种因素都会导致雌激素受体状态检测结果的不一致;有研究指出,确定雌激素受体状态的免疫组化染色检测中,大约20%的检测都是不准确的,这就意味着这些患者有可能无法接受到最佳的治疗。
我们发现在HE染色的切片中,肿瘤细胞的形态学特征中包含能够预测分子标志物的信息;另外,机器学习算法能够直接从HE染色的切片中预测雌激素受体的状态。
形态学是生物学的反映;这种情况下,依赖分子标志物进行的预测和依赖细胞形态进行的预测结果可能会有所不同。我们发现机器学习算法从HE染色切片中提取到的形态学特征能够对免疫组化染色中表达的分子标志物进行预测。
我们的算法使用临床可用的雌激素受体状态记录,并且不需要任何图像标注。近年来的研究已经表明可以从细胞形态学染色决定雌激素受体状态,但是这些研究都是基于单中心的组织微阵列数据(TMAs)。组织微阵列的创建需要病理工作人员人工选择感兴趣区域(ROIs)。相反,我们的方法能够自动从组织中选择感兴趣区域(ROIs),而且我们使用了大型,多中心的病理图像数据,我们的方法能够改善工作流程,并且不需要病理人员进行任何的人工标注。还有研究使用了TCGA的肺癌数据从病理图像中对分子标志物进行了预测。
使用HE染色的组织切片有许多好处。首先,它比免疫组化染色检查便宜;其次,不同中心之间的变异小;另外,在临床病理检查中被广泛使用。自动化确定雌激素受体状态,能够减少乳腺癌治疗过程中的失误,改善生存结局。另外,识别出和分子标志物相关的细胞学形态特征,这将为研究激素如何驱动肿瘤的生长提供生物学思路。
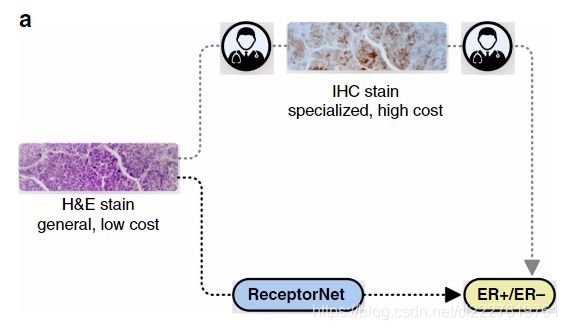
以往研究中,使用病理图像训练机器学习算法时,大多需要对病理组织图像进行标注,标注过程昂贵又耗时。由于病理人员无法直接从HE染色图像中判断雌激素受体状态,所以他们无法直接在HE染色图像上对雌激素受体状态进行标注。然而,如果我们通过免疫组化检测确定了肿瘤为雌激素受体阴性(ER-),那么我们可以认为它对应的HE染色图像中不包含雌激素受体阳性相关的特征。相反,如果免疫组化确定为ER+,那么我们可以认为HE染色图像中至少有一部分区域中包含雌激素受体阳性相关的特征。因此,我们可以使用HE染色图像作为输入,免疫组化检测结果作为标签。这个问题适合使用多示例学习(MIL),多示例学习近期已经被用于基于机器学习的病理诊断和预后。
多示例学习的目的是利用有标签的bag和无标签的instance进行学习。标签为阳性的bag中至少有一个instance为阳性;标签为阴性的bag中所有instance都为阴性。训练得到的多示例模型能够对未知的bag标签进行预测。我们使用多示例学习对来自病理图像的,由多个图像块构成的bag进行预测,预测其雌激素受体状态。
除了要求准确性以外,利用多示例学习对雌激素受体状态进行预测的模型还需要具有可解释性,即它要能够为我们指出哪些区域对于决策是重要的。从临床角度出发,可解释性对于获取医生的信任、对于建立稳健的决策系统、对于打消监管机构的顾虑都是非常重要的。从科学研究的角度出发,一个可解释的模型能够从HE染色图像中定位预测相关的图像块区域,识别出和激素驱动肿瘤生长相关的病理形态学特征。为了实现模型的可解释性,我们引入了注意力机制,我们的模型叫做ReceptorNet。

一只成熟的ReceptorNet是能够学会为重要的图像块分配较大的注意力权重的,它同时也会为不重要的图像块分配较小的注意力权重。通过对不同图像块的权重进行分析,我们可以确认哪些图像块可以用来对雌激素受体状态进行评估。
从0.5μm/pixel的分辨率下随机选择图像块构成bag,使用ReceptorNet预测其雌激素受体状态为阳性的概率。我们将数字病理图像切割为 256 × 256 256\times256 256×256像素大小的不重叠的图像块。ReceptorNet包括三个部分:① 一个特征提取器将一个bag中的每个 256 × 256 256\times256 256×256的图像块转变为一个512维的向量;② 一个注意力模块将一个bag中的所有图像块的特征整合为一个512维的特征向量,注意力加权的时候是根据其区分能力进行加权;③ 一个分类层根据输入的整合后的特征向量对该bag进行分类,计算其阳性的概率。我们在现有的attention-based MIL算法基础上做了改进,我们使用了cutout regularization, hard-negative mining, 以及我们提出的一种mean pixel regularization技术。我们不断迭代对ReceptorNet进行训练以预测雌激素受体阳性的概率,在测试的时候,我们从数字病理图像中采样多个bags,对它们的概率进行整合,以提高预测的准确率。
结果
定量评估
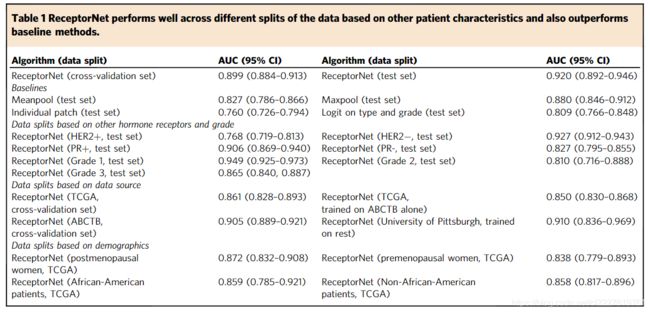
为了训练和测试我们提出的ReceptorNet,我们使用了两个不同的数据集:一个Australian Breast Cancer Tissue Bank (ABCTB)数据集,其包含来自2535个患者的2535张HE染色图像;另一个是TCGA数据集,包括来自939名患者的1014张HE染色图像。TCGA数据库的图像来自美国、波兰以及德国的42个不同的肿瘤机构。两个数据集都有免疫组化检查得到的激素受体状态的报告结果。这些数据集在样本制备,染色以及图像扫描方面都有较大的差异。移除一些严重被笔迹污染的图像以后,我们将这两个数据集整合在一起的数据集划分为训练集和测试集,其中训练集有2728个患者,测试集有671名患者。在训练集上进行训练和5折交叉验证,在测试集上进行评估。
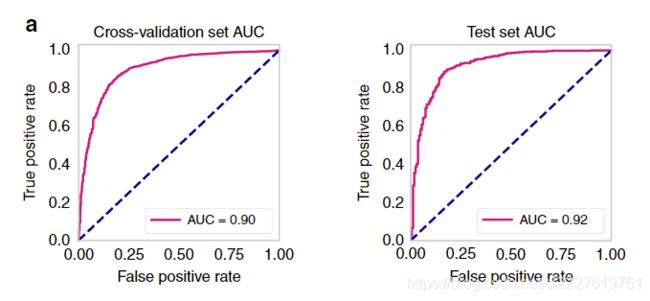
我们使用AUC对ER+/ER-的二分类问题进行评估,使用bootstrapping计算AUC的95%置信区间。在训练集的交叉验证的AUC结果为0.899 (95%CI为0.884-0.913),在测试集上的AUC为0.92 (95%CI为0.892-0.946)。在测试集上的阳性预测值和阴性预测值分别为0.932和0.741(阈值为0.25)。由于我们无法获取到原始的病理组织,所以无法进行精确的人机对比。
进行算法选择的时候,我们将ReceptorNet和Meanpool以及Maxpool进行了比较,这两种方法是两个传统的,被广泛使用的MIL方法。在测试集上,它们都要低于ReceptorNet的表现(使用DeLong test检验),其中Meanpool的AUC为0.827 (95%CI为0.786-0.866);Maxpool的AUC为0.880 (95%CI为0.846-0.912)。为了验证是否能从单独的图像块预测雌激素受体状态,我们搭建了一个基础的模型(移除了注意力模块),然后使用单独的图像块进行训练,损失为二分类交叉熵损失,得到的模型AUC为0.760 (95%CI为0.725-0.794),这表明使用注意力模块整合多个图像块信息能够提高模型的表现。我们还使用病理分型和肿瘤分级建立了一个logistic回归模型,该模型的AUC仅为0.809 (95%CI为0.766-0.848),显著低于ReceptorNet(DeLong test)。
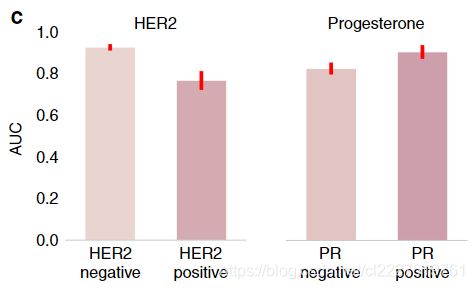
除了雌激素受体以外,孕酮受体阴性(PR-)和人类表皮生长因子受体2阴性(HER2-)也会影响肿瘤的生长,因此会影响HE染色组织中的病理形态学结构。由于HER2过表达是肿瘤中的主要转化机制,所以在HER2+的样本中,我们很难识别出ER相关的细胞形态学特征。事实上,和HER2+的样本相比,在HER2-阴性的样本中ReceptorNet会取得更好的结果(HER2-样本中AUC为0.927,HER2+阳性样本中AUC为0.768),对于PR也一样,在PR+样本中要更好一些。这反应了ER和PR之间存在高度的相关。考虑到研究的完整性,我们还将PR和HER2作为标签进行了同样的训练,结果PR的AUC为0.810,HER2的AUC为0.778.
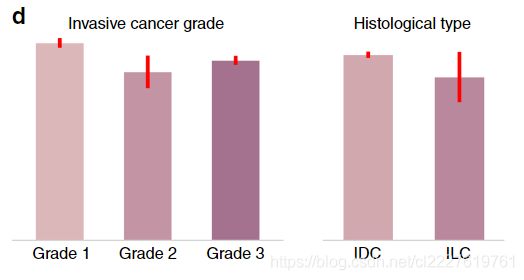
我们还发现在不同肿瘤分级中,模型的AUC有着显著的差异,比如grade I AUC为0.949,grade II AUC为0.810,grade III AUC为0.865. 相反,在导管癌和小叶癌中,AUC无差异。
在不同的数据集上,该模型的AUC是没有显著性差异的(F-test)。

为了进行额外的验证,我们仅使用ABCTB数据训练ReceptorNet,然后在TCGA数据集上进行验证,AUC为0.850 (95%CI为0.830-0.868),与合并数据集的结果相比,AUC的下降是合乎情理的。因为和ABCTB相比,TCGA数据集在染色和患者的人口学信息方面变异较大。ABCTB数据来自澳大利亚一个州的6个不同的肿瘤机构,而TCGA数据则来自于美国,波兰和德国的42个不同的肿瘤机构。最后,我们把训练集中来University of Pittsburgh的数据剔除,然后训练模型,再在剔除的那些数据上进行测试,得到AUC为0.91。
我们还在不同绝经期妇女以及不同种族分别进行了预测,发现AUC都没有差异,由于ABCTB数据没有人口学信息,所以只针对TCGA数据进行了计算。
定性评估
接下来,我们评估ReceptorNet提取到的哪些病理形态学特征对于雌激素受体状态的预测是重要的。我们邀请一个乳腺癌病理专家对那些注意力权重大的图像块进行评估。
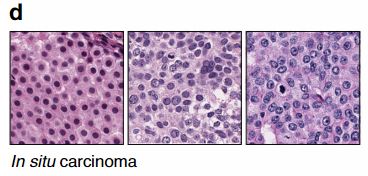
第一组预测ER+的图像块由均匀的细胞组成、细胞核小,核多形性可忽略不计,染色质模式变化不大,有丝分裂率低,这些都是低级别肿瘤的特征。

与此相反,一组预测ER-的图像块表现为中等实质性核,腺体缺乏,肿瘤生长迅速,这是高级别肿瘤的特征。
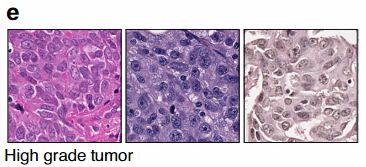
第二组ER+的图像块中细胞线性排列,细胞被间质包围,细胞核形态和大小各异,没有导管形成。这是浸润性小叶癌的特点。这也验证了有关研究报道浸润性小叶癌主要为雌激素受体阳性的结论。
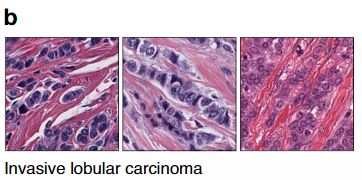
相反,ER-的图像块中没有图像块表现出浸润性小叶癌的特征。在第三组ER+的图像块中,图像块位于导管癌或者小叶癌原发病变灶内部,该区域为小的,大小均匀的肿瘤细胞,有适度的多形性,无间质。
其他的一些ER+的图像块则包括伴有干预反应性间质的肿瘤细胞,这些间质包括肿瘤相关成纤维细胞和肌成纤维细胞),以及一定数量的炎性细胞。这些浸润性癌细胞表现为轻度到中度的核多形性,并有零星的结缔组织,主要由胶原构成。
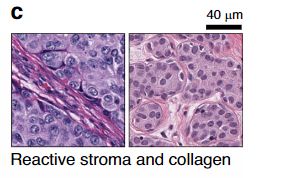
另一组ER-的图像块则包含坏死的碎片以及反应性淋巴细胞和巨噬细胞清除碎片。
综上所述,ReceptorNet发现了能够预测ER+的组织病理学形态特征,这些特征包括低级别肿瘤,浸润性小叶癌和原发乳腺癌。相关研究已经发现这些特征和ER+阳性乳腺癌有关,因此也验证了我们的模型提取到的特征的有效性。
ReceptorNet模型为以下组织类型分配了较低的注意力权重:含有脂肪组织的图像块、不含或含有较少肿瘤细胞的结缔组织图像块、少数肿瘤细胞和反应性间质夹杂在脂肪细胞之间的图像块以及含有吞噬了碎片和脂肪的巨噬细胞的图像块。
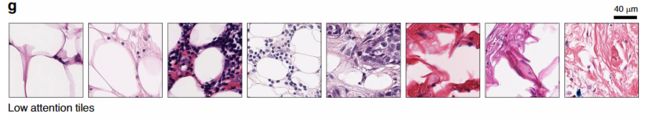
在预测雌激素受体状态的时候,我们的模型能够自动忽略这些形态学特征,并且训练过程中不需要任何人为的标注。
我们还利用t-SNE对bag的特征向量进行了可视化,结果表明RecptorNet学习到了能够将ER+和ER-很好的分开的特征。
讨论
总的来说,我们使用来自多个城市的HE染色病理图像和其对应的免疫组化检测标签构成的数据,训练了一个深度神经网络,用来对雌激素受体状态进行准确预测。我们在不同的肿瘤分级,分型以及不同人口学分层,以及其他不同受体存在状态下,对模型的稳健性进行了评估,我们发现其他受体状态以及肿瘤的分级能够明显影响模型的预测结果,而其他的一些因素则不会影响。从HE染色的图像中直接预测雌激素受体状态,能够降低结果的变异性,降低临床花费。另外,使用这种自动化的手段,我们可以加快开始治疗的时间,这也许会影响治疗的结果。在本研究中,我们为将来比较医生在有无这种机器学习手段辅助情况下的表现奠定了基础。更广泛的说,我们的研究可以提高医技人员的工作能力,并且能够利用病理人员肉眼察觉不到的生物标志物改善预后和诊断。
方法
数据集
我们对两个数据集进行了合并,一个数据集是Australian Breast Cancer Tissue Bank (ABCTB),其中有2535个患者的2535张HE染色图像;另一个数据集是TCGA数据集,其中有939名患者的1014张HE染色图像。两个数据集中都有经病理医生确认的免疫组化检查的ER, PR和HER2状态的检查报告。数字病理图像是在20x倍或者更高分辨率下扫描得到的。
数据准备
为了确保训练用的图像都包含组织,我们使用大津法进行图像分割,弃掉背景部分。然后我们在20x倍分辨率下从前景中提取 256 × 256 256\times256 256×256像素大小的图像块,只要图像块中包含1%的组织就被认为是前景部分。我们在提取图像块的时候是彼此没有重叠的。平均每张slide可以提取出19944个图像块,具体从949到67368不等。
模型结构
ReceptorNet模型包括三个彼此连接的部分:一个特征提取模块,一个注意力模块和一个分类模块。其中特征提取模块是去掉了softmax层的ResNet-50,紧跟两个全连接层将ResNet50得到的1000维特征转变为512维的特征向量,dropout为0.5。ResNet50的权重初始化自ImageNet预训练得到的权重,全连接层的权重使用He进行随机初始化。训练的时候,每个iteration将一个bag的N张图像块送入特征提取模块,产生 N × 512 N\times512 N×512的特征矩阵。这个特征矩阵被送入注意力模块进行整合。这个注意力模块包括两部分:首先是一个线性层将每个图像块的特征向量转变为一个128维的向量,并使用*tanh()*激活函数进行非线性操作,将特征的值变为-1到1之间;然后是再接一个线性层和一个softmax,得到注意力权重,注意力权重的取值范围在0-1,因此,对于一个bag中的N个图像块我们就可以得到一个N维的注意力权重,然后我们将 N × 512 N\times512 N×512的特征矩阵与这个N维的注意力权重做内积,就得到一个512维的特征向量。接下来将这个512维的特征向量输入到一个有512个神经元的全连接层,紧跟一个sigmoid函数,输出一个概率值,这个概率值就是含有N个图像块的bag的阳性概率。
# 最烦那些罗里吧嗦一堆公式还不给代码实现的人
import torch
from torch import nn
import torch.nn.functional as F
from torchvision.models import resnet50
class ReceptorNet(nn.Module):
"""
按照论文的描述,实现它的网络
"""
def __init__(self):
super(ReceptorNet, self).__init__()
self.L = 512 # 文中描述的512维向量维度
self.D = 128 # 文中描述的128维向量维度
self.K = 1 # 注意力网络的输出神经元
self.feature_extractor = resnet50(pretrained=True) # 由于PyTorch的resnet模型本身没有接softmax,所以这样就可以
self.two_fc_layers = nn.Sequential(
nn.Linear(self.feature_extrator.fc.out_features, self.L),
nn.ReLU(),
nn.Dropout(p=0.5),
nn.Linear(self.L, self.L),
nn.ReLU(),
nn.Dropout(p=0.5)
)
self.attention = nn.Sequential(
nn.Linear(self.L, self.D),
nn.Tanh(),
nn.Linear(self.D, self.K)
)
self.classifier = nn.Sequential(
nn.Linear(self.L*self.K, 1),
nn.Sigmoid()
)
def forward(self, x):
x = x.squeeze(0)
H = self.feature_extractor(x)
H = self.two_fc_layers(H)
A = self.attention(H)
A = torch.transpose(A, 1, 0)
A = F.softmax(A, dim=1)
M = torch.mm(A, H)
Y_prob = self.classifier(M)
Y_hat = torch.ge(Y_prob, 0.5).float()
return Y_prob, Y_hat, A
训练过程
我们从预先得到的图像块中随机选择50个作为一个bag,然后送入到ReceptorNet,进行模型的训练。我们对图像进行了数据增强操作。具体来说,以50%的概率对图像块进行左右翻转;进行0,90,180,270度等概率旋转;随机颜色扰动;进行cutout regression (length 为100)。损失为交叉熵损失,优化器为Adam,学习率为 1 × 1 0 − 5 1\times10^{-5} 1×10−5,权值衰减为 5 × 1 0 − 5 5\times10^{-5} 5×10−5,epoch为500,训练的时候每个epoch使用hard-negative mining 技术。由于我们的数据集存在明显的类别不平衡(ER+的数量是ER-的数量的3.7倍),所以我们每个epoch训练的时候,进行平衡抽样,保持ER+和ER-的图像块比例为50:50。为了减少过拟合,我们以0.75的概率随机将图像块替换为一种特定的图像,这种图像是这么得来的:它的所有像素设置为该数据集的所有图像的像素的均值。这种平均像素正则化方式显著提高了模型的表现。我们使用AUC,PPV和NPV来评价二分类任务的表现。灵敏度和特异度计算的时候使用的阈值为0.25。
和基线方法的比较
我们将我们的方法和两个被广泛使用的方法:Meanpool和Maxpool进行比较。在Meanpool中,一个bag中的N个图像块的特征通过取均值的方式进行特征整合。在Maxpool中,通过取特征最大值的方式进行特征的整合。这些方法在训练的时候都是基于ReceptorNet的架构,除了在我们的方法中将Meanpool或者Maxpool运算替换为了注意力模块。
Pathologist review
我们选择了一些能够显著预测ERS的图像块,然后请病理专家对其进行review。我们首先在测试集中穷尽抽样,来测试我们的模型。对于每个slide,我们对其bag中的图像块得到的512维整合后的特征进行获取,然后使用这个特征进行k-means聚类。对于每个聚类簇,我们计算ER+的比例。对于位于高比例ER+或者ER-的簇中的图像块,我们利用每个slide中注意力权重前1%的图像块进行k-means聚类。然后我们根据这些图像块距离聚类中心的距离进行排序,每个slide展示一个图像块,每个聚类簇最多展示五个图像块。这种操作能够确保我们选出来的图像块都具有高度可预测ER+状态的特征。然后请病理专家对这些图像块进行review,观察并记录它们的细胞形态学特征和组织结构。
硬件和软件
我们使用配有Nvidia P-100 Pascal显卡的计算机集群进行模型训练。使用OpenSlide进行图像块的提取。深度学习框架为PyTorch。使用Python的scikit-learn进行AUC的计算和其置信区间的计算。使用R进行统计学检验;使用R的Daim包进行DeLong检验。所有代码均使用开源工具进行编写。
统计学方法
略
数据集处理
我们移除了75个有严重笔迹的slide。