MICCAI 2020 | 基于3D监督预训练的全身病灶检测SOTA(预训练代码和模型已公开)...
关注公众号,发现CV技术之美
▊ 研究背景介绍
由于深度学习任务往往依赖于大量的标注数据,医疗图像的样本标注又会涉及到较多的专业知识,标注人员需要对病灶的大小、形状、边缘等信息进行准确的判断,甚至需要经验丰富的专家进行两次以上的评判,这增加了深度学习在医疗领域应用的难度。
目前,虽然有一些公开可用的数据集(如LIDC-IDRI、LUNA等),但是这些公开数据集存在图像数量少,偏倚度高的问题,导致模型不可避免地出现过拟合。为了解决该问题,一般会借助迁移学习使用ImageNet等大规模数据集上的预训练参数加快模型的收敛;然而,对于3D医学图像(例如CT、MRI等),目前并没有很好的3D模型预训练参数。
今天为大家介绍一篇针对3D医学影像中病灶检测任务的研究,该研究针对CT层面中的2D病灶检测问题提出了一种可以有效利用3D上下文信息的新框架,同时提出了一种预训练3D卷积神经网络的新思路。
文章发表于MICCAI 2020 [1],该研究在迄今规模最大的CT图像数据集NIH DeepLesion上进行了实验,取得了SOTA的病灶检测结果。当[email protected],相比当前SOTA方法提升3.48%,而相比2.5D的baseline方法,提升高达4.93%。
此外,实验还表明文中提出的有监督预训练方法可以有效提升3D模型训练的收敛速度,以及在小规模数据集上的模型精度。研究者在四个基准的3D医学数据集上进行了大规模的实验验证,结果表明对我们的预训练3D模型进行调优(finetune)不仅可以显著优于从头训练(training from scratch)的3D模型,而且与现有的最先进的自监督和全监督预训练模型相比,我们的模型效果在多数任务上也都具有明显优势。
预训练相关的代码以及基于ResNet3D-18的预训练模型已经公开,链接为:
https://github.com/urmagicsmine/CSPR
▊ 文章信息
Shu Zhang, Jincheng Xu, Yu Chun Chen, Jiechao Ma, Zihao Li, Yizhou Wang, and Yizhou Yu.
"Revisiting 3D Context Modeling with Supervised Pre-training for Universal Lesion Detection on CT Slices." In International Conference on Medical Image Computing and Computer-Assisted Intervention(MICCAI), pp. 542-551. Springer, Cham, 2020。
论文:https://arxiv.org/pdf/2012.08770.pdf
一、Revisiting 3D Context Modeling with Supervised Pre-training for Universal Lesion Detection in CT Slices.
1. CT关键层面中病灶检测中的3D上下文建模
对于自然图像,一般的深度学习技术都是基于2D CNN解决相关问题。但是对于医学图像,特别是3D医学图像(CT,MRI等),不同层面上的影像实质上是展示了同一个病灶或者器官的不同切面,这些不同层面上的信息具有高度的相关性和互补性。如果只在单个层面上进行图像检测,图像的大部分信息并没有有效利用,不但造成了信息的浪费,并且可导致诊断结果出现偏差。
对于CT影像的关键层面中的2D病灶检出问题,一个比较直接的解决方案是将连续的三层CT影像拼接成一个3通道的二维图像,输入到2D网络中进行病灶检测。该方法可以有效利用2D自然图像上的预训练模型提升特征表达能力,但是缺乏对多层面之间相关信息的处理和解释能力,很难对3D上下文建模,因此效果有限。
当前很多研究也意识到3D上下文建模的重要性,比如MVP-Net[2]等工作采用2.5D的方式来提升3D上下文建模能力,他们通过构造多通道的2D网络来融合更多连续层面(9层或者27层)的2D特征,实验结果表明这种方法对比单纯的2D方法有比较大的性能提升。
本文针对3D上下文建模这一问题则提出了一种更直接的解决方案,即直接对连续多层CT图像使用3D卷积进行特征提取,这样能更好地提取连续层面之间的结构和纹理特征。同时,为了解决3D卷积计算量大和训练收敛慢的问题,我们提出了针对性的模型结构改进和3D预训练方法。
2. 3D上下文信息增强网络(MP3D)
本研究针对CT图像关键层面中的病灶检测问题,开发了一个通用和高效的能够增强3D上下文信息建模的网络框架。首先我们提出一种改进的伪3D框架来对连续多层输入进行高效的3D上下文特征提取,配合一个组卷积变换模块,我们在特征输入到检测头之前将3D特征转换为2D特征,从而适配我们要进行的2D目标检测任务。
具体的,为了提高普通三维ResNet的计算和参数存储效率,我们采用伪3d残差网络(P3D ResNet)作为我们的原型骨干网络。伪3d卷积模拟的是3 × 3 × 3卷积在轴向视图切片上使用1 × 3 × 3卷积核加上3 × 1 × 1卷积核来构建相邻CT上的上下文信息(如图1所示)。
此外在关键层面病灶检测这种问题设定中,通常我们输入的图像层数(文中n=9)远小于轴位上的图像尺寸(通常是512*512)。我们在整个特征提取过程中,只对XY方向进行降采样,保持Z方向的尺寸不变,从而确保模型始终具有3D上下文建模的能力。
图1:MP3D网络框架3. 基于变维度转换的3D模型预训练
我们发现在NIH DeepLesion这种较大数据量的数据集中,在使用3D backbone进行建模时,虽然使用从头训练的方式也可以得到比较好的模型效果,但其收敛所需要的时间通常是Finetune模式的3倍以上。而在数据量较小的情况下,即便训练足够长的轮次,其收敛效果也难以和Finetune模式相比。
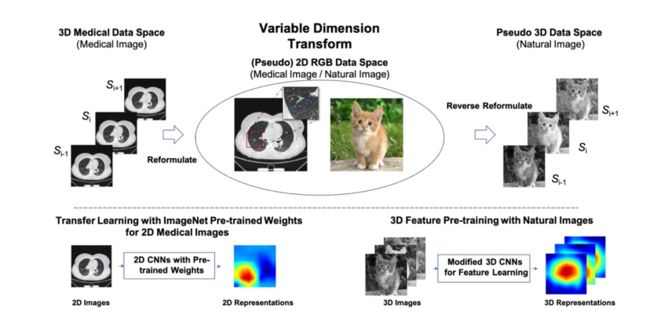
为此,我们设计了一种有监督的预训练方法来增强MP3D的训练以及收敛性能(图2)。本工作提出一种基于变维度转换的3D模型预训练方法:具体的我们将2D空间中的通道数(channel 维度)转换为3D空间中的层面数(depth维度),将原始具有色彩信息的RGB三通道二维图像转化到三维空间中的三个连续层面。
通过变维度转换,丰富的二维空间的颜色信息以3D结构信息的形式被保存下来。基于这些伪3D数据学习的3D卷积核则具有了表达存在于3D医学图像中的复杂3D结构和纹理信息的能力。
使用变维度转换模块得到的伪3D图像进行3D模型预训练的时候,和上文中介绍的检测模型类似,需要注意不要在Z方向进行降采样操作,确保Z方向始终保持depth=3,从而可以不断学习3D上下文信息。预训练的代理任务,根据使用的数据集不同可以是基于ImageNet的分类任务、基于COCO的检测任务或者基于分割数据集的分割任务等。
最终,通过该方法学习到的3D网络参数可以用于下游医学任务的微调以及优化,其迁移学习能力远超于在下游3D医学数据上的从头训练的实验效果。同时,由于可以利用现有2D数据集进行3D模型预训练,相比其他预训练方法,该方案避免了去采集大规模医疗数据,更不用做新的数据标注。
值得注意的是,在本文之前,将三个连续层面的3D医学图像合并成RGB图像,并使用在自然图像领域预训练好的2D模型进行特征表达已经成为处理医学影像的一个标准流程。本文提出的变维度转换可以认为是上述过程的一个逆变换。基于该逆变换,我们则可以有效的利用2D自然图像进行3D模型的预训练。后面的实验结论证明了这种变维度转换模块的有效性。
图2:基于变维度转换的3D模型预训练4. 关键层面病灶检出问题的实验设计与结果分析
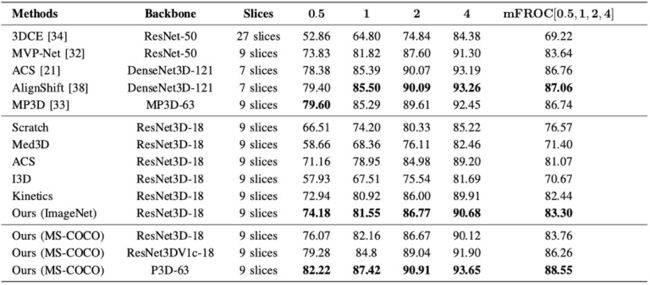
该研究采用NIH DeepLesion数据集作为模型的对比,模型的效果评价方法使用每幅图像在不同误报(FPs)下的灵敏度以及[email protected]。从表1中可以看出,与之前的SOTA方法相比较,我们的模型在不同的灵敏度上都超过了其他所有的方法,展示了三维模型的优越性,以及MP3D网络对上下文信息的建模能力。
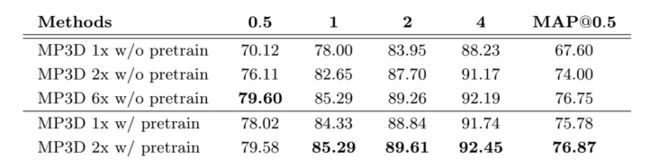
表1: 在DeepLesion测试集上,每幅图像在不同FPs下的灵敏度(%)为了进一步证明预训练方法的效果,表2中的定量结果可以看出,使用所提预训练方法可以在仅训练1/3的轮次(2x vs 6x的lr-schedule)的条件下取得和从头开始训练网络一样的效果。在同样使用1x和2x的训练时长条件下,使用预训练模型的结果要远好于从头开始训练的模型。
表2: 在DeepLesion测试集上,预训练方法的消融实验效果5. 预训练效果的实验设计与结果分析
本节中对上一工作中的预训练参数方法的有效性做出全面分析验证,相关实验细节请参考我们arXiv上的最新技术报告[3](Advancing 3D Medical Image Analysis with Variable Dimension Transform based Supervised 3D Pre-training:https://arxiv.org/abs/2201.01426)
目前在医学图像的预训练参数生成的任务中,由于缺乏大规模标注数据集,一般是采用自监督学习的方法,对未标注的数据使用对比学习的方法来学习预训练参数。然而,对于一些通用的医学任务,由于缺少一定程度的监督信息(语义不变性和判别性),会导致这些判别特征很难从进行挖掘。
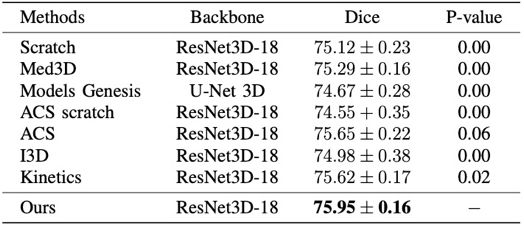
本研究为了全面对比不同预训练方法在不同数据集上不同任务上的效果。分别在LIDC-IDRI数据集,LITS肝脏数据集,NIH DeepLesion数据集上进行分类,分割以及检测任务。并且和在这些任务上的SOTA方法进行比较,我们的方法在不同数据集上均能取得较大提升。同时,在图3对肝脏器官分割的可视化中,所提方法能够完整地分割出相应的区域,证明了该方法的有效性和临床应用价值。
表3:LIDC以及LITS分割数据集效果 表4:LIDC分类数据集效果 表5: NIH DeepLesion检测数据集效果 图3:LITS可视化为了进一步验证我们的预训练方法在有限数据场景下的模型效果,我们基于20%,40%,60%,80% 以及100% NIH DeepLesion数据分别微调了预训练模型,用来比较使用我们提出的预训练模型做迁移学习与直接从头训练之间的性能差异。
图4的每一幅图展示了在某一种具体指标上的效果对比,其中最后一幅图展示的meanFROC是对检测模型整体效果的一个描述。可以看出在同样的训练轮次,我们的预训练模型在每一种数据量下都可以取得比从头训练更好的效果,而且随着数据量的缩小,这个效果的差距会被不断地放大。
这说明预训练模型对于小数据量的场景具有更突出的优化效果。此外,从最后一幅图可以看出,在使用不到40%的总数据量时,基于我们提出的预训练模型训练的模型效果好于在全量数据上从头训练的模型效果。因此,相对于从头开始训练的方法,该预训练模型可以节省大约60%的数据标注成本。
图4:预训练模型在不同数据量下的效果对比代码:https://github.com/urmagicsmine/CSPR
参考文献:
[1] Shu Zhang, Jincheng Xu, Yu Chun Chen, Jiechao Ma, Zihao Li, Yizhou Wang, and Yizhou Yu. "Revisiting 3D Context Modeling with Supervised Pre-training for Universal Lesion Detection on CT Slices." In International Conference on Medical Image Computing and Computer-Assisted Intervention(MICCAI), pp. 542-551. Springer, Cham, 2020.
[2] Li, Zihao, Shu Zhang, Junge Zhang, Kaiqi Huang, Yizhou Wang, and Yizhou Yu. "Mvp-net: Multi-view fpn with position-aware attention for deep universal lesion detection." In International Conference on Medical Image Computing and Computer-Assisted Intervention, pp. 13-21. Springer, Cham, 2019.
[3] Shu Zhang, Zihao Li, Hong-Yu Zhou, Jiechao Ma and Yizhou Yu. "Advancing 3D Medical Image Analysis with Variable Dimension Transform based Supervised 3D Pre-training"arXiv preprint arXiv: 2201.01426 2022 Jan 6.
END
欢迎加入「医学影像」交流群备注:Med