数据分析-聚类
@数据分析-聚类
目录
聚类概念
K-Means
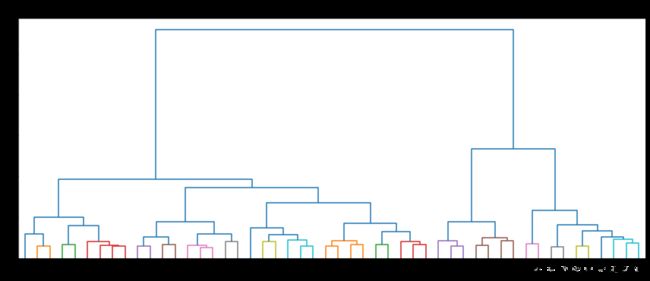
AgglomerativeClustering
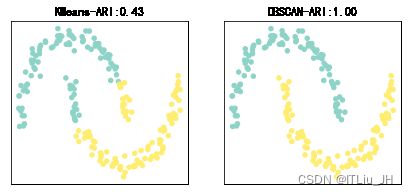
DBSCAN
MeanShift
SpectralClustering
评价
轮廓系数
兰德指数(rand index)
聚类概念
”物以类聚,人以群分“,聚类是典型的无监督学习方法。不同于分类,分类是有监督学习,样本都有标签,分类模型重点考查的是模型的泛化能力(predict);而聚类是按要求给样本加标签,重点考查模型聚类的效果,通常无训练集与测试集的划分。
聚类将数据集中相似的样本分到一组,每个组称为一个簇(cluster),相同簇的样本之间相似度较高,不同簇的样本之间相似度较低,样本之间的相似度通常是通过距离定义的,距离越远,相似度越低。
如根据学生的不同行为习惯,将学生分类,以便因才施教;根据全球各地观测到的气候特征,将全球划分为不同的气候区域 ;根据客户的消费记录将客户分成不同的消费群体。
聚类也常用于数据预处理,找出离群值。
常用的聚类方法有:K-Means,AgglomerativeClustering,DBSCAN,MeanShift,SpectralClustering等。
K-Means
K-Means聚类算法
1)从数据集中随机抽取k个样本作为初始聚类的中心,由这个中心代表各个聚类。
2)计算数据集中所有的样本到这k个中心点的距离,并将样本点归到离其最近的聚类里。
3)将聚类的中心点移动到各类的几何中心(即平均值)处。
4)重复第2步直到聚类的中心不再移动,此时算法收敛;或者迭代的次数达到上限。
kmeans算法时间、空间复杂度是:
时间复杂度:上限为O(tKmn),下限为O(Kmn)
空间复杂度:O((m+K)n)
其中,t为迭代次数,K为簇的数目,m为样本数,n为维数
距离的度量:
欧氏距离、曼哈顿距离、闵可夫斯基距离、余弦相似度等。
如何度量样本的距离,决定了聚类的效果,需要用一个量来定量的描述,具体的距离度量方式有很多,不同的场合使用哪种需要根据不同问题具体探讨。
K值的确定:
k一般不会设置很大。可以通过枚举,令k从2到一个固定值(如8),在每个k值上重复运行数次kmeans(避免局部最优解),并计算当前k的平均轮廓系数,最后选取轮廓系数最大的值对应的k作为最终的集群数目。
初始点的选择:
方法1:kmeans++
1、从输入的数据点集合中随机选择一个点作为第一个聚类中心
2、对于数据集中的每一个点i,计算它与最近聚类中心(指已选择的聚类中心)的距离D(i)
3、选择一个新的数据点作为新的聚类中心,选择的原则是:D(i)较大的点,被选取作为聚类中心的概率较大
4、重复2和3直到k个聚类中心被选出来
5、利用这k个初始的聚类中心来运行标准的k-means算法
方法2:选用层次聚类或Canopy算法进行初始聚类,然后从k个类别中分别随机选取k个点,来作为kmeans的初始聚类中心点
方法3:指定点
用法
from sklearn.cluster import KMeans
model_km=KMeans(n_clusters=3,random_state=10).fit(data)
auto_label=model_km.labels_
auto_cluster=model_km.cluster_centers_
metrics.silhouette_score(data_auto_sc,auto_label) #评价
'''
KMeans(
n_clusters=8,
*,
init='k-means++',
n_init=10,
max_iter=300,
tol=0.0001,
precompute_distances='deprecated',
verbose=0,
random_state=None,
copy_x=True,
n_jobs='deprecated',
algorithm='auto',
)
'''AgglomerativeClustering
层次聚类(hierarchical clustering)有两种常用的形式,自顶向下和自底向上。
其中,自底向上的主要做法是,在开始时,将每个样本视为一个簇,重复的合并最近的两个簇,直到簇的个数达到给定值
通常用谱系图来描述簇合并的过程。
度量簇间距离的方式有:
complete(完整连接法):两簇之间最远的样本之间的距离
average(平均连接法): 两簇间所有样本对的距离的平均值
single(单连接法):两簇之间最近的样本之间的距离
ward(离差平方和法):两簇的离差平方和之和(合并后的n*方差增量最小)
from sklearn.cluster import AgglomerativeClustering
model = AgglomerativeClustering(n_clusters=3,linkage=‘average’)
model.fit(X) # 训练模型
auto_label = model.labels_ # 输出模型结果
'''
AgglomerativeClustering(
n_clusters=2,
*,
affinity='euclidean',
memory=None,
connectivity=None,
compute_full_tree='auto',
linkage='ward',
distance_threshold=None,
compute_distances=False,
)
'''DBSCAN
DBSCAN主要思想是把样本空间中不同的高密度区域划分为不同簇。
对于一个样本点,如果在它的半径为的超球体(邻域)内的样本点个数大于,则认为这个样本点在一个高密度区域内,称这个点为核心对象。
对于两个都处在高密度区域的样本点,如果他们之间的距离足够近(即互相在对方的邻域内),则认为它们同属于一个簇。
算法:
1) 从一个点开始,对其半径为的邻域内的点赋予同一个簇标记,并寻找邻域内的其它核心对象
2) 对于找到的核心对象,重复步骤1
3) 重复上述过程直到无法找到新的高密度点为止,认为一个簇被完整找出
4) 从未被访问过的点开始,寻找下一个簇,最终没有出现在任何簇内的点被标记为孤立点。
from sklearn.cluster import DBSCAN
model = DBSCAN(eps=1.5, min_samples=4)
model.fit(X) # 训练模型
auto_label = model.labels_ # 保存模型结果
model.core_sample_indices_ # 核心对象的索引
model.components_ # 核心对象
'''
DBSCAN(
eps=0.5,
*,
min_samples=5,
metric='euclidean',
metric_params=None,
algorithm='auto',
leaf_size=30,
p=None,
n_jobs=None,
)
'''MeanShift
假设簇的质心在簇内点最密集的地方,寻找质心的方法是随机选择一个点(seed),从这个点开始不断的将这个点更新为这个点邻域内点的均值点,这样,这个点就会不断的向高密度区域移动,直至到达最密集处。
1) 初始从多个起始点(seed)开始的,如果几个不同的起始点经过移动最终聚集在一起,则认为他们找了同一个簇的质心,将他们合并。
通常不使用全部样本点作为seed,而是根据窗宽bandwidth作为网格间隔,选择起始点。选好簇质心后,将样本点分到最近的簇。
2) 在更新seed点的过程中,求均值时考虑临近点与当前seed点的距离,使用高斯核函数进行加权平均,即距离越近的点权重越大。
3) 高斯核需要输入一个参数bandwidth(窗宽),用来控制seed点邻域范围的大小
Bandwidth参数可选,如果不输入,则使用estimate_bandwidth函数来计算。默认是使用样本两两之间距离的0.3分位数作为窗宽。
Seeds参数同样,若没有设置且bin_seeding=True则使用get_bin_seeds函数来计算,默认使用bandwidth作为网格大小来选择起始点。
cluster_all为True时,孤立点会被分到最近的簇内;若为False,则对孤立点标记为-1
用法:
from sklearn.cluster import MeanShift
model = MeanShift(bandwidth=2)
model.fit(X)
data_label = model.labels_ #样本的标签
data_cluster = model.cluster_centers_ #簇的质心
'''
MeanShift(
*,
bandwidth=None,
seeds=None,
bin_seeding=False,
min_bin_freq=1,
cluster_all=True,
n_jobs=None,
max_iter=300,
)
'''SpectralClustering
谱聚类原理(转)
原文链接:https://blog.csdn.net/qq_24519677/article/details/82291867
谱聚类是一种基于图论的聚类方法,通过对样本数据的拉普拉斯矩阵的特征向量进行聚类,从而达到对样本数据聚类的母的。谱聚类可以理解为将高维空间的数据映射到低维,然后在低维空间用其它聚类算法(如KMeans)进行聚类。
谱聚类算法
输入:n个样本点 ![]() 和聚类簇的数目k;
和聚类簇的数目k;
输出:聚类簇![]()
(1)使用下面公式计算![]() 的相似度矩阵W;
的相似度矩阵W;
![]()
W为![]() 组成的相似度矩阵。
组成的相似度矩阵。
(2)使用下面公式计算度矩阵D;
![]() ,即相似度矩阵W的每一行元素之和
,即相似度矩阵W的每一行元素之和
D为![]() 组成的
组成的![]() 对角矩阵。
对角矩阵。
(3)计算拉普拉斯矩阵![]() ;
;
(4)计算L的特征值,将特征值从小到大排序,取前k个特征值,并计算前k个特征值的特征向量![]() ;
;
(5)将上面的k个列向量组成矩阵![]() ,
,![]() ;
;
(6)令![]() 是的第行的向量,其中
是的第行的向量,其中![]() ;
;
(7)使用k-means算法将新样本点![]() 聚类成簇
聚类成簇![]() ;
;
(8)输出簇![]() ,其中,
,其中,![]()
用法:
from sklearn.cluster import SpectralClustering
model= SpectralClustering(n_clusters=3)
model.fit(X)
data_label=model.labels_
'''
SpectralClustering(
n_clusters=8,
*,
eigen_solver=None,
n_components=None,
random_state=None,
n_init=10,
gamma=1.0,
affinity='rbf',
n_neighbors=10,
eigen_tol=0.0,
assign_labels='kmeans',
degree=3,
coef0=1,
kernel_params=None,
n_jobs=None,
verbose=False,
)
'''评价
轮廓系数
评价指标-轮廓系数:
轮廓系数结合了聚类的类内凝聚度Cohesion和类间分离度Separation,用于评估聚类的效果。该值处于-1~1之间,值越大,表示聚类效果越好。
对于每个样本点i,计算点i与其同一个簇内的所有其他元素距离的平均值,记作a(i),用于量化簇内的凝聚度。
选取i外的一个簇B,计算i与B中所有点的平均距离,遍历所有其他簇,找到最近的这个平均距离,记作B(i),即为i的邻居类,用于量化簇之间分离度。
对于样本点i,轮廓系数
s(i) = (B(i) – a(i))/max{a(i),B(i)}
计算所有i的轮廓系数,求出平均值即为当前聚类的整体轮廓系数,度量数据聚类的紧密程度。
若s(i)小于0,说明i与其簇内元素的平均距离小于最近的其他簇,表示聚类效果不好。如果a(i)趋于0,或者B(i)足够大,即a(i)< 用法: 在类别信息已知的情况下可以采用兰德指数评价聚类结果 给定真实类别信息C和聚类结果K: RI=(a+b)/(a+b+c+d) a:表示在C和K中都为同一类的样本对的数量 b:表示在C和K中都属于不同类的样本对的数量 c:表示在C中属于同一类,在K中属于不同类的样本对的数量 d:表示在C中属于不同类,在K中属于同一类的样本对的数量 取值为[0,1],越大聚类结果与真实情况越接近 兰德指数的缺点:在聚类结果随机产生的情况下,不能保证系数接近于0 调整兰德指数(adjusted rand index): ARI=(RI-E(RI))/(max(RI)-E(RI)) ARI取值范围为[-1,1],负数代表结果不好,越接近于1越好from sklearn.metrics import silhouette_score
silhouette_score(data,model.labels_)兰德指数(rand index)
from sklearn.metrics.cluster import adjusted_rand_score
adjusted_rand_score(y,clusters) #需要有真实的类别标签