多种方法对建模数据做特征选择
多种方法对建模数据做特征选择
-
- 前言
- 一:基于统计方法选取特征
- 二:基于机器学习方法选取特征
- 三:实验代码
- 四:递归消除法
- 五:总结
前言
特征选择和特征提取都属于降维,就是试图去减少特征数据集中的属性(或者称为特征)的数目,但是两者所采用的方式方法却不同。特征提取的方法主要是通过属性间的关系,如组合不同的属性得到新的属性,这样就改变了原来的特征空间。特征选择的方法是从原始特征数据集中选择出子集,是一种包含的关系,没有更改原始的特征空间。特征提取和特征选择都是从原始特征中找出最有效(同类样本的不变性、不同样本的鉴别性、对噪声的鲁棒性)的特征。
一:基于统计方法选取特征
1:两两之间比较
对某个特征 f f f ,假设数据集 [ x 1 , x 2 , . . . , x n ] [x_1,x_2,...,x_n] [x1,x2,...,xn] 存在 k k k 类 ( k ≤ n ) ( k\leq n ) (k≤n),观察f在任意两类数据集之间是否显著不同
常见的方法:参数检验有 t t t 检验, z z z 检验等;非参数方法有 W i l c o x o n Wilcoxon Wilcoxon 符号秩检验等等;还有就是观察 f 在两两不同类别组数据的平均值,用差异倍数来定夺是否表现不同。
2:总体一起选择
对某个特征 f f f ,假设数据集 [ x 1 , x 2 , . . . , x n ] [x_1,x_2,...,x_n] [x1,x2,...,xn] 存在 k k k 类 ( k ≤ n ) (k\leq n ) (k≤n),观察f在所有数据集之间是否显著不同
常见的方法:非参数检验之 K r u s k a l − W a l l i s Kruskal-Wallis Kruskal−Wallis检验;还有一种方法是基于变异系数 ( C V ) (CV) (CV)值,即求出每个类别中每个特征的变异系数,其中的最小值(或者均值)大于设定的阈值,可以把此特征视为此数据集的显著特征,当然 C V CV CV法对于无监督数据也一样适用。
- 解释CV
当需要比较两组数据离散程度小的时候,如果两组数据的测量尺度相差太大,或者数据量纲的不同,直接使用标准差来进行比较不合适,此时就应当消除测量尺度和量纲的影响,而变异系数可以做到这一点,它是原始数据标准差与原始数据平均数的比。 c v = σ u cv=\frac{\sigma}{u} cv=uσ 没有量纲,这样就可以进行客观比较了。
3:基于距离运算
这里我们之前的文章提到过,不再赘述!
基于距离运算筛选特征
4:基于卡方检验方法
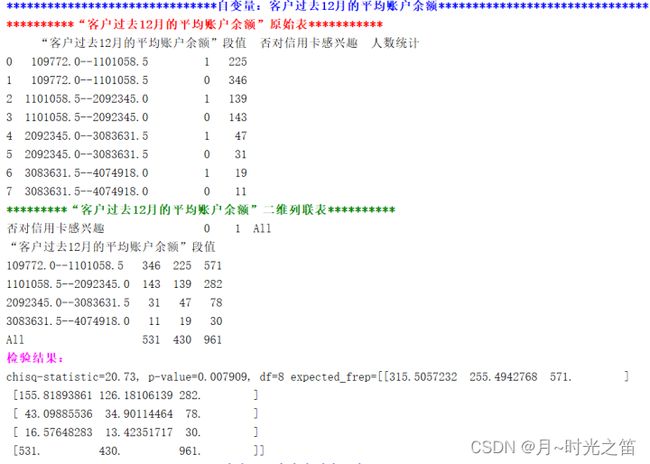
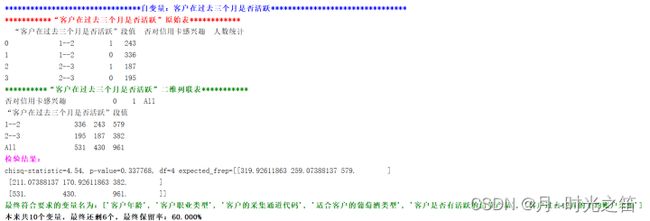
卡方检验我们都知道其原理,首先建立二维列联表,然后求的统计量,再结合概率分布求得显著性p值。同样此方法也是可以进行特征显著性检验的,但是此方法也是要有标签数据的,否则是建立不出来二维列联表的。接下来我们将基于实际数据集进行相关实验。
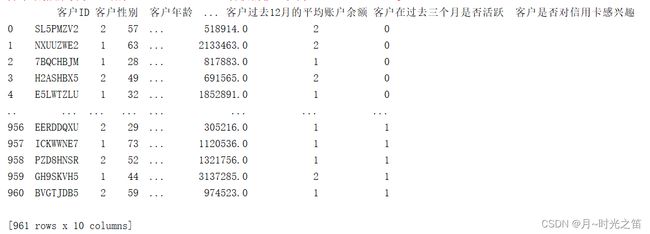
此次建模数据集大致如下图所示:
####二联表卡方检验
def Chi_test(col):
print('\033[1;34m{0:*^80}\033[0m'.format('卡方检验筛选变量'))
p_ls = []
for i in col:
print('\033[1;34m{0:*^80}\033[1;0m'.format('自变量:%s'%i))
datax = res3[i]
datay = res3['客户是否对信用卡感兴趣']
minx = datax.min()
maxx = datax.max()
LStemp1, LStemp2, LStemp3 = [], [], []
if i == '客户性别' or i == '客户在过去三个月是否活跃' or i == '客户是否有活跃的信贷产品':
interval = [1, 2, 3]##因为此字段下的变量只有两个值,离散结果只有两种,所以要分开处理!!
count = 0
for l in range(len(interval) - 1):
count = count + 1
forwardvalue = interval[l]
backvalue = interval[l + 1]
lstemp1, lstemp2 = [], []
for j, k in zip(list(datax), list(datay)):
if forwardvalue <= j < backvalue:
strmsg = str(round(forwardvalue, 2)) + '--' + str(round(backvalue, 2))
lstemp1.append(strmsg)
lstemp2.append(k)
# print('\033[1;31m段%s基础信息:%s\033[0m' % (count, lstemp1))
# print('\033[1;32m段%s违约信息:%s\033[0m' % (count, lstemp2))
LStemp1.append(lstemp1[0])
LStemp1.append(lstemp1[0])
LStemp2.append(1)
LStemp2.append(0)
LStemp3.append(lstemp2.count(1))
LStemp3.append(lstemp2.count(0))
else:
interval = np.linspace(minx, maxx, 5)
count = 0
LStemp1, LStemp2, LStemp3 = [], [], []
for l in range(len(interval) - 1):
count = count + 1
forwardvalue = interval[l]
backvalue = interval[l + 1]
lstemp1, lstemp2 = [], []
for j, k in zip(list(datax), list(datay)):
if forwardvalue <= j <= backvalue:
strmsg = str(round(forwardvalue, 2)) + '--' + str(round(backvalue, 2))
lstemp1.append(strmsg)
lstemp2.append(k)
LStemp1.append(lstemp1[0])
LStemp1.append(lstemp1[0])
LStemp2.append(1)
LStemp2.append(0)
LStemp3.append(lstemp2.count(1))
LStemp3.append(lstemp2.count(0))
df = pd.DataFrame({'“%s”段值' % i: LStemp1, '否对信用卡感兴趣': LStemp2, '人数统计': LStemp3})
print('\033[1;31m{0:*^40}\033[1;0m'.format('“%s”原始表' % i))
print(df)
cross_tab = pd.pivot_table(data=df, values='人数统计', index='“%s”段值' % i, columns='否对信用卡感兴趣', margins=True,
aggfunc=np.sum)
print('\033[1;32m{0:*^40}\033[1;0m'.format('“%s”二维列联表' % i))
print(cross_tab)
kf = chi2_contingency(cross_tab)
print('\033[1;35m检验结果:\033[0m')
print('chisq-statistic=%.2f, p-value=%.6f, df=%s expected_frep=%s' % kf)
p_ls.append(kf[1])
select_ls = []
for i,j in zip(p_ls,col):
if i <= 0.01:
select_ls.append(j)
print('\033[1;32m最终符合要求的变量名为:%s\033[0m'%select_ls)
print('\033[1;38m本来共%s个变量,最终还剩%s个,最终保留率:%.3f%%'%(data.shape[1]-1,len(select_ls),100*len(select_ls)/(data.shape[1]-1)))
return select_ls
res = Chi_test(col_ls3)
上图2-3就是检验的过程(截取其中两个),所以这种非参数检验方法同样可以进行数据筛选,但前提是一定要建立正确的列联表,理解检验方法逻辑。
5:其他方法
比如简单的有特征之间相关性计算,相关性高的多个数据,可以取其中之一,对于监督和无监督学习数据都适用。还有就是一些新方法其他论文中提到的比如Relief-Based特征选择,XGBSFS算法等等。
二:基于机器学习方法选取特征
这里主要还是对于有监督学习数据而言。
1:随机森林
随机森林特征重要性的原理解释(注:这里直接跳过决策树-随机森林算法相关基础知识,比如剪枝、袋外等)。
思想:判断每个特征在随机森林中的每颗树上做了多大的贡献,然后取个平均值,最后比一比特征之间的贡献大小。其中关于贡献的计算方式可以是基尼指数或袋外数据错误率。
- 基尼指数
k k k 代表第 k k k 个类别,共 K K K 个, p k p_k pk 代表类别 k k k 的样本权重,
基尼指数公式为: G i n i ( p ) = ∑ k = 1 K p k ( 1 − p k ) Gini(p)=\sum_{k=1}^{K}{p_k(1-p_k)} Gini(p)=∑k=1Kpk(1−pk) ,
那么对于特征 X j X_j Xj ,在节点 m m m 的重要性,用节点 m m m 分枝前后的基尼指数变化量表示:
V I M j m G i n i = G I m − G I l − G I r VIM_{jm}^{Gini}=GIm-GIl-GIr VIMjmGini=GIm−GIl−GIr ,这里 G I l GIl GIl 和 G I r GIr GIr 分别表示分枝后两个新节点的 G i n i Gini Gini 指数。
如果特征 X j X_j Xj 在决策树 i 中出现的节点在节点集合 M M M 中,那么 X j X_j Xj在第 i i i 棵树的重要性为:
V I M j i G i n i = ∑ m ∈ M V I M j m G i n i VIM_{ji}^{Gini}=\sum_{m\in M}VIM_{jm}^{Gini} VIMjiGini=∑m∈MVIMjmGini ,
假设随机森林共 n n n 棵树,那么 V I M j G i n i = ∑ i = 1 n V I M j i G i n i VIM_{j}^{Gini}=\sum_{i=1}^{n}{VIM_{ji}^{Gini}} VIMjGini=∑i=1nVIMjiGini ,最后,我们再做归一化处理:
r V I M j G i n i = V I M j G i n i / ∑ f = 1 F V I M t G i n i rVIM_{j}^{Gini}=VIM_{j}^{Gini}/\sum_{f=1}^{F}{VIM_{t}^{Gini}} rVIMjGini=VIMjGini/∑f=1FVIMtGini ,
其中分母为所有特征(共 F F F 个)的增益之和,分子为特征 X j X_j Xj 的基尼指数。
- 基于袋外数据OOB法
对于其中一棵树 T i T_i Ti ,用 O O B OOB OOB样本可以得到误差 e 1 e_1 e1 ,然后随机改变 O O B OOB OOB中的第 j j j 列,保持其他列不变,对第 j j j 列进行随机的上下置换,得到误差 e 2 e_2 e2 。至此,可以用 ∣ e 1 − e 2 ∣ \left| e_1-e_2 \right| ∣e1−e2∣ 来刻画特征 j j j 的重要性。其依据就是,如果一个特征很重要,那么其变动后会非常影响测试误差,如果测试误差没有怎么改变,则说明特征 j j j 不重要。 而该方法中涉及到的对数据进行打乱的方法通常有两种:
1:是使用uniform或者gaussian抽取随机值替换原特征;
2:是通过permutation的方式将原来的所有 N N N 个样本的第 i 个特征值重新打乱分布(相当于重新洗牌)。
2:Xgboost和Lasso
对于Xgboost模型的基础知识,我们知道Xgboost是一个优化的分布式梯度增强库,旨在实现高效,灵活和便携。它的很大特点一是在求解损失函数时候,使用了牛顿法,将损失函数泰勒展开到二阶,误差精度提高,当然带来的坏处就是计算变得更加复杂;二是损失函数中加入了正则化项,训练时的目标函数由两部分构成,第一部分为梯度提升算法损失,第二部分为正则化项;三是节点的分裂方式经过了优化;最后我认为则是处理缺失值做了不一样的逻辑替换。
那么基于树结构的Xgboost在进行属性特征重要性选择时,则根据结构分数的增益情况计算出来选择哪个特征作为分割点,而某个特征的重要性就是它在所有树中出现的次数之和。也就是说一个属性越多的被用来在模型中构建决策树,它的重要性就相对越高。
Lasso说白了就是简单线性回归的损失函数加入了L-范数,在特征过多的时候不容易产生过拟合现象。因为Lasso算法可以使特征的系数进行压缩并且可以使某些回归系数为0,即不选用该特征,因此可以进行特征选择,可以有效的解决各特征之间存在的多重共线性问题。
三:实验代码
关于统计方法的相关代码,请参考上一篇文章!
共同代码
from xgboost import XGBClassifier
import matplotlib.pylab as plt
from sklearn import ensemble
import seaborn as sns
import pandas as pd
import numpy as np
plt.rcParams['font.sans-serif']=['SimHei']##中文乱码问题!
plt.rcParams['axes.unicode_minus']=False#横坐标负号显示问题!
plt.figure(figsize=(15, 8))
data = pd.read_excel('redwine.xlsx',index_col=0)
winenames = np.array(list(data))
def makefigure(arr1,arr2,c='b'):
plt.figure(figsize=(15, 8))
sorted_idx = np.argsort(arr1)
barpos = np.arange(sorted_idx.shape[0]) + 0.5
plt.barh(barpos, arr2[sorted_idx], align='center', color=c)
plt.yticks(barpos, winenames[sorted_idx], fontsize=18)
plt.xlabel('CV value', fontsize=18)
plt.tick_params(labelsize=20, rotation=10)
plt.show()
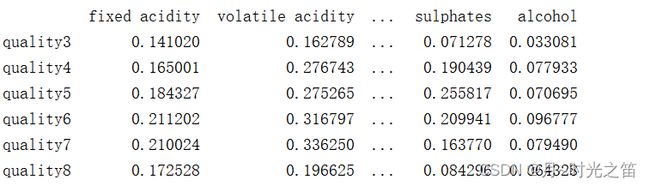
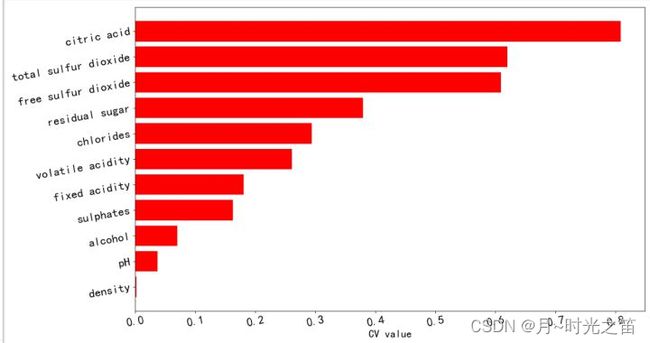
1:CV相关代码
quality_ls = list(set(data['quality']))
cols = list(data.iloc[:,:-1])
mean_ls = []
std_ls = []
nums = 2
for i in cols:
m_ls = []
s_ls = []
for j in quality_ls:
data_temp = data[data.quality==j][i]
ls_sort = sorted(list(data_temp),reverse=True)
ls_sort = ls_sort[nums:-nums]##去除前后极端值
mean_res = np.array(ls_sort).mean() + 1e-8#防止分母为0
std_res = np.array(ls_sort).std()
m_ls.append(mean_res)
s_ls.append(std_res)
mean_ls.append(m_ls)
std_ls.append(s_ls)
mean_df = pd.DataFrame(mean_ls,index=cols,columns=['quality' + str(i) for i in quality_ls]).T
std_df = pd.DataFrame(std_ls,index=cols,columns=['quality' + str(i) for i in quality_ls]).T
CV_df = std_df / mean_df
print(CV_df)
CV_df_mean = CV_df.mean()
CV_fin = np.array(list(CV_df_mean))
res1 = makefigure(CV_df_mean,CV_fin,'r')
2:倍数计算相关代码
winename_ls = []
mul1 = 1.25
mul2 = 1.15
for i in cols:
ls_means = []
for j in quality_ls:
data_temp = data[data.quality == j][i]
mean_v = data_temp.mean()
ls_means.append(mean_v)
###定义筛选逻辑
meant_ls = sorted(ls_means,reverse=True)
Max_v = max(ls_means)
Min_v = min(ls_means)
loc_Mn = quality_ls[ls_means.index(Max_v)]##最大值对应的数据标签位置
Max_sec = meant_ls[1]##第二大值
if Max_sec != 0:
if Max_v / Max_sec >= mul1 and Max_v / Min_v >= mul2:
D = dict(zip(quality_ls,ls_means))
print('\033[1;32m符合要求的特征是:%s,在标签%s中,数值:%s,各类表现:%s\033[0m'%(i,loc_Mn,Max_v,D))
winename_ls.append(i)
print('\033[1;34m最终满足上述要求的共%s个特征,分别是:%s\033[0m'%(len(set(winename_ls)),set(winename_ls)))
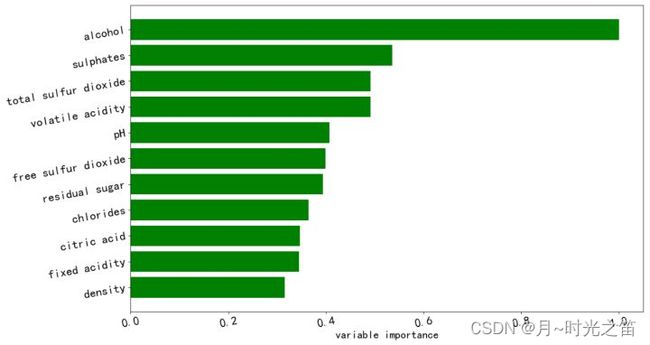
3:随机森林相关代码
x = data.iloc[:,:-1]
y = data.iloc[:,-1]
itrees = 180
depth = None
maxfeat = 11
#如果是回归,用RandomForestRegressor方法
winerfm = ensemble.RandomForestRegressor(n_estimators=itrees,max_depth=depth,max_features=maxfeat,oob_score=False,random_state=521)
####n_estimators:小会欠拟合,大会过拟合,大到一定层度提升不会太大,选个100左右;oob_score:是否采用袋外评估模型好坏
###min_samples_leaf:如果样本量不大,不需要管这个值。如果样本量数量级非常大,则推荐增大这个值
##决策树最大深度max_depth: 默认可以不输入,如果不输入的话,决策树在建立子树的时候不会限制子树的深度。一般来说,数据少或者特征少的时候可以不管这个值。如果模型样本量多,特征也多的情况下,推荐限制这个最大深度。常用的可以取值10-100之间。
##max_features有以下几种选取方法:"auto", "sqrt", "log2", None。auto与sqrt都是取特征总数的开方,log2取特征总数的对数,None则是令max_features直接等于特征总数,而max_features的默认值是"auto"。
winerfm.fit(x,y)
featureimportance = winerfm.feature_importances_
featureimportance = featureimportance / featureimportance.max()
res2 = makefigure(featureimportance,featureimportance)
4:Xgboost和Lasso相关代码
plt.figure(figsize=(15, 8))
params={'max_depth':11,
'n_estimators':100,
'learning_rate':0.1,
'nthread':4,
'subsample':1.0,
'colsample_bytree':0.5,
'min_child_weight' : 3,
'seed':361}
###XGBRegressor做回归预测
selectmodel = XGBClassifier(params)
bst = selectmodel.fit(x, y)
featureimportance = bst.feature_importances_
featureimportance = featureimportance / featureimportance.max()
sorted_idx = np.argsort(featureimportance)
sort_id = np.argsort(-featureimportance)
res3 = makefigure(featureimportance,featureimportance,'g')
####################
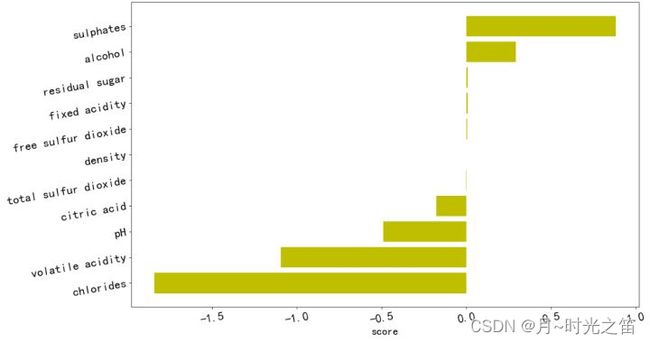
Lass_model = Lasso(alpha=0.0001,tol=1e-5)
Lass_model.fit(x,y) #用筛选后的特征数据来训练模型
co = Lass_model.coef_
res4 = makefigure(co,co,'y')
注:以上学习模型基于回归方法同样能得到特征贡献值!
四:递归消除法
给定一个为特征分配权重的外部估计器(例如,线性模型的系数),递归特征消除(RFE)的目标是通过递归地考虑越来越小的特征集来选择特征。首先,估计器在初始特征集上进行训练,每个特征的重要性通过任何特定属性或可调用获得。然后,从当前的特征集中剪除最不重要的特征。该过程在修剪后的集合上递归重复,直到最终达到要选择的所需特征数量,这种方法原理性比较简单,简单有效但运算量大。
我们可以利用机器学习库:
from sklearn.ensemble import RandomForestClassifier as RFC
官方链接点击即可
五:总结
特征提取和特征筛选是有很大区别的,特征提取之后,原数据集可以说变得“面目全非”,特征筛选可以说是原数据集的一个子集,但两则的目的可以说是殊途同归的,都是为了降维,减少过拟合现象,减少运算复杂度。
但是每种检验方法我们务必要理解其底层含义,才能做到游刃有余,才能事半功倍,否则一味地堆积方法是不可取的,是很难解决实际问题的。