Seurat分析单细胞标准流程
目录
一、数据下载
二、Setup the Seurat Object(设置Seurat对象)
1、安装R包
2、加载R包
3、读入数据
三、Standard pre-processing workflow(标准的预处理流程)
1、QC and selecting cells for further analysis(QC和细胞筛选)
常规质控标准
可视化QC指标
数据标准化
高变基因鉴定
数据归一化
2、线性降维
3、确定数据集的“维度”
JackStrawPlot
ElbowPlot
四、Cluster the cells(细胞聚类)
1、参数调整
2、非线性降维(UMAP/tSNE)
3、寻找差异基因
4、Marker gene可视化
VlnPlot
FeaturePlot
DoHeatmap
五、Assigning cell type identity to clusters(细胞类型鉴定)
1、鉴定方法
2、可视化
六、可视化方法拓展
1、RidgePlot
编辑
2、VlnPlot
3、FeaturePlot
4、DotPlot
5、DoHeatmap
6、FeaturePlot(样本间比较)
7、VlnPlot(样本间比较)
8、DotPlot(样本间比较)
一、数据下载
我们将分析从10X Genomics免费提供的外周血单个核细胞(PBMC)数据集。在Illumina NextSeq 500上测序了2700个单细胞。原始数据可使用下方链接下载:
https://cf.10xgenomics.com/samples/cell/pbmc3k/pbmc3k_filtered_gene_bc_matrices.tar.gz
二、Setup the Seurat Object(设置Seurat对象)
1、安装R包
install.packages("Seurat")
install.packages("dplyr")
install.packages("patchwork")
2、加载R包
library(dplyr)
library(Seurat)
library(patchwork)3、读入数据
# Load the PBMC dataset(导入数据)
pbmc.data <- Read10X(data.dir = "../data/pbmc3k/filtered_gene_bc_matrices/hg19/")
# Initialize the Seurat object with the raw (non-normalized data)(创建Seurat对象).
pbmc <- CreateSeuratObject(counts = pbmc.data, project = "pbmc3k", min.cells = 3, min.features = 200)
#过滤检测少于200个基因的细胞(min.features = 200)和少于3个细胞检测出的基因(min.cells = 3)
pbmc#注释
pbmc <- CreateSeuratObject(counts = pbmc.data, project = "pbmc3k", min.cells = 3, min.features = 200)
counts:#未标准化的数据,如原始计数或TPMs
project:#设置Seurat对象的项目名称
min.cells:#包含至少在这些细胞检测到的features。
min.features:#包含至少检测到这些features的细胞
三、Standard pre-processing workflow(标准的预处理流程)
1、QC and selecting cells for further analysis(QC和细胞筛选)
常规质控标准
(1)在每个细胞中检测到的特异基因的数量:低质量的细胞或空液滴通常只有很少的基因,两个或多个细胞被捕获后可能表现出异常高的基因计数。
(2)细胞内检测到的分子总数(与特异基因密切相关)
(3)每个细胞中线粒体基因的占比:低质量细胞会出现异常的线粒体污染,使用PercentageFeatureSet()函数计算线粒体QC指标,同时,使用以MT-开头的所有基因集作为线粒体基因集。
# The [[ operator can add columns to object metadata. This is a great place to stash QC stats
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")可视化QC指标
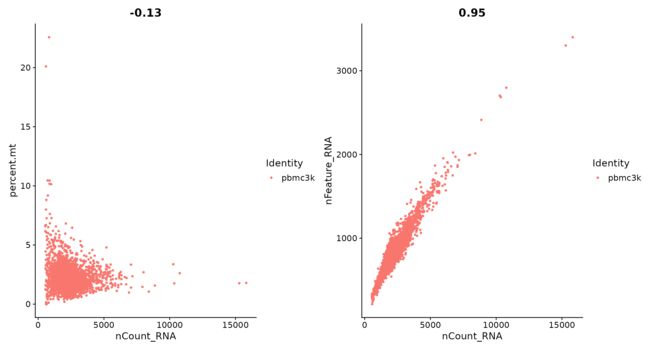
# FeatureScatter is typically used to visualize feature-feature relationships, but can be used
# for anything calculated by the object, i.e. columns in object metadata, PC scores etc.
plot1 <- FeatureScatter(pbmc, feature1 = "nCount_RNA", feature2 = "percent.mt")
plot2 <- FeatureScatter(pbmc, feature1 = "nCount_RNA", feature2 = "nFeature_RNA")
plot1 + plot2pbmc <- subset(pbmc, subset = nFeature_RNA > 200 & nFeature_RNA < 2500 & percent.mt < 5)
#过滤线粒体基因表达占比过高的细胞,和一些极值细胞(可以根据小提琴图判断,查看两端离群值)。数据标准化
从数据集中删除不需要的单元格之后,下一步是规范化数据。默认情况下,我们使用全局缩放归一化方法“LogNormalize”,该方法通过总表达式对每个单元的特征表达式测量进行归一化,将其乘以一个比例因子(默认为10,000),然后对结果进行对数转换。
pbmc <- NormalizeData(pbmc, normalization.method = "LogNormalize", scale.factor = 10000)为了清晰起见,在前面这行代码中(以及在以后的命令中),我们为函数调用中的某些参数提供默认值。然而,这不是必需的,同样的行为可以通过以下方式实现:
pbmc <- NormalizeData(pbmc)高变基因鉴定
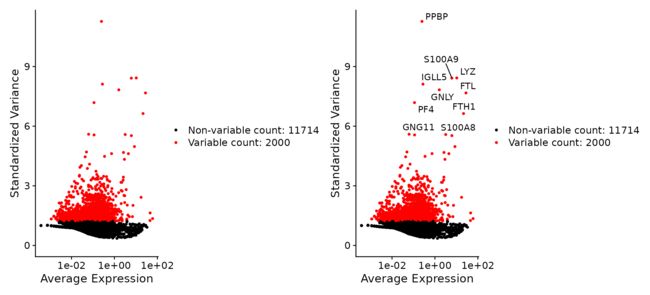
计算在数据集中显示高细胞间变异的特征子集(即,它们在一些细胞中高度表达,在另一些细胞中低表达)。在下游分析中,这些基因有助于突出单细胞数据集中的生物信号。这一过程在FindVariableFeatures()函数中实现。默认情况下,每个数据集选择2000个特征。这些将用于下游分析,如PCA。
pbmc <- FindVariableFeatures(pbmc, selection.method = "vst", nfeatures = 2000)
# Identify the 10 most highly variable genes(显示top10的高变基因,也可根据实际需要调整参数,如top20)
top10 <- head(VariableFeatures(pbmc), 10)
# plot variable features with and without labels
plot1 <- VariableFeaturePlot(pbmc)
plot2 <- LabelPoints(plot = plot1, points = top10, repel = TRUE)
plot1 + plot2数据归一化
这是PCA降维前的一个标准出来步骤,改变每个基因的表达,使细胞间的平均表达为0,缩放每个基因的表达,使细胞间的方差为1(这一步骤在下游分析中具有同等的权重,因此高表达基因不会占主导地位)。
all.genes <- rownames(pbmc)
pbmc <- ScaleData(pbmc, features = all.genes)2、线性降维
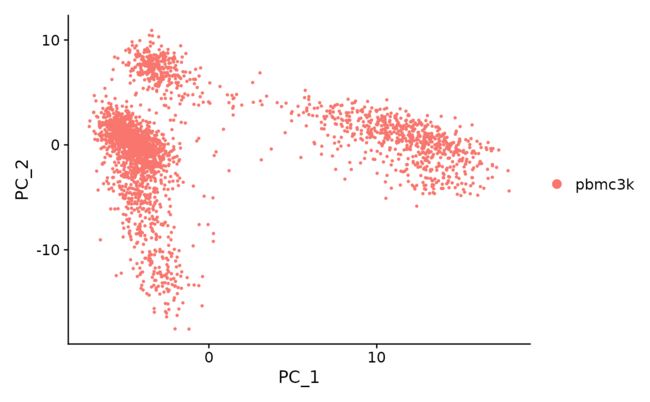
接下来我们对缩放后的数据进行PCA分析。默认情况下,只使用先前确定的变量特征作为输入,但如果希望选择不同的子集,可以使用features参数定义。
pbmc <- RunPCA(pbmc, features = VariableFeatures(object = pbmc))# Examine and visualize PCA results a few different ways
print(pbmc[["pca"]], dims = 1:5, nfeatures = 5)## PC_ 1
## Positive: CST3, TYROBP, LST1, AIF1, FTL
## Negative: MALAT1, LTB, IL32, IL7R, CD2
## PC_ 2
## Positive: CD79A, MS4A1, TCL1A, HLA-DQA1, HLA-DQB1
## Negative: NKG7, PRF1, CST7, GZMB, GZMA
## PC_ 3
## Positive: HLA-DQA1, CD79A, CD79B, HLA-DQB1, HLA-DPB1
## Negative: PPBP, PF4, SDPR, SPARC, GNG11
## PC_ 4
## Positive: HLA-DQA1, CD79B, CD79A, MS4A1, HLA-DQB1
## Negative: VIM, IL7R, S100A6, IL32, S100A8
## PC_ 5
## Positive: GZMB, NKG7, S100A8, FGFBP2, GNLY
## Negative: LTB, IL7R, CKB, VIM, MS4A7可视化PCA结果(几种方式)
VizDimLoadings(pbmc, dims = 1:2, reduction = "pca")DimPlot(pbmc, reduction = "pca")值得注意的是DimHeatmap()允许轻松地探索数据集中的主要异构源,并且在试图决定将哪些pc包含到进一步的下游分析中时非常有用。细胞和特征都是根据它们的PCA分数排序的。这显然是一个有监督的分析,但这是一个有价值的工具来探索相关的特征集。
DimHeatmap(pbmc, dims = 1, cells = 500, balanced = TRUE)DimHeatmap(pbmc, dims = 1:15, cells = 500, balanced = TRUE)
#dims的参数值可以根据自己的需要调整,比如dims = 1:6,则会输出前6个PCA的热图3、确定数据集的“维度”
为了克服scRNA-seq数据中任何单一特征中广泛的技术噪声,Seurat基于它们的PCA分数对细胞进行聚类,每个PC本质上代表一个“元特征”,该“元特征”结合了相关特征集的信息。但是,我们应该选择包含多少PCA呢?概括来说就是选择包含信息量大的维度(features),去除信息量少的“干扰”维度。可以通过下面两种方式选择:
JackStrawPlot
JackStrawPlot()函数提供了一个可视化工具,用于将每个PC的p值分布与均匀分布(虚线)进行比较。“有意义”的pc将表现出较低p值(虚线上方的实线)特征的强烈富集。在这种情况下,在第一批10-12台pc之后,重要性似乎急剧下降。
# NOTE: This process can take a long time for big datasets, comment out for expediency. More
# approximate techniques such as those implemented in ElbowPlot() can be used to reduce
# computation time
pbmc <- JackStraw(pbmc, num.replicate = 100)
pbmc <- ScoreJackStraw(pbmc, dims = 1:20)JackStrawPlot(pbmc, dims = 1:15)ElbowPlot
另一种选择方法是“肘部图”:基于每个组件解释的方差百分比(ElbowPlot()函数)对主要组件进行排序。在这个例子中,我们可以观察到PC9-10附近的一个“肘”,这表明大多数的真实信号是在前10个pc中捕获的。
ElbowPlot(pbmc)四、Cluster the cells(细胞聚类)
1、参数调整
首先基于PCA空间中的欧氏距离构造一个KNN图,并基于其局部邻域的共享重叠(Jaccard相似度)细化任意两个细胞之间的边缘权重。这一步是使用FindNeighbors()函数执行的,并将先前定义的数据集维度(前10个pc)作为输入。
FindClusters()函数实现了这个过程,并包含一个解析参数,该参数设置下游集群的“粒度”,值越大,集群数量就越多。我们发现,对于大约3K细胞的单细胞数据集,将该参数设置在0.4-1.2之间通常会返回良好的结果。对于较大的数据集,最佳分辨率通常会增加。可以使用Idents()函数找到集群。
pbmc <- FindNeighbors(pbmc, dims = 1:10)
pbmc <- FindClusters(pbmc, resolution = 0.5)
#####最重要的知识点:dims = 1:10和resolution = 0.5可以调整,值越大,聚类越多,根据实际需要不断调整参数,即可得到自己想要的细胞聚类数。## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck
##
## Number of nodes: 2638
## Number of edges: 95927
##
## Running Louvain algorithm...
## Maximum modularity in 10 random starts: 0.8728
## Number of communities: 9(9即为得到9个聚类吗,随着resolution的数值变化,聚类数也随着变化)
## Elapsed time: 0 seconds
#可在这一步不断调整,得到自己想要的聚类数后再进行后续分析# Look at cluster IDs of the first 5 cells
head(Idents(pbmc), 5)## AAACATACAACCAC-1 AAACATTGAGCTAC-1 AAACATTGATCAGC-1 AAACCGTGCTTCCG-1
## 2 3 2 1
## AAACCGTGTATGCG-1
## 6
## Levels: 0 1 2 3 4 5 6 7 82、非线性降维(UMAP/tSNE)
Seurat提供了几种非线性降维技术,如tSNE和UMAP,以可视化和探索这些数据集。
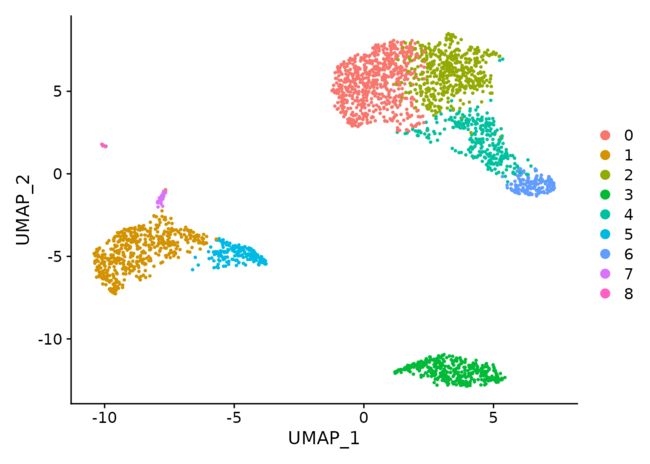
# 若要使用UMAP聚类
pbmc <- RunUMAP(pbmc, dims = 1:10)
DimPlot(pbmc, reduction = "umap")
若要显示标签,可加参数如下
DimPlot(pbmc, reduction = "umap", label = TRUE)
# 若要使用t-SNE聚类
pbmc <- RunTSNE(pbmc, dims = 1:10)
DimPlot(pbmc, reduction = "tsne")
# 显示聚类标签可输入如下参数
DimPlot(pbmc, reduction = "tsne", label = TRUE)保存数据,用语后续分析
saveRDS(pbmc, file = "../output/pbmc_tutorial.rds")
#若无法保存,考虑是否是电脑环境变量配置有误,搜索相关资料进行R环境变量配置即可解决3、寻找差异基因
Seurat可以帮助找到通过差异表达式定义集群的标记。默认情况下,与所有其他单元相比,它识别单个集群(在identity .1中指定)的阳性和阴性标记。FindAllMarkers()为所有集群自动执行此过程,也可以测试集群组之间的对比,或测试所有单元格。每个聚类的差异基因可用于后续该细胞的标记基因。
# find all markers of cluster 0(寻找cluster0的基因)
cluster0.markers <- FindMarkers(pbmc, ident.1 = 0, min.pct = 0.25)
head(cluster0.markers, n = 5)
#可以根据需要,将每个聚类的差异基因都找一遍,方便后续分析
#head(cluster0.markers, n = 5),可以将n = 5进行调整,比如换成10,即可显示10个差异基因
###参数解释
#dent.1 设置待分析的细胞类别
#min.pct 在两组细胞中的任何一组中检测到的最小百分
#thresh.test 在两组细胞间以一定数量的差异表达(平均)
#max.cells.per.ident 通过降低每个类的采样值,提高计算速度## p_val avg_log2FC pct.1 pct.2 p_val_adj
## IL32 2.892340e-90 1.2013522 0.947 0.465 3.966555e-86
## LTB 1.060121e-86 1.2695776 0.981 0.643 1.453850e-82
## CD3D 8.794641e-71 0.9389621 0.922 0.432 1.206097e-66
## IL7R 3.516098e-68 1.1873213 0.750 0.326 4.821977e-64
## LDHB 1.642480e-67 0.8969774 0.954 0.614 2.252497e-63# find all markers distinguishing cluster 5 from clusters 0 and 3
cluster5.markers <- FindMarkers(pbmc, ident.1 = 5, ident.2 = c(0, 3), min.pct = 0.25)
head(cluster5.markers, n = 5)
###找出区分cluster 5与cluster 0和cluster 3的所有标记,可根据自己需要调整重点来了
# find markers for every cluster compared to all remaining cells, report only the positive
# ones
pbmc.markers <- FindAllMarkers(pbmc, only.pos = TRUE, min.pct = 0.25, logfc.threshold = 0.25)
pbmc.markers %>%
group_by(cluster) %>%
slice_max(n = 2, order_by = avg_log2FC)
## 找出每个cluster的标记与所有剩余的细胞相比较,只报告阳性细胞
#这一步特别重要,可以在后续可视化时将尽可能的调出差异基因,可以是后续细胞类型鉴定结果更加准确## # A tibble: 18 × 7
## # Groups: cluster [9]
## p_val avg_log2FC pct.1 pct.2 p_val_adj cluster gene
##
## 1 9.57e- 88 1.36 0.447 0.108 1.31e- 83 0 CCR7
## 2 3.75e-112 1.09 0.912 0.592 5.14e-108 0 LDHB
## 3 0 5.57 0.996 0.215 0 1 S100A9
## 4 0 5.48 0.975 0.121 0 1 S100A8
## 5 1.06e- 86 1.27 0.981 0.643 1.45e- 82 2 LTB
## 6 2.97e- 58 1.23 0.42 0.111 4.07e- 54 2 AQP3
## 7 0 4.31 0.936 0.041 0 3 CD79A
## 8 9.48e-271 3.59 0.622 0.022 1.30e-266 3 TCL1A
## 9 5.61e-202 3.10 0.983 0.234 7.70e-198 4 CCL5
## 10 7.25e-165 3.00 0.577 0.055 9.95e-161 4 GZMK
## 11 3.51e-184 3.31 0.975 0.134 4.82e-180 5 FCGR3A
## 12 2.03e-125 3.09 1 0.315 2.78e-121 5 LST1
## 13 3.13e-191 5.32 0.961 0.131 4.30e-187 6 GNLY
## 14 7.95e-269 4.83 0.961 0.068 1.09e-264 6 GZMB
## 15 1.48e-220 3.87 0.812 0.011 2.03e-216 7 FCER1A
## 16 1.67e- 21 2.87 1 0.513 2.28e- 17 7 HLA-DPB1
## 17 1.92e-102 8.59 1 0.024 2.63e- 98 8 PPBP
## 18 9.25e-186 7.29 1 0.011 1.27e-181 8 PF4 4、Marker gene可视化
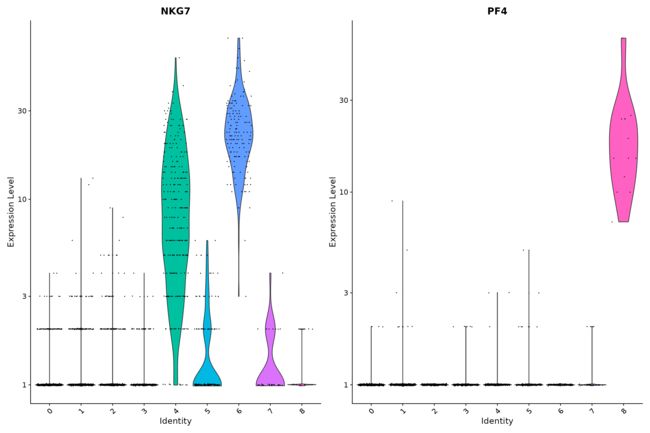
VlnPlot
# you can plot raw counts as well
VlnPlot(pbmc, features = c("NKG7", "PF4"), slot = "counts", log = TRUE)FeaturePlot
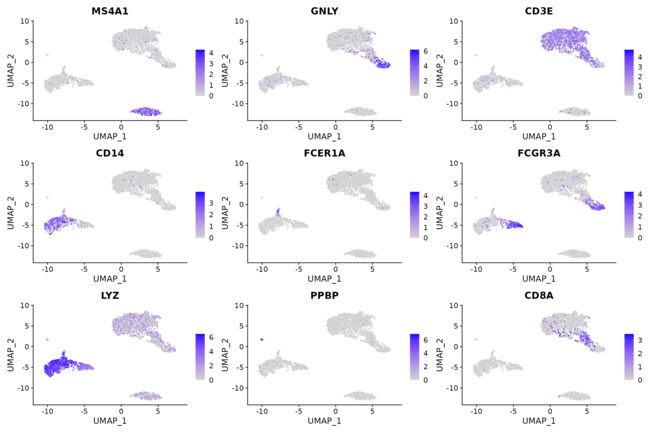
FeaturePlot(pbmc, features = c("MS4A1", "GNLY", "CD3E", "CD14", "FCER1A", "FCGR3A", "LYZ", "PPBP",
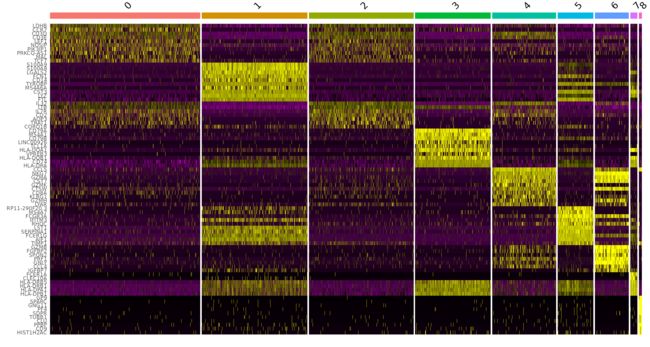
"CD8A"))DoHeatmap
pbmc.markers %>%
group_by(cluster) %>%
top_n(n = 10, wt = avg_log2FC) -> top10
DoHeatmap(pbmc, features = top10$gene) + NoLegend()五、Assigning cell type identity to clusters(细胞类型鉴定)
1、鉴定方法
通过前面的分析,得出了每个聚类的差异基因,对这些基因与已知细胞的标记基因进行比对,更多的是查阅文献,或者参考CellMarker数据库(CellMarker),对细胞类型进行鉴定。这是单细胞分析中最简单,但是最耗时间的一步,需要大量的资料支撑。
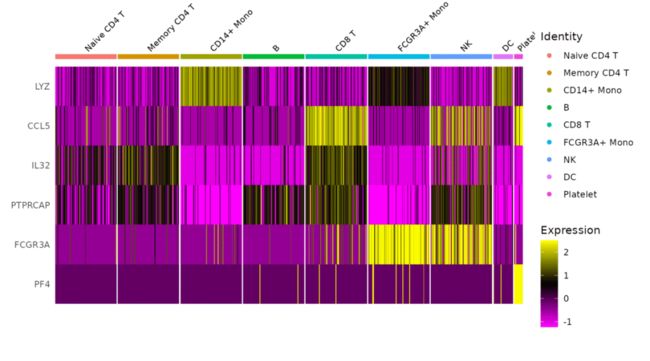
2、可视化
| Cluster ID | Markers | Cell Type |
|---|---|---|
| 0 | IL7R, CCR7 | Naive CD4+ T |
| 1 | CD14, LYZ | CD14+ Mono |
| 2 | IL7R, S100A4 | Memory CD4+ |
| 3 | MS4A1 | B |
| 4 | CD8A | CD8+ T |
| 5 | FCGR3A, MS4A7 | FCGR3A+ Mono |
| 6 | GNLY, NKG7 | NK |
| 7 | FCER1A, CST3 | DC |
| 8 | PPBP | Platelet |
new.cluster.ids <- c("Naive CD4 T", "CD14+ Mono", "Memory CD4 T", "B", "CD8 T", "FCGR3A+ Mono",
"NK", "DC", "Platelet")
names(new.cluster.ids) <- levels(pbmc)
pbmc <- RenameIdents(pbmc, new.cluster.ids)
DimPlot(pbmc, reduction = "umap", label = TRUE, pt.size = 0.5) + NoLegend()
#根据资料查阅,最后确定细胞类型,将细胞类型名称按顺序输入即可命名
#pt.size = 0.5 这是调整可视化图颗粒大小的,如果觉得颗粒太小,可以根据需要调整,如pt.size = 1保存文件
saveRDS(pbmc, file = "../output/pbmc3k_final.rds")六、可视化方法拓展
面对顶级文章的图形内卷,我们需要更多高大上的图形来提高文章档次
1、RidgePlot
RidgePlot(pbmc3k.final, features = features, ncol = 2)
#ncol = 2 调整组间聚类的,如果组太多,可以调大数值,显的敞亮
2、VlnPlot
VlnPlot(pbmc3k.final, features = features)
3、FeaturePlot
FeaturePlot(pbmc3k.final, features = features)4、DotPlot
DotPlot(pbmc3k.final, features = features) + RotatedAxis()5、DoHeatmap
DoHeatmap(subset(pbmc3k.final, downsample = 100), features = features, size = 3)6、FeaturePlot(样本间比较)
FeaturePlot(pbmc3k.final, features = c("MS4A1", "CD79A"), split.by = "groups")7、VlnPlot(样本间比较)
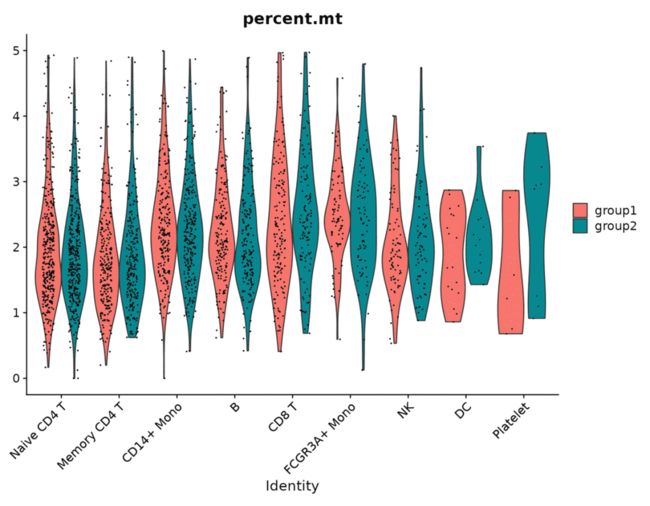
VlnPlot(pbmc3k.final, features = "percent.mt", split.by = "groups")8、DotPlot(样本间比较)
DotPlot(pbmc3k.final, features = features, split.by = "groups") + RotatedAxis()