cox回归RCS阈值效应函数cut.tab1.3发布
写在前面的话,本次只发布了cox回归RCS阈值函数,请看清楚再购买,觉得贵的可以等一等,这个函数最终会放在ggrcs包上面,免费供大家使用,急用的可以先看看。
接下来聊聊cox回归RCS阈值函数是干什么用的,随便抓一篇论文给大家看看,粉丝发给我的。
L-shaped association of serum 25-hydroxyvitamin D concentrations
with cardiovascular and all-cause mortality in individuals with osteoarthritis: results
from the NHANES database prospective cohort study

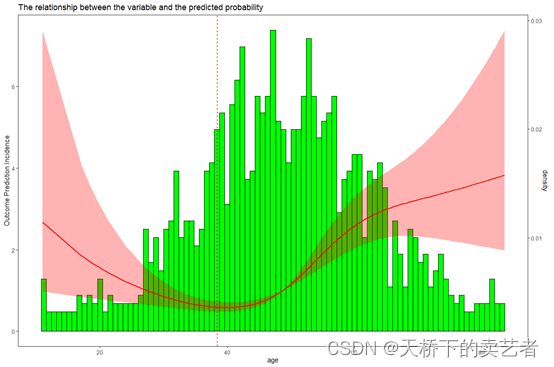
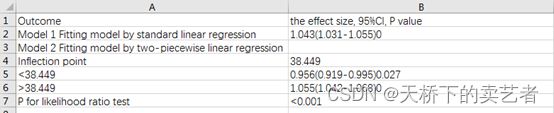
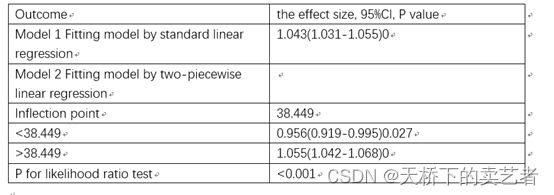
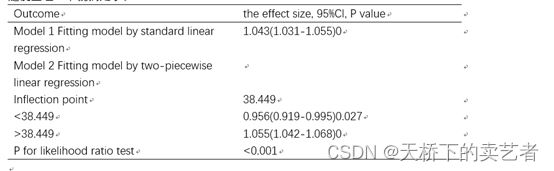
我们看到它曲线拟合后直接就是一个表
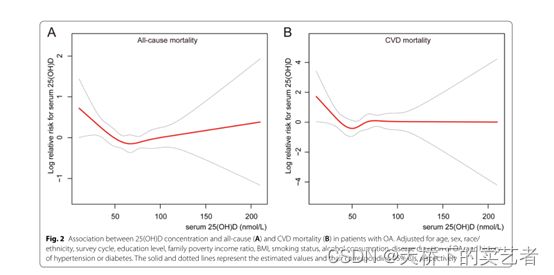
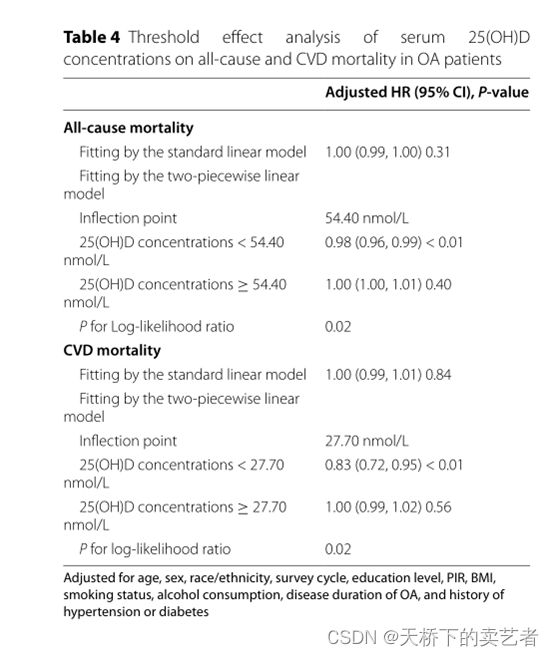
列出了25(OH)D在54.4就是转弯处前对结局影响的HR,还有在转弯后对结局影响的HR。今天我们通过ggrcs包做出这样一个图和自写的函数cut.tab1.3来做出这样一个表,为什么叫cut.tab,乱取的,不要在意这种小细节,我也没有什么好的文艺细胞,为什么是1.3,因为前面两个版本写废了。我们先导入ggrcs包2.6版本,我们通过它自带的数据来演示
我们先导入R包和数据
library(ggrcs)
library(rms)
library(ggplot2)
library(scales)
dt<-smoke
这是ggrcs包自带的吸烟数据status结局,time时间,age年龄,gender性别,我们先整理数据
dd<-datadist(dt)
options(datadist='dd')
建立模型
fit <- cph(Surv(time,status==1) ~ rcs(age,4), x=TRUE, y=TRUE,data=dt)
绘图
ggrcs(data=dt,fit=fit,x="age")
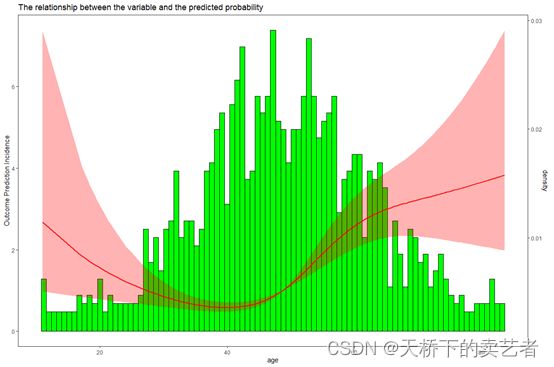
RCS绘图好后我们想了解它的拐点,也就是阈值,因为函数刚写好,还没上下兼容,需要重新建一个一样的模型,但是不是rms包的cph函数,而是survival包的coxph函数。这就里rcs这个函数不需要了。对比一下前后两个函数有什么不同。
fit1 <-coxph(Surv(time,status==1) ~ age,data=dt)
然后cut.tab函数登场了,它有几个配套的小函数,一共4个函数需要全部加载,加载后如图所示
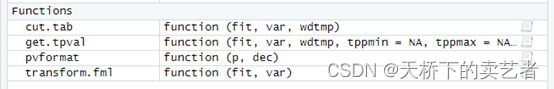
我们只需要用到cut.tab,它的格式是cut.tab(fit,var,wdtmp),我们需要填入3个变量,fit就是你的模型,var就是你研究的变量,wdtmp就是我们的数据,这个数据一定要是数据框形式,不是的话你使用as.data.frame()强制转换一下。
cut.tab(fit1,"age",dt)
p<-ggrcs(data=dt,fit=fit,x="age")
p+geom_vline(aes(xintercept=38.449),colour="#BB0000", linetype="dashed")
tab<-cut.tab(fit1,"age",dt)
write.csv(tab,file = "1.csv",row.names = F)
方法学和解释部分自己看看开头的参考文献把,我这里就不废话了。不过他用的是广义可加模型,我们这里用的是rcs,广义可加模型后面再写,原理都是一样的。
后面附函数代码