HM-GNN:基于分子-基序异构图神经网络的分子表示学习模型
今天给大家介绍一篇由爱荷华州立大学的Zhaoning Yu 与Hongyang Gao发表在ICML(International Conference on Machine Learning)上的文章。作者在该论文中考虑学习分子的基序级特征表示以改善分子整体的特征学习,基于此搭建了分子-基序异构图神经网络模型(HM-GNN),并在多个数据集上与其他模型比较取得了最优的表现,另外作者提出可以将此方法作为小分子数据集多任务学习的一种范式。
1.摘要
本文考虑分子图的特征表示学习问题。目前,基于图神经网络成为分子图的特征表示学习的普遍方法。现有的分子图神经网络学习方法主要考虑聚合更新分子的原子级信息,再提取表示为整个分子图的特征。这些方法在一定程度上都忽略了分子中子结构的信息,比如分子中的基序。本文据此提出构造一个包含基序和分子两种节点的异构基序图,基序节点对应于从分子中提取的基序。在构造的异构基序图上学习分子基序结构的特征信息。为此,本文提出了一种异构图神经网络(HM-GNN),来学习异构模图中每个节点的特征表示。
另外,本文考虑基于异构基序图可以根据基序词汇连接不同的小分子数据集,从而实现有效的多任务学习。然后,在模型训练过程中,考虑到图神经网络的学习会随图节点和边的规模增大产生计算资源紧张的问题,本文设计了一种边缘采样策略在尽可能保留图中信息的条件下来缩小图的规模。实验结果表明,本文提出的模型在多个数据集上优于以前最先进的模型。在组合数据集上实验的良好结果证明,本文的方法可以作为一种小分子数据集的多任务学习范式。另外,实验还表明边缘采样策略可以显著减少的计算资源,并尽可能保持模型原本的性能。
2.研究背景
图神经网络(GNN)已被证明能有效地解决图嵌入中的各种挑战性任务,比如节点分类、图分类和链接预测,这些方法已广泛应用于社交网络、分子属性预测、自然语言处理等领域。
在分子属性预测任务中,大多数现有的GNN使用基本的分子图拓扑,通过邻域特征聚合和池化方法获得结构信息。现有很多方法没有考虑分子图之间的联系,特别是分子图中的基序模式的共享。
Motif是一段典型的序列或者一个结构,指构成任何一种特征序列的基本结构,一般来说称为基序或者模体。在生物学上,基序这种有特征的短序列,被认为是拥有生物学功能的保守序列,可能包含特异性的结合位点,或者是涉及某一个特定生物学过程的有共性的序列区段。比如蛋白质序列特异性结合位点,如核酸酶和转录因子等。
本文认为分子图与其他图结构比如社交网络图和引用图的一个显著区别就是Motif,Motif可以看作分子图中具有特定含义的子图。例如,分子图中的代表化学键的边,代表分子环结构的环。而且之前研究证明,作为基序的碳环和NO2基团往往具有致突变性,说明基序可以在一定程度上反映分子的特征。因此,本文提出构建一个基序异构图,并通过HM-GMM模型去学习分子基序上的特征来改善分子的表示。
3.模型方法
本文模型学习进行的工作可以分为提取基序词汇、构造分子-基序异构图、搭建基序异构图神经网络、基于异构基序图进行多任务学习和边缘采样策略等部分。
3.1 提取基序
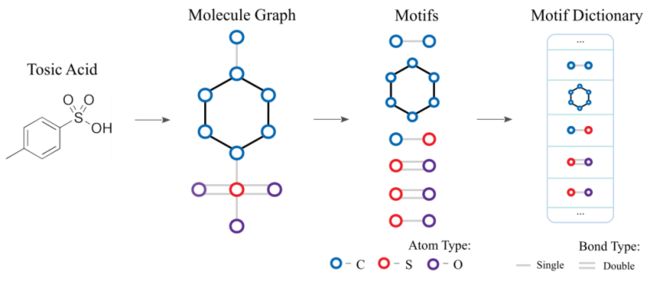
在这项工作中,基序提取包括分子图中的化学键和环结构,并删去重复的词汇。如图1所示,以甲苯磺酸分子为例,首先从其原子图中提取六个键和环。删除重复部分后,在词汇表中添加五个基序。特别地,为了去除基序词典中的噪声词汇,本文将包含基序的分子的TF-IDF(词频-逆文档频率)平均为基序的TF-IDF值。通过TF-IDF对词汇进行排序,将最基本的基序保留为最终词汇。其中,词频表示为分子中基序的频率,逆文档频率指包含基序的分子数量,TF-IDF值为两者的乘积。
图1.基序提取示例图
3.2 构造分子-基序异构图
分子-基序异构图中包括两种节点和两种边,如图2所示。其中,节点分为分子节点和基序节点。边分为以下两种:分子-基序边:如果分子包含基序,将分子节点与基序节点连接。权重使用TF-IDF计算。基序-基序边:如果两个基序节点在分子中共享至少一个原子,将两个节点相连。权重为两个基序的PIM(点互信息),点互信息是一种常用的相关性度量方法,本文中用来比较基序之间的相似性,计算使用分子同时包含两个基序的概率,除以分子分别只包含两个基序的概率。
图2.分子-基序异构图
3.3搭建基序异构图神经网络
本阶段,作者构建了一个HM-GNN来分别学习原子级和模体级的图形特征表示。首先,基于异构图可以应用异构图神经网络学习图中每个分子的模体级特征嵌入。另外,原始分子图拓扑和节点特征包含原子级图形信息,模型使用另一个图神经网络来学习原子级特征嵌入。最终,将两个图神经网络的特征嵌入连接起来,并将其送入多层感知器(MLP)进行预测。HM-GNN模型的示例如图3所示。
图3.HM-GNN模型示例
3.4 基于异构基序图进行多任务学习
大多数分子数据集相对较小,使用图神经网络时很容易发生过拟合。多任务学习能有效降低过拟合并提高模型泛化能力。然而,由于不同数据集间缺乏明确的联系,很难直接使用多任务学习。异构基序图的构建可以很方便地连接一组分子数据,从而进行多任务学习以提升模型性能。
给定分子的数据集,首先从分子数据集中构建基序词典,其中基序只需要在部分数据集中共享。然后构建异构基序图,使用HM-GNN学习每个分子的特征表示输入到单独的MLP中进行预测。在这个过程中,基序节点被认为是连接不同数据集的分子的连接器。
3.5 边缘采样策略
大多数GNN遵循邻域聚合学习方案,时间复杂度和边数密切相关,因此作者提出可以通过边采样方法来减少异构基序图边的数量。边缘采样的示例如图4所示。
采样策略如下:
1.随机选择一个分子节点作为起始节点,进行广度优先搜索,每层随机采样固定数量的边。
2. 第一跳以分子节点为起点,选择分子-基序边。
3.从第二跳开始,只对基序-基序边进行采样,以尽可能多地保留基序信息。
图4.边缘采样示例
4.验证实验
模型验证实验分为七个实验:
4.1 分子图数据集上的性能研究
作者根据TUDataset中的五个流行生物信息学图基准数据集来评估模型,其中包括四个分子数据集和一个蛋白质数据集。结果如表1所示,表明由基序词汇构建的基序节点可以帮助GNN更好地学习分子图的基序级特征表示。在蛋白质数据集上模型也表现得最好,表明蛋白质分子中的基序也包含有用的结构信息。
表1.分子图数据集上性能比较
4.2 大规模数据集上的性能研究
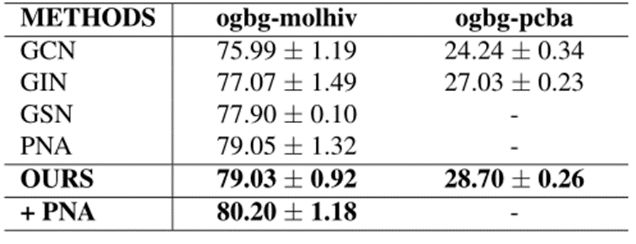
本文为了评估在大规模数据集上的性能,使用了来自开放图基准(OGB)的两个生物信息学数据集:ogbg-molhiv和ogbg-molpcba。表2中显示了Ogbg-molhiv数据集的ROC-AUC结果和在Ogbg-molpcba数据集上的AP结果。可以看出,本文提出的方法明显优于GIN、GCN、GSN和PNA。结果表明,该模型在大规模数据集上具有优越的泛化能力。
表2.大规模数据集上性能比较
4.3 异构基序图消融实验
为了证明基序特征学习在HM-GNN中的有效性,实验中将异构图和相应的GNN从HM-GNNs中移除,模型退化到GIN。然后在三个数据集上比较了HM-GNN和GIN的效果。表3结果显示,本文的模型在PTC、MUTAG和PROTEINS数据集上分别显著优于GIN 14.2%、6.9%和3.7%。
表3.异构基序图消融实验结果
4.4 基序-基序相互作用的消融实验
实验从异构图中删除所有基序-基序边来创建异构基序图的变体,并比较了在变体上和原模型上的结果。如表4所示,对于删除基序-基序边的异构图模型,模型在三个数据集的性能分别下降2.4%、1.7%和1.6%。这表明基序之间的相互作用对分子基序级特征学习很重要。
表4.基序-基序相互作用消融实验结果
4.5 小型分子数据集的多任务学习
PTC数据集根据致癌性标记的化合物啮齿动物分为雄性小鼠(MM)、雄性大鼠(MR)、雌性小鼠(FM)、雌性大鼠(FR)。这里,PTC(PTC-MR)和PTC-FR都是大鼠数据集,PTC-MM是小鼠数据集。通过将PTC与PTC-MM和PTC- FR分别结合,可以创建另外两个数据集:PTC+PTC-MM以及PTC+PTC-FR。重叠比率表明数据集共同拥有的分子基序比例。PTC数据集与PTC-FR和PTC-MM的基序重叠率分别为94.8%和83.5%。
本实验分别构建了单独的异质基序图,并在其上评估了HM-GNN模型的性能。在这里,对三种数据划分比例设置进行评估:90%、50%和10%用于训练,10%、50%和90%用于测试。在较小的训练数据集上训练的模型具有较高的过拟合风险,这可以更好地揭示对性能的影响。
ROC-AUC结果如表5所示,结果表明将PTC与PTC-MM和PTC-FR相结合可以在三种设置中持续带来性能改进。与使用PTC-MM相比,结合PTC FR可以带来更大的性能提升。这是因为PTC与PTC FR有更大的基序词汇重叠。因此,将具有相似的基序词汇量的数据集相结合将有利于小分子数据集上的多任务学习。
表5.多任务学习实验结果
4.6 计算效率研究
本部分对边缘采样方法中的超参数起始采样节点数量进行实验探究,起始节点的数量会影响训练效率和模型性能。在Ogbg-moliv数据集上进行实验探究起始节点的数量的影响,该数据集包含41127个分子图。
结果如图5所示,蓝线ROC-AUC值显示,当起始节点数从5000个变为15000个时,模型性能显著提高。从15000个开始,ROC-AUC的改进逐渐放缓,直到收敛。红线显示,内存使用量几乎随着起始节点数的增加而线性增加。因此,当选择25000个启动节点时,该模型可以达到最佳的利用效率。此时,模型可以用相对较少的计算资源实现较高性能。
图5.计算效率研究实验结果
4.7 基序词典大小实验
在这一部分中,通过实验来研究不同词汇保留率对模型性能的影响。
从图6中可以看到,开始时模型性能随着词汇保留率的增加而提高。保留率越高,词汇量中的基序就越多,图中的分子可以有更多的连接(基序)与其他分子通信,使得基序级特征传播的效果更好。当保留率大于80%时,模型性能开始下降,表明最后20%基序是有噪声的,会影响模型的泛化和鲁棒性。
基序词典通过TF-IDF值过滤基序词汇中的噪声基序,来提高模型的泛化能力和鲁棒性。
图6.基序词典大小实验结果
5.总结
本文提出了一种新的异构基序图和HM-GNN模型用于分子图表示学习。通过提取分子图中所有基序的基序词汇和构建了一个包含基序节点和分子节点的异构基序图,并使用HM-GNN来学习异构基序图,得到基序级图的嵌入。使用另一个GNN来学习原始图的原子级图嵌入。这两个GNN模型可以分别从原子级和基序级学习图形特征表示。实验结果表明, HM-GNN在图分类任务上可以显著提高性能。此外,多任务学习探究实验表明本文方法在小分子数据集上的有不错的多任务学习结果。
本文创新点总结如下:
1.考虑了分子的基序级特征,构建了一个包含基序节点和分子节点的异构基序图。构建HM-GNN模型来学习异构基序图,得到基序级图的嵌入。
2.异构基序图中通过共同拥有的基序连接两个分子,从而实现分子图之间的信息传递。这个思想在小分子数据集上的多任务学习结果是有希望的,拥有相似基序词典的分子数据集可以实现较好的多任务学习效果。
3.边缘采样方法进行图上的高效计算。图上的消息聚合和更新随图中节点和边的增加,所需计算资源会高度紧张,边采样是此问题的一种良好对应策略。
原文:https://proceedings.mlr.press/v162/yu22a/yu22a.pdf
代码:https://github.com/ZhaoningYu1996/HM-G