Yolov5 中添加Network Slimming剪枝--稀疏训练部分
前言:Network Slimming剪枝过程让如下
1. 稀疏化
2. 剪枝
3. 反复迭代这个过程
![]()
一、稀疏化:
通过Network Slimming 的核心思想是:添加L1正则来约束BN层系数,从而剪掉那些贡献比较小的通道channel
原理如下:BN层的计算是这样的:
上边介绍了,Network Slimming的核心思想是剪掉那些贡献比较小的通道channel,它的做法是从BN层下手。BN层的计算公式如下:
![]()
通过BN层的计算公式可以看出每个channe的Zout的大小和系数γ正相关,因此我们可以拿掉哪些γ-->0的channel,但是由于正则化,我们训练一个网络后,bn层的系数是正态分布的。这样的话,0附近的值则很少,那剪枝的作用就很小了。因此要先给BN层加上L1正则化进行一步稀疏训练(为什么要用L1正则化可以看该博客:l1正则与l2正则的特点是什么,各有什么优势? - 知乎)。
为BN层加入L1正则化后,损失函数公式为:![]()
上面第一项是正常训练的loss函数,第二项是约束对于L1正则化,g(s)=|s|,λ是正则系数,引入L1正则来控制γ, 要把稀疏表达加在γ 上, 得到每个特征的重要性 λ
- 每个通道的特征对应的权重是 γ
- 稀疏表达也是对 γ 来说的, 所以正则化系数 λ 也是针对 γ, 而不是 W
- 稀疏化后, 做γ 值的筛选因此在进行反向传播时候:′=∑′+∑′()=∑′+∑||′=∑′+∑()
那如何把程序加到yolov5呢?
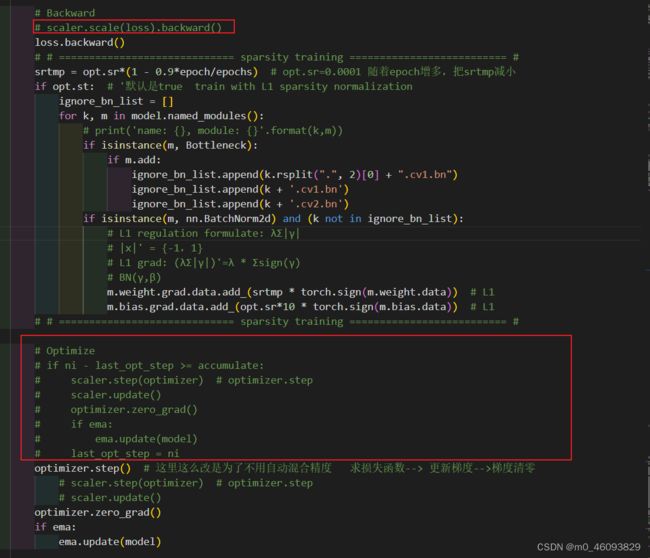
在yolov5 train.py的程序中找到反向传播部分程序:
1.1 稀疏训练核心代码
将scaler.scale(loss).backward()注释,并添加下方代码:
# Backward
# scaler.scale(loss).backward()
loss.backward()
# # ============================= sparsity training ========================== #
srtmp = opt.sr*(1 - 0.9*epoch/epochs) # opt.sr=0.0001 随着epoch增多,把srtmp减小
if opt.st: # '默认是true train with L1 sparsity normalization
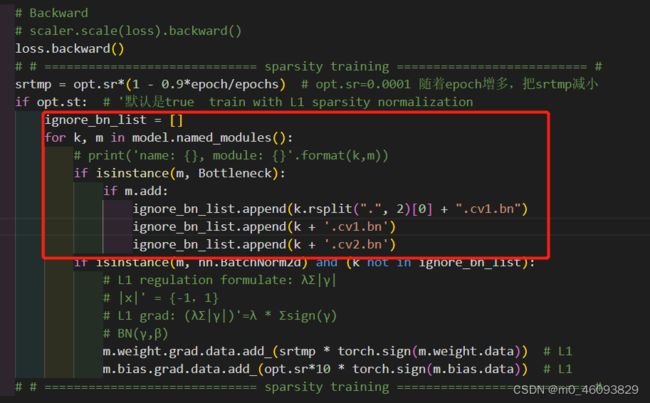
ignore_bn_list = []
for k, m in model.named_modules():
# print('name: {}, module: {}'.format(k,m))
if isinstance(m, Bottleneck):
if m.add:
ignore_bn_list.append(k.rsplit(".", 2)[0] + ".cv1.bn")
ignore_bn_list.append(k + '.cv1.bn')
ignore_bn_list.append(k + '.cv2.bn')
if isinstance(m, nn.BatchNorm2d) and (k not in ignore_bn_list):
# L1 regulation formulate: λΣ|γ|
# |x|' = {-1,1}
# L1 grad: (λΣ|γ|)'=λ * Σsign(γ)
# BN(γ,β)
m.weight.grad.data.add_(srtmp * torch.sign(m.weight.data)) # L1
m.bias.grad.data.add_(opt.sr*10 * torch.sign(m.bias.data)) # L1
# # ============================= sparsity training ==========================
# if ni - last_opt_step >= accumulate:
# scaler.step(optimizer) # optimizer.step
# scaler.update()
# optimizer.zero_grad()
# if ema:
# ema.update(model)
# last_opt_step = ni
optimizer.step()
# scaler.step(optimizer) # optimizer.step
# scaler.update()
optimizer.zero_grad()
if ema:
ema.update(model)
其中sr,st 需要添加参数
parser.add_argument('--st', action='store_true',default=True, help='train with L1 sparsity normalization')
parser.add_argument('--sr', type=float, default=0.0002, help='L1 normal sparse rate')其中需要注意的点1:
红框处程序是因为这里并没有选择所有的bn层进行裁剪,这里选择去除那些有shortcut的Bottleneck层(对应代码中m.add = True),主要是为了保证shortcut和残差层channel一样可以add。
--------------------在这里我曾经有这样的疑问:(该部分可以不看) -----------------------------------------
这两个if 我能理解最终目的是:去除那些有shortcut的Bottleneck层,但是为什么要有 +cv1.bn等等那三步呢?不能直接把k添加到 ignore_bn_list吗?
再说了添加了之后,加入ignore_bn_list的名字就变了呀,此时再运行下一个if的时候k是不在ignore_bn_list
为什么不能改成指令:
if isinstance(m, Bottleneck):
if m.add:
ignore_bn_list.append(k)
if isinstance(m, nn.BatchNorm2d) and (k in ignore_bn_list):
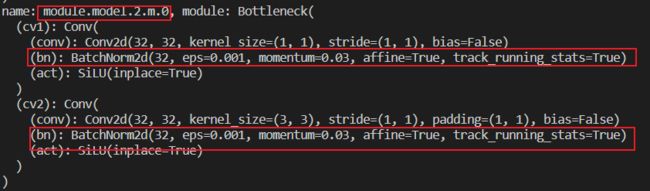
后来我明白了,这里是为了不对Bottlenack中的BatchNorm2d加正则化,因上述改名字的那个步骤其实是找的该Bottleneck下面BatchNorm2d 的名字。比如我断点调试了一下:
其中名为Module.model.2.m.0的模型
其下的BatchNorm2d的名字分别如下:
![]()
![]()
注意的点2:
# Anchors
if not opt.noautoanchor:
check_anchors(dataset, model=model, thr=hyp['anchor_t'], imgsz=imgsz)
# model.half().float()
model.float() # pre-reduce anchor precision# Save model
if (not nosave) or (final_epoch and not evolve): # if save
ckpt = {'epoch': epoch,
'best_fitness': best_fitness,
# 'model': deepcopy(de_parallel(model)).half(),
'model': deepcopy(de_parallel(model)),
# 'ema': deepcopy(ema.ema).half(),
'ema': deepcopy(ema.ema),
'updates': ema.updates,
'optimizer': optimizer.state_dict(),
'wandb_id': loggers.wandb.wandb_run.id if loggers.wandb else None,
'date': datetime.now().isoformat()} if RANK in [-1, 0]:
LOGGER.info(f'\n{epoch - start_epoch + 1} epochs completed in {(time.time() - t0) / 3600:.3f} hours.')
for f in last, best:
if f.exists():
strip_optimizer(f) # strip optimizers
if f is best:
LOGGER.info(f'\nValidating {f}...')
results, _, _ = val.run(data_dict,
batch_size=batch_size // WORLD_SIZE * 2,
imgsz=imgsz,
# model=attempt_load(f, device).half(),
model=attempt_load(f, device),
iou_thres=0.65 if is_coco else 0.60, # best pycocotools results at 0.65
single_cls=single_cls,
dataloader=val_loader,
save_dir=save_dir,
save_json=is_coco,
verbose=True,
plots=True,
callbacks=callbacks,
compute_loss=compute_loss) # val best model with plots以上就将对BN层添加L1正则化的程序加好了,核心思想就是修改反向传播的梯度。
1.2 查看稀疏训练效果
如果想查看系数训练的效果,可加入下方程序:
# =============== show bn weights ===================== #
module_list = []
module_bias_list = []
for i, layer in model.named_modules():
if isinstance(layer, nn.BatchNorm2d) and i not in ignore_bn_list:
bnw = layer.state_dict()['weight']
bnb = layer.state_dict()['bias']
module_list.append(bnw)
module_bias_list.append(bnb)
# bnw = bnw.sort()
# print(f"{i} : {bnw} : ")
size_list = [idx.data.shape[0] for idx in module_list]
bn_weights = torch.zeros(sum(size_list))
bnb_weights = torch.zeros(sum(size_list))
index = 0
for idx, size in enumerate(size_list):
bn_weights[index:(index + size)] = module_list[idx].data.abs().clone()
bnb_weights[index:(index + size)] = module_bias_list[idx].data.abs().clone()
index += size
# print("bn_weights:", torch.sort(bn_weights))
# print("bn_bias:", torch.sort(bnb_weights))
# tb_writer.add_histogram('bn_weights/hist', bn_weights.numpy(), epoch, bins='doane')
# tb_writer.add_histogram('bn_bias/hist', bnb_weights.numpy(), epoch, bins='doane')
loggers.tb.add_histogram('bn_weights/hist', bn_weights.numpy(), epoch, bins='doane')
loggers.tb.add_histogram('bn_bias/hist', bnb_weights.numpy(), epoch, bins='doane')将其加到:一个batch训练结束之后的程序后边就好
我在tensorboard中的图:
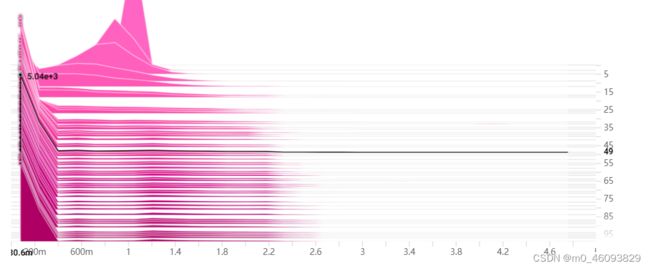 纵轴是epoch,横轴是权重,可以看到我一共进行了100轮稀疏训练,我的项目中非bottoltneck中的bn层加起来参数大概有8000多个,那可以看到在49个epoch的时候,0附近的权重已经有5000多个了。那接下来我可以设置60%的剪枝率,把它们都剪掉
纵轴是epoch,横轴是权重,可以看到我一共进行了100轮稀疏训练,我的项目中非bottoltneck中的bn层加起来参数大概有8000多个,那可以看到在49个epoch的时候,0附近的权重已经有5000多个了。那接下来我可以设置60%的剪枝率,把它们都剪掉