目标分割算法之分水岭算法
分水岭算法
1.经典算法原理及实现
传统的目标分割算法主要分为两种
1.基于像素相似性:阈值分割、k-means分割
2.基于像素邻域关系:区域生长、分水岭、基于标记+分水岭
分水岭算法原理
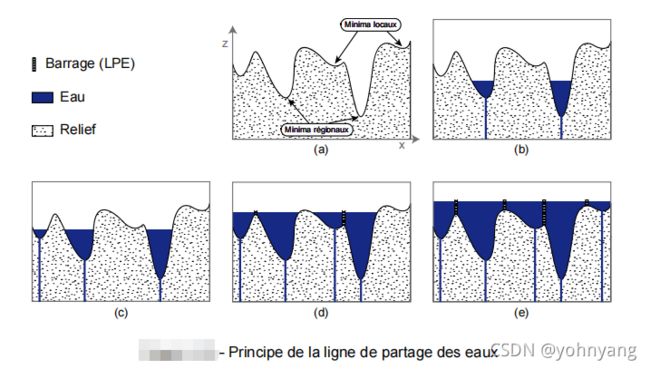
如图中展现了凹凸不平的地貌,视觉上明显的位置有盆地及丘陵,用一维曲线讲对应波峰与波谷,向盆地注水,水会顺这地势先注入地势最低的波谷,然后随着水势升高再注入高一级的波谷,为了保证先注满第一个波谷,需要在右侧波峰出修建大坝,随后依次分水岭注满。
在图像中,地貌对应整个图像的背景,地势对应图像像素点大小。
分水岭算法整个过程:
1.把梯度图像中的所有像素按照灰度值进行分类,并设定一个测地距离阈值。
2.找到灰度值最小的像素点(默认标记为灰度值最低点),让threshold从最小值开始增长,这些点为起始点。
3.水平面在增长的过程中,会碰到周围的邻域像素,测量这些像素到起始点(灰度值最低点)的测地距离,如果小于设定阈值,则将这些像素淹没,否则在这些像素上设置大坝,这样就对这些邻域像素进行了分类。
4.随着水平面越来越高,会设置更多更高的大坝,直到灰度值的最大值,所有区域都在分水岭线上相遇,这些大坝就对整个图像像素的进行了分区。
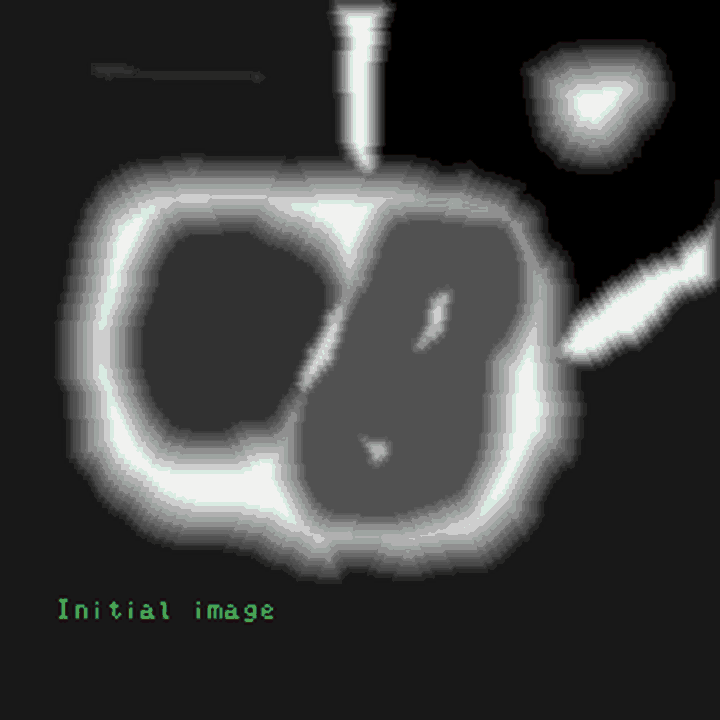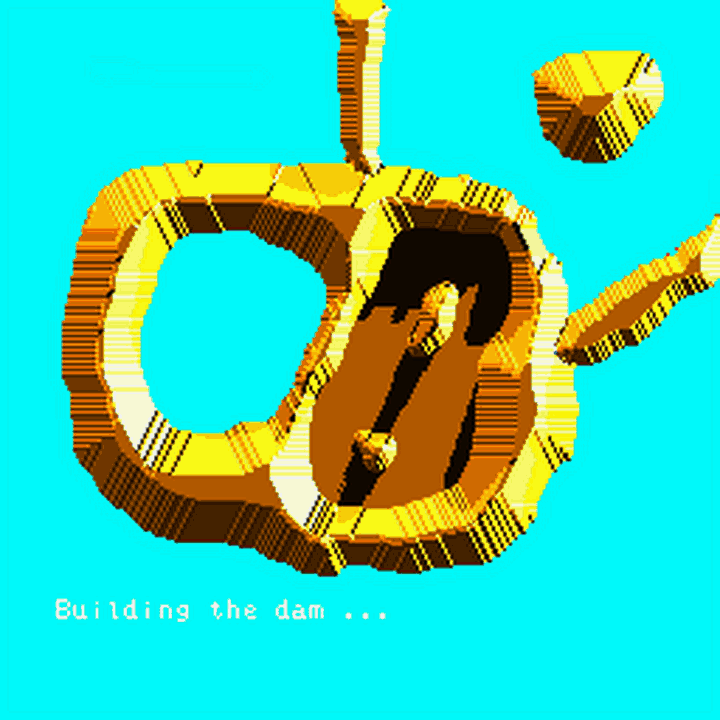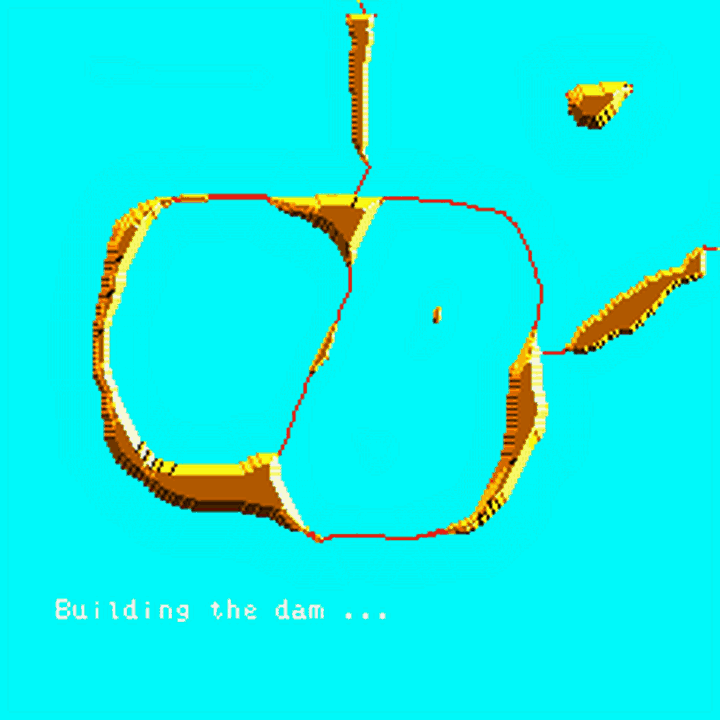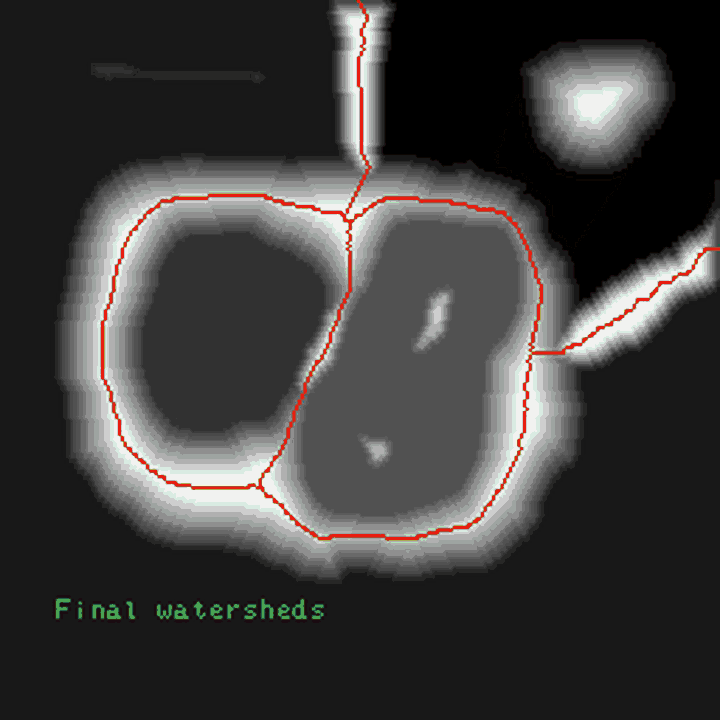
用上面的算法对图像进行分水岭运算,由于噪声点或其它因素的干扰,可能会得到密密麻麻的小区域,即图像被分得太细(over-segmented,过度分割),这因为图像中有非常多的局部极小值点,每个点都会自成一个小区域。
其中的解决方法:
对图像进行高斯平滑操作,抹除很多小的最小值,这些小分区就会合并。
不从最小值开始增长,可以将相对较高的灰度值像素作为起始点(需要用户手动标记),从标记处开始进行淹没,则很多小区域都会被合并为一个区域,这被称为基于图像标记(mark)的分水岭算法。
opencv 函数
void watershed( InputArray image, InputOutputArray markers );
分割流程总结:
step1: 图像灰度化、滤波、Canny边缘检测
step2:查找轮廓,并且把轮廓信息按照不同的编号绘制到watershed的第二个入参merkers上,相当于标记注水点。
step3:watershed分水岭运算
step4:绘制分割出来的区域,视觉控还可以使用随机颜色填充,或者跟原始图像融合以下,以得到更好的显示效果。
/**
*定义一个使用分水岭算法的辅助类
*/
class WatershedSegmenter{
{
private:
Mat markers;
public:
void setMarkers(Mat&markerImage)
{
markerImage.convertTo(markers,CV_32S);
}
Mat process(Mat&image)
{
watershed(image,markers);
markers.convertTo(markers,CV_8U);
return markes;;
}
};
//接受一个参数,显示结果
void watershedSegment (Mat img){
Mat gray(img.rows, img.cols,CV_8UC1);
cvtColor(img, gray, CV_BGR2GRAY); //转换为8-bit,3通道的灰度图
Mat binary = Mat::zeros(gray.rows, gray.cols, CV_8UC1);
adaptiveThreshold(gray, binary, 255, ADAPTIVE_THRESH_GAUSSIAN_C, THRESH_BINARY_INV, 5, 10); //将灰度图转换为二值图
Mat markers = Mat::zeros(gray.rows, gray.cols, CV_8UC1);
//使用findContour()函数找出图像的轮廓
vector<vector<Point> > contours;
vector<Vec4i> hierarchy;
findContours(binary, contours, hierarchy, CV_RETR_LIST, CV_CHAIN_APPROX_NONE);
//将contours结果放入到markers中,便于访问
int idx = 0;
for( ; idx >= 0; idx = hierarchy[idx][0]){
Scalar color(rand()&255, rand()&255, rand()&255);
drawContours(markers, contours, idx, color, CV_FILLED, 8, hierarchy);
}
//调用分水岭算法分割图像
WatershedSegmenter segmenter;
segmenter.setMarkers(markers);
cv::Mat result = segmenter.process(img);
//显示分割结果
namedWindow("segmentation_result", 0);
imshow("segmentation_result", result);
}
2.分水岭算法C++实现
#include 参考:
https://blog.csdn.net/qingyafan/article/details/44260817
https://blog.csdn.net/twowind/article/details/8988282

