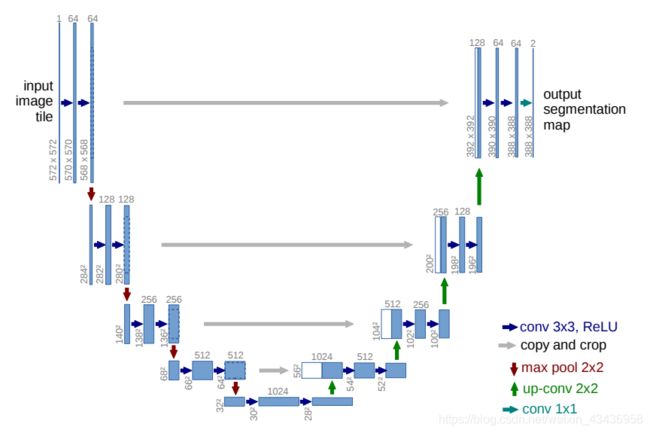
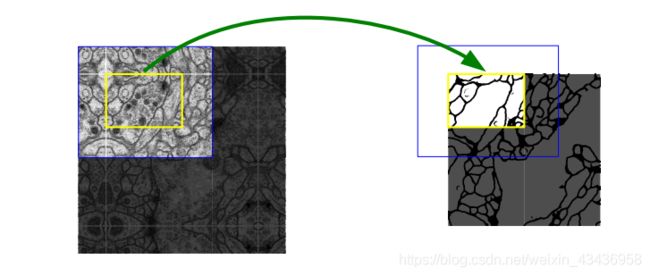
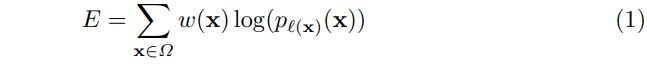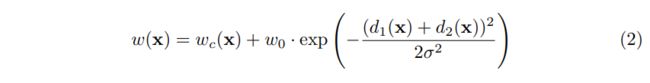- 什么是证书吊销列表?CRL 解释
WoTrusSSL
sslhttps
数字证书是安全在线互动的支柱,用于验证身份和确保加密通信。但是,当这些证书被盗用或滥用时,必须立即撤销它们以维持信任。这就是证书撤销列表(CRL)的作用所在。CRL由证书颁发机构(CA)维护,对于识别和撤销已撤销的证书,防止其造成危害至关重要。在本指南中,我们将探讨什么是CRL、它们如何运作以及为什么它们对网络安全至关重要。什么是证书吊销列表(CRL)?证书吊销列表(CRL)是证书颁发机构(CA)
- k8s:安装 Helm 私有仓库ChartMuseum、helm-push插件并上传、安装Zookeeper
云游
dockerhelmhelm-push
ChartMuseum是Kubernetes生态中用于存储、管理和发布HelmCharts的开源系统,主要用于扩展Helm包管理器的功能核心功能集中存储:提供中央化仓库存储Charts,支持版本管理和权限控制。跨集群部署:支持多集群环境下共享Charts,简化部署流程。离线部署:适配无网络环境,可将Charts存储在本地或局域网内。HTTP接口:通过HTTP协议提供服务,用户
- Android 开源组件和第三方库汇总
gyyzzr
AndroidAndroid开源框架
转载1、github排名https://github.com/trending,github搜索:https://github.com/search2、https://github.com/wasabeef/awesome-android-ui目录UIUI卫星菜单节选器下拉刷新模糊效果HUD与Toast进度条UI其它动画网络相关响应式编程地图数据库图像浏览及处理视频音频处理测试及调试动态更新热更新
- ARM嵌入式可编程控制器技术开发
拉勾科研工作室
arm开发
PLC自动化设计|毕业设计指导|工业自动化解决方案✨专业领域:PLC程序设计与调试工业自动化控制系统HMI人机界面开发工业传感器应用电气控制系统设计工业网络通信擅长工具:西门子S7系列PLC编程三菱/欧姆龙PLC应用触摸屏界面设计电气CAD制图工业现场总线技术自动化设备调试主要内容:PLC控制系统设计工业自动化方案规划电气原理图绘制控制程序编写与调试毕业论文指导毕业设计题目与程序设计✅具体问题可以
- 理解TCP连接中的进程阻塞与CPU调度机制
109702008
编程#C语言网络tcp/ip网络人工智能
引言在计算机网络通信中,TCP连接的建立是一个经典的三次握手过程。当用户调用connect()函数发起连接时,内核会发送SYN报文并等待对方的SYN-ACK响应。此时,调用进程通常会进入阻塞状态,暂停执行直至连接成功或超时。这一机制看似简单,但其背后的内核实现却涉及进程调度、等待队列管理和CPU资源分配等复杂操作。本文将深入探讨阻塞状态的实现原理,并解析CPU在进程阻塞期间的行为。一、进程阻塞的实
- Cesium加载各类数据总结
zhu_zhu_xia
cesiumJavaScriptjavascript
接触到的加载数据类型:源地图、shp、Geojson、png、wms、地形底图一.Cesium加载各类底图#此类加载的本质在于newCesium.ImageryProvider()Apidefination:“Providesimagerytobedisplayedonthesurfaceofanellipsoid.Thistypedescribesaninterfaceandisnotinten
- 如何解决 NPM proxy, 当我们在终端nodejs应用程序时出现代理相关报错
Thisisaproblemrelatedtonetworkconnectivity.npmERR!networkInmostcasesyouarebehindaproxyorhavebadnetworksettings.在使用npminstall下载包的时候总是报以下错误:在控制台或VisualStudioCode终端中运行以下命令:npmconfigrmproxynpmconfigrmhttp
- php SPOF
贵哥的编程之路(热爱分享 为后来者)
PHP语言经典程序100题php开发语言
1.什么是单点故障(SPOF)?单点故障指的是系统中某个组件一旦失效,整个系统或服务就会不可用。常见的单点有:数据库、缓存、Web服务器、负载均衡、网络设备等。2.常见单点故障场景只有一台数据库服务器,宕机后所有业务不可用只有一台Redis缓存,挂掉后缓存全部失效只有一台Web服务器,挂掉后网站无法访问只有一个负载均衡节点,挂掉后流量无法分发只有一条网络链路,断开后所有服务失联3.消除单点故障的主
- 计算机网络技术
CZZDg
计算机网络
目录一.网络概述1.网络的概念2.网络发展是3.网络的四要素4.网络功能5.网络类型6.网络协议与标准7.网络中常见的概念8.网络拓补结构二.网络模型1.分层思想2.OSI七层模型3.TCP/IP五层模型4.数据的封装与解封装过程三.IP地址1.进制转换2.IP地址定义3.IP地址组成成分4.IP地址分类5.地址划分6、相关概念一.网络概述1.网络的概念两个主机通过传输介质和通信协议实现通信和资源
- cesium添加原生MVT矢量瓦片方案
zhu_zhu_xia
cesiumvuearcgiscesiumwebgljavascript
项目中需要基于cesium接入mvt格式的服务并支持属性拾取查询,通过一系列预研测试,最后选择cesium-mvt-imagery-provider开源插件完成,关键源码信息如下:npmicesiumcesium-mvt-imagery-provider//安装依赖包//加载图层importCesiumMVTImageryProviderfrom"cesium-mvt-imagery-provid
- 玩转Docker | 使用Docker部署gopeed下载工具
心随_风动
玩转Dockerdocker容器运维
玩转Docker|使用Docker部署gopeed下载工具前言一、gopeed介绍Gopeed简介主要特点二、系统要求环境要求环境检查Docker版本检查检查操作系统版本三、部署gopeed服务下载镜像创建容器检查容器状态检查服务端口安全设置四、访问gopeed应用五、测试与下载六、总结前言在当今信息爆炸的时代,高效地获取和管理网络资源变得尤为重要。无论是下载大型文件还是进行日常的数据传输,一个稳
- Docker指定网桥和指定网桥IP
$dockernetworklsNETWORKIDNAMEDRIVER7fca4eb8c647bridgebridge9f904ee27bf5nonenullcf03ee007fb4hosthostBridge默认bridge网络,我们可以使用dockernetworkinspect命令查看返回的网络信息,我们使用dockerrun命令是将网络自动应用到新的容器Host如果是hosts模式,启动容
- UNIX域套接字
1、UNIX域套接字的定义UNIX域套接字是进程间通信(IPC)的一种方式,不涉及网络协议栈,因此在同一台主机上的通信中,它比基于TCP/IP协议的网络套接字更快速、更高效。2、UNIX域套接字的分类字节流套接字(SOCK_STREAM):提供面向连接的、可靠的数据传输服务。数据报套接字(SOCK_DGRAM):提供无连接的数据传输服务,数据以独立的数据报形式传输。3、UNIX套接字与TCP/IP
- 【目标检测】机场内部目标检测数据集4106张YOLO+VOC格式
数据集格式:VOC格式+YOLO格式压缩包内含:3个文件夹,分别存储图片、xml、txt文件JPEGImages文件夹中jpg图片总计:4106Annotations文件夹中xml文件总计:4106labels文件夹中txt文件总计:4106标签种类数:7标签名称:["Ground_vehicles","Horizontal_sign","Runaway_limit","Taxiway","Ver
- Kimi Chat 1.5 与 2.0 架构升级对比
charles666666
人工智能transformer深度学习产品经理chatgpt
1.5版的MoE架构优化KimiChat1.5采用了优化后的MoE架构,其核心在于“专家网络动态路由”。这一机制类似于快递系统智能选择最优路径,能够根据输入数据的特性动态分配计算资源。这种优化显著提升了模型的计算效率,同时降低了硬件资源的浪费。在实际应用中,这意味着开发者可以在相同的硬件配置下处理更复杂的任务,或者在有限的资源下实现更高的性能。2.0的混合专家系统创新点与1.5版相比,KimiCh
- Excel控件Spire.XLS 更新至7.12.144 | 附下载
cocacola456
文档管理更新Excel控件Spire.XLS更新Spire.XLSSpire.XLS下载
Excel控件Spire.XLS更新至7.12.144,修复了转换PDF时字幕对齐的问题。Spire.XLS7.12.144更新修复修复了将Chart转换为Image时图表数据标签重复的问题。修复了CalculateAllValue方法抛出异常的问题。修复了将工作表转换为PDF时图表字幕对齐不正确的问题。
- 什么是OA系统?使用OA系统对企业有哪些好处?
OA系统(OfficeAutomationSystem),即办公自动化系统,是将现代化办公和计算机网络功能结合起来的一种新型的办公方式。是现代企业管理中一种重要的信息化工具,它通过计算机技术、网络技术和数据库技术等手段,实现企业内部办公流程的自动化和信息化管理。使企业的信息交流更加顺畅,办公流程更加高效,从而提高企业的运营效率和管理水平。一、主要功能1.文档管理文档存储与检索:OA系统可以集中存储
- Docker容器底层原理详解:从零理解容器化技术
Debug Your Career
面试docker容器dockerjava
一、容器本质:一个“隔离的进程”关键认知:Docker容器并不是一个完整的操作系统,而是一个被严格隔离的进程。这个进程拥有独立的文件系统、网络、进程视图等资源,但它直接运行在宿主机内核上(而虚拟机需要模拟硬件和操作系统)。类比理解:想象你在一个办公楼里租了一间独立办公室(容器)。你有自己的桌椅(文件系统)、电话分机(网络)、门牌号(主机名),但共享整栋楼的水电(宿主机内核)和电梯(硬件资源)。办公
- .NET 一款基于BGInfo的红队内网渗透工具
dot.Net安全矩阵
网络.net安全.netcoreweb安全矩阵
01阅读须知此文所提供的信息只为网络安全人员对自己所负责的网站、服务器等(包括但不限于)进行检测或维护参考,未经授权请勿利用文章中的技术资料对任何计算机系统进行入侵操作。利用此文所提供的信息而造成的直接或间接后果和损失,均由使用者本人负责。本文所提供的工具仅用于学习,禁止用于其他方面02基本介绍在内网渗透过程中,白名单绕过是红队常见的技术需求。Sharp4Bginfo.exe是一款基于微软签名工具
- 如何发现 Redis 中的 BigKey?
sevevty-seven
redisbootstrap数据库
如何发现Redis中的BigKey?Redis因其出色的性能,常被用作缓存、消息队列和会话存储。然而,在Redis的使用过程中,BigKey是一个不容忽视的问题。BigKey指的是存储了大量数据或包含大量成员的键。它们不仅会占用大量内存,还可能导致网络延迟、主从同步延迟,甚至在极端情况下引发Redis服务崩溃。因此,有效地发现和处理BigKey对于维护Redis服务的稳定性和性能至关重要。本文将深
- 11. TCP 滑动窗口、拥塞控制是什么,有什么区别
yqcoder
前端面试-服务协议tcp/ip网络php
总结滑动窗口:早期网络,通信双方不考虑网络拥挤情况,导致掉包。滑动窗口大小意味着有多少缓冲区接受数据。拥塞控制:防止过多数据注入网络中,拥塞控制是一个全局过程,控制网络流量。区别:滑动窗口解决掉包问题,拥塞控制解决网络拥塞问题。TCP滑动窗口与拥塞控制详解在TCP协议中,为了实现可靠传输和高效通信,引入了两个核心机制:滑动窗口(SlidingWindow)和拥塞控制(CongestionContr
- 上位机知识篇---Linux中的文件挂载
Atticus-Orion
上位机操作篇linux运维网络文件挂载
文章目录前言1.挂载的基本概念文件系统挂载点设备文件2.挂载的命令挂载文件系统示例卸载文件系统示例3.挂载的常用选项示例4.自动挂载(/etc/fstab文件)示例使用UUID挂载5.挂载网络文件系统(NFS)挂载NFS示例6.挂载ISO文件挂载ISO文件示例7.查看已挂载的文件系统8.挂载的注意事项9.挂载的常见问题挂载失败卸载失败10.总结前言在Linux系统中,文件挂载是指将一个文件系统(如
- 深度学习篇---昇腾NPU&CANN 工具包
Atticus-Orion
上位机知识篇图像处理篇深度学习篇深度学习人工智能NPU昇腾CANN
介绍昇腾NPU是华为推出的神经网络处理器,具有强大的AI计算能力,而CANN工具包则是面向AI场景的异构计算架构,用于发挥昇腾NPU的性能优势。以下是详细介绍:昇腾NPU架构设计:采用达芬奇架构,是一个片上系统,主要由特制的计算单元、大容量的存储单元和相应的控制单元组成。集成了多个CPU核心,包括控制CPU和AICPU,前者用于控制处理器整体运行,后者承担非矩阵类复杂计算。此外,还拥有AICore
- 计算机科学与技术
柳依依@
学习前端c4前端后端
计算机科学是一个庞大且关联性强的学科体系,初学者常面临以下痛点:-**知识点零散**:容易陷入"只见树木不见森林"的学习困境-**方向不明确**:面对海量技术栈不知从何入手-**体系缺失**:难以建立完整的知识网络1.计算机基础-计算机组成原理-冯·诺依曼体系-CPU/内存/IO设备-操作系统-进程与线程-内存管理-文件系统-计算机网络-TCP/IP模型-HTTP/HTTPS-网络安全2.编程能力
- Ubuntu 服务器虚拟主机,ubuntu云服务器虚拟机
Gamer42
Ubuntu服务器虚拟主机
ubuntu云服务器虚拟机内容精选换一换通过云服务器或者外部镜像文件创建私有镜像时,如果云服务器或镜像文件所在虚拟机的网络配置是静态IP地址时,您需要修改网卡属性为DHCP,以使私有镜像发放的新云服务器可以动态获取IP地址。本节以WindowsServer2008R2操作系统为例。其他操作系统配置方法略有区别,请参考对应操作系统的相关资料进行操作,文档中不对此进行详细说明后端虚拟机绑定EIP。登录
- 深度学习图像分类数据集—桃子识别分类
AI街潜水的八角
深度学习图像数据集深度学习分类人工智能
该数据集为图像分类数据集,适用于ResNet、VGG等卷积神经网络,SENet、CBAM等注意力机制相关算法,VisionTransformer等Transformer相关算法。数据集信息介绍:桃子识别分类:['B1','M2','R0','S3']训练数据集总共有6637张图片,每个文件夹单独放一种数据各子文件夹图片统计:·B1:1601张图片·M2:1800张图片·R0:1601张图片·S3:
- ModBus总线协议
小仇学长
STM32网络Modbus协议
一、知识点1.什么是Modbus协议?Modbus是一种工业通信协议,最早由Modicon公司在1979年提出,目的是用于PLC(可编程逻辑控制器)之间的数据通信。它是主从式通信,即一个主机(主设备)控制一个或多个从机(从设备)。它常用于RS-232、RS-485串口通信,也可以用于TCP/IP网络通信(叫做ModbusTCP)。2.核心特征特征项内容通信结构主从式(Master/Slave)通信
- 2.4 基于dpdk的用户态协议栈的实现
百亿苍狗
高性能网络设计专栏开发语言网络
操作系统PosixAPI所提供的网络接口,数据收发是基于用户态与内核态的频繁切换实现。而dpdk实现了绕过内核监管,直接在用户态访问网络硬件,避免频繁状态切换。DPDK安装与配置虚拟机环境配置检查是否支持多队列网卡cat/proc/interrupts|grepens33(获取整个机器的终端),结果19:4202120IO-APIC19-fasteoiens33,不支持多队列网卡。虚拟机关机,修改
- 使用NVIDIA NeRF将2D图像转换为逼真的3D模型(Python)
ByteWhiz
3dpython计算机视觉Python
使用NVIDIANeRF将2D图像转换为逼真的3D模型(Python)NeuralRadianceFields(NeRF)是一种强大的方法,可以将2D图像转换为逼真的3D模型。它使用神经网络来建模场景的辐射场,并通过渲染多个视角的图像来重建3D模型。在本文中,我们将使用Python和NVIDIANeRF库来实现这一过程。首先,我们需要安装所需的库。我们可以通过以下命令使用pip安装NVIDIANe
- TCP和UDP协议区别+应用场景+优缺点+常用协议
马拉萨的春天
一天一读基础知识点tcp/ipudp网络
文章目录1.TCP协议特点应用场景优点缺点运行于TCP协议之上的协议2.UDP协议特点应用场景优点缺点运行于UDP协议之上的协议TCP(TransmissionControlProtocol)和UDP(UserDatagramProtocol)是两种常用的传输层协议,它们在网络通信中扮演不同的角色,各有优缺点。1.TCP协议特点提供面向连接的、可靠的数据传输服务。使用三次握手建立连接,四次挥手断开
- apache ftpserver-CentOS config
gengzg
apache
<server xmlns="http://mina.apache.org/ftpserver/spring/v1"
xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance"
xsi:schemaLocation="
http://mina.apache.o
- 优化MySQL数据库性能的八种方法
AILIKES
sqlmysql
1、选取最适用的字段属性 MySQL可以很好的支持大数据量的存取,但是一般说来,数据库中的表越小,在它上面执行的查询也就会越快。因此,在创建表的时候,为了获得更好的 性能,我们可以将表中字段的宽度设得尽可能小。例如,在定义邮政编码这个字段时,如果将其设置为CHAR(255),显然给数据库增加了不必要的空间,甚至使用VARCHAR这种类型也是多余的,因为CHAR(6)就可以很
- JeeSite 企业信息化快速开发平台
Kai_Ge
JeeSite
JeeSite 企业信息化快速开发平台
平台简介
JeeSite是基于多个优秀的开源项目,高度整合封装而成的高效,高性能,强安全性的开源Java EE快速开发平台。
JeeSite本身是以Spring Framework为核心容器,Spring MVC为模型视图控制器,MyBatis为数据访问层, Apache Shiro为权限授权层,Ehcahe对常用数据进行缓存,Activit为工作流
- 通过Spring Mail Api发送邮件
120153216
邮件main
原文地址:http://www.open-open.com/lib/view/open1346857871615.html
使用Java Mail API来发送邮件也很容易实现,但是最近公司一个同事封装的邮件API实在让我无法接受,于是便打算改用Spring Mail API来发送邮件,顺便记录下这篇文章。 【Spring Mail API】
Spring Mail API都在org.spri
- Pysvn 程序员使用指南
2002wmj
SVN
源文件:http://ju.outofmemory.cn/entry/35762
这是一篇关于pysvn模块的指南.
完整和详细的API请参考 http://pysvn.tigris.org/docs/pysvn_prog_ref.html.
pysvn是操作Subversion版本控制的Python接口模块. 这个API接口可以管理一个工作副本, 查询档案库, 和同步两个.
该
- 在SQLSERVER中查找被阻塞和正在被阻塞的SQL
357029540
SQL Server
SELECT R.session_id AS BlockedSessionID ,
S.session_id AS BlockingSessionID ,
Q1.text AS Block
- Intent 常用的用法备忘
7454103
.netandroidGoogleBlogF#
Intent
应该算是Android中特有的东西。你可以在Intent中指定程序 要执行的动作(比如:view,edit,dial),以及程序执行到该动作时所需要的资料 。都指定好后,只要调用startActivity(),Android系统 会自动寻找最符合你指定要求的应用 程序,并执行该程序。
下面列出几种Intent 的用法
显示网页:
- Spring定时器时间配置
adminjun
spring时间配置定时器
红圈中的值由6个数字组成,中间用空格分隔。第一个数字表示定时任务执行时间的秒,第二个数字表示分钟,第三个数字表示小时,后面三个数字表示日,月,年,< xmlnamespace prefix ="o" ns ="urn:schemas-microsoft-com:office:office" />
测试的时候,由于是每天定时执行,所以后面三个数
- POJ 2421 Constructing Roads 最小生成树
aijuans
最小生成树
来源:http://poj.org/problem?id=2421
题意:还是给你n个点,然后求最小生成树。特殊之处在于有一些点之间已经连上了边。
思路:对于已经有边的点,特殊标记一下,加边的时候把这些边的权值赋值为0即可。这样就可以既保证这些边一定存在,又保证了所求的结果正确。
代码:
#include <iostream>
#include <cstdio>
- 重构笔记——提取方法(Extract Method)
ayaoxinchao
java重构提炼函数局部变量提取方法
提取方法(Extract Method)是最常用的重构手法之一。当看到一个方法过长或者方法很难让人理解其意图的时候,这时候就可以用提取方法这种重构手法。
下面是我学习这个重构手法的笔记:
提取方法看起来好像仅仅是将被提取方法中的一段代码,放到目标方法中。其实,当方法足够复杂的时候,提取方法也会变得复杂。当然,如果提取方法这种重构手法无法进行时,就可能需要选择其他
- 为UILabel添加点击事件
bewithme
UILabel
默认情况下UILabel是不支持点击事件的,网上查了查居然没有一个是完整的答案,现在我提供一个完整的代码。
UILabel *l = [[UILabel alloc] initWithFrame:CGRectMake(60, 0, listV.frame.size.width - 60, listV.frame.size.height)]
- NoSQL数据库之Redis数据库管理(PHP-REDIS实例)
bijian1013
redis数据库NoSQL
一.redis.php
<?php
//实例化
$redis = new Redis();
//连接服务器
$redis->connect("localhost");
//授权
$redis->auth("lamplijie");
//相关操
- SecureCRT使用备注
bingyingao
secureCRT每页行数
SecureCRT日志和卷屏行数设置
一、使用securecrt时,设置自动日志记录功能。
1、在C:\Program Files\SecureCRT\下新建一个文件夹(也就是你的CRT可执行文件的路径),命名为Logs;
2、点击Options -> Global Options -> Default Session -> Edite Default Sett
- 【Scala九】Scala核心三:泛型
bit1129
scala
泛型类
package spark.examples.scala.generics
class GenericClass[K, V](val k: K, val v: V) {
def print() {
println(k + "," + v)
}
}
object GenericClass {
def main(args: Arr
- 素数与音乐
bookjovi
素数数学haskell
由于一直在看haskell,不可避免的接触到了很多数学知识,其中数论最多,如素数,斐波那契数列等,很多在学生时代无法理解的数学现在似乎也能领悟到那么一点。
闲暇之余,从图书馆找了<<The music of primes>>和<<世界数学通史>>读了几遍。其中素数的音乐这本书与软件界熟知的&l
- Java-Collections Framework学习与总结-IdentityHashMap
BrokenDreams
Collections
这篇总结一下java.util.IdentityHashMap。从类名上可以猜到,这个类本质应该还是一个散列表,只是前面有Identity修饰,是一种特殊的HashMap。
简单的说,IdentityHashMap和HashM
- 读《研磨设计模式》-代码笔记-享元模式-Flyweight
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
import java.util.ArrayList;
import java.util.Collection;
import java.util.HashMap;
import java.util.List;
import java
- PS人像润饰&调色教程集锦
cherishLC
PS
1、仿制图章沿轮廓润饰——柔化图像,凸显轮廓
http://www.howzhi.com/course/retouching/
新建一个透明图层,使用仿制图章不断Alt+鼠标左键选点,设置透明度为21%,大小为修饰区域的1/3左右(比如胳膊宽度的1/3),再沿纹理方向(比如胳膊方向)进行修饰。
所有修饰完成后,对该润饰图层添加噪声,噪声大小应该和
- 更新多个字段的UPDATE语句
crabdave
update
更新多个字段的UPDATE语句
update tableA a
set (a.v1, a.v2, a.v3, a.v4) = --使用括号确定更新的字段范围
- hive实例讲解实现in和not in子句
daizj
hivenot inin
本文转自:http://www.cnblogs.com/ggjucheng/archive/2013/01/03/2842855.html
当前hive不支持 in或not in 中包含查询子句的语法,所以只能通过left join实现。
假设有一个登陆表login(当天登陆记录,只有一个uid),和一个用户注册表regusers(当天注册用户,字段只有一个uid),这两个表都包含
- 一道24点的10+种非人类解法(2,3,10,10)
dsjt
算法
这是人类算24点的方法?!!!
事件缘由:今天晚上突然看到一条24点状态,当时惊为天人,这NM叫人啊?以下是那条状态
朱明西 : 24点,算2 3 10 10,我LX炮狗等面对四张牌痛不欲生,结果跑跑同学扫了一眼说,算出来了,2的10次方减10的3次方。。我草这是人类的算24点啊。。
然后么。。。我就在深夜很得瑟的问室友求室友算
刚出完题,文哥的暴走之旅开始了
5秒后
- 关于YII的菜单插件 CMenu和面包末breadcrumbs路径管理插件的一些使用问题
dcj3sjt126com
yiiframework
在使用 YIi的路径管理工具时,发现了一个问题。 <?php
- 对象与关系之间的矛盾:“阻抗失配”效应[转]
come_for_dream
对象
概述
“阻抗失配”这一词组通常用来描述面向对象应用向传统的关系数据库(RDBMS)存放数据时所遇到的数据表述不一致问题。C++程序员已经被这个问题困扰了好多年,而现在的Java程序员和其它面向对象开发人员也对这个问题深感头痛。
“阻抗失配”产生的原因是因为对象模型与关系模型之间缺乏固有的亲合力。“阻抗失配”所带来的问题包括:类的层次关系必须绑定为关系模式(将对象
- 学习编程那点事
gcq511120594
编程互联网
一年前的夏天,我还在纠结要不要改行,要不要去学php?能学到真本事吗?改行能成功吗?太多的问题,我终于不顾一切,下定决心,辞去了工作,来到传说中的帝都。老师给的乘车方式还算有效,很顺利的就到了学校,赶巧了,正好学校搬到了新校区。先安顿了下来,过了个轻松的周末,第一次到帝都,逛逛吧!
接下来的周一,是我噩梦的开始,学习内容对我这个零基础的人来说,除了勉强完成老师布置的作业外,我已经没有时间和精力去
- Reverse Linked List II
hcx2013
list
Reverse a linked list from position m to n. Do it in-place and in one-pass.
For example:Given 1->2->3->4->5->NULL, m = 2 and n = 4,
return
- Spring4.1新特性——页面自动化测试框架Spring MVC Test HtmlUnit简介
jinnianshilongnian
spring 4.1
目录
Spring4.1新特性——综述
Spring4.1新特性——Spring核心部分及其他
Spring4.1新特性——Spring缓存框架增强
Spring4.1新特性——异步调用和事件机制的异常处理
Spring4.1新特性——数据库集成测试脚本初始化
Spring4.1新特性——Spring MVC增强
Spring4.1新特性——页面自动化测试框架Spring MVC T
- Hadoop集群工具distcp
liyonghui160com
1. 环境描述
两个集群:rock 和 stone
rock无kerberos权限认证,stone有要求认证。
1. 从rock复制到stone,采用hdfs
Hadoop distcp -i hdfs://rock-nn:8020/user/cxz/input hdfs://stone-nn:8020/user/cxz/运行在rock端,即源端问题:报版本
- 一个备份MySQL数据库的简单Shell脚本
pda158
mysql脚本
主脚本(用于备份mysql数据库): 该Shell脚本可以自动备份
数据库。只要复制粘贴本脚本到文本编辑器中,输入数据库用户名、密码以及数据库名即可。我备份数据库使用的是mysqlump 命令。后面会对每行脚本命令进行说明。
1. 分别建立目录“backup”和“oldbackup” #mkdir /backup #mkdir /oldbackup
- 300个涵盖IT各方面的免费资源(中)——设计与编码篇
shoothao
IT资源图标库图片库色彩板字体
A. 免费的设计资源
Freebbble:来自于Dribbble的免费的高质量作品。
Dribbble:Dribbble上“免费”的搜索结果——这是巨大的宝藏。
Graphic Burger:每个像素点都做得很细的绝佳的设计资源。
Pixel Buddha:免费和优质资源的专业社区。
Premium Pixels:为那些有创意的人提供免费的素材。
- thrift总结 - 跨语言服务开发
uule
thrift
官网
官网JAVA例子
thrift入门介绍
IBM-Apache Thrift - 可伸缩的跨语言服务开发框架
Thrift入门及Java实例演示
thrift的使用介绍
RPC
POM:
<dependency>
<groupId>org.apache.thrift</groupId>