SIR模型和Python实现
一、SIR模型介绍
SIR模型时传染病中最基础最核心的模型,研究的是某个封闭地区的疫情传播规律。
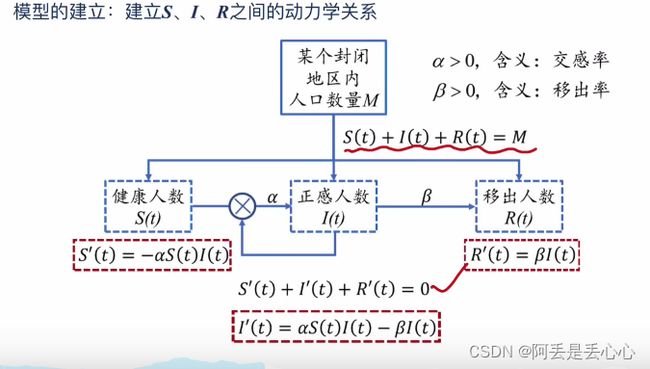
SIR模型的动力学关系如下图:
健康人数S的变化与 健康人数S和正感人数I的乘积(代表健康人数和正感人数的接触)成正比,其中α代表交叉感染率
移出人数的变化与正感人数的数量成正比,其中β代表回复率。
基于上面的是自,SIR模型可以表示成一个常微分方程组如下图:
当s(t)=β/α时就是病毒最严重的时候;
![]() 表示S的反函数,代表S越大,t越小,越早结束疫情
表示S的反函数,代表S越大,t越小,越早结束疫情
封城就是为了较低交叉感染率α,并提高治愈率β
以上就是关于SIR模型的简单介绍,我是从B站上学习到的,更多可以看遇见数学的视频:数学建模系列之SIR传染病模型(1)——模型的建立与演绎_哔哩哔哩_bilibili
接下来我将用Python实现SIR模型在某真实社交网络(基于networkx)上的传播过程模拟。
代码参考了这篇文章:人群接触网络中的 SIR 疫情模拟 - 云+社区 - 腾讯云
二、接触网络中的SIR模型
在 SIR 模型中,假设人之间是随机接触的。如果人之间的接触关系不是随机的,而是形成了一个接触网络。那么在这个网络中,每个人接触到感染者的概率不再相等,而与他在网络中的位置相关。
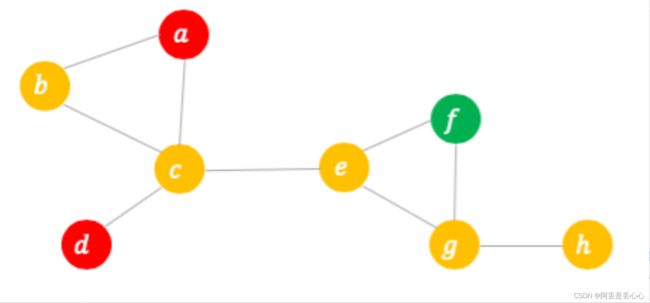
比如对于这张图,c接触到感染者的概率远远大于h,因此其染病的概率也会更高。
因此我们可以更新SIR模型的规则:(这也是参考了上面腾讯云的文章)
与传统 SIR 模型类似,有两个重要的参数:感染率 α 和恢复率 β。我们需要给每个节点引入一个状态,取值为 S,I,R 中的一种。每一个时间步中,需要动态对每一个节点的状态进行更新。更新规则如下:
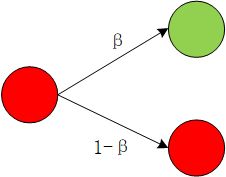
- 如果当前节点是恢复者,则下一步,节点状态依然是恢复者。
- 如果当前节点是感染者,则下一步,β的概率转化为恢复者。1 - β的概率依然是感染者。
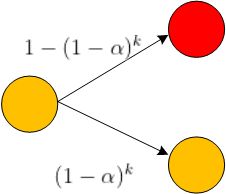
- 如果当前节点是易感者(健康者),则需要计算其邻居节点中感染者的数量,假设其有 k 个邻居为感染者。则该节点下一步转化为感染者的概率为
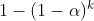 ,否则继续保持易感者状态。
,否则继续保持易感者状态。
下面这个概率的含义就是对于每个邻居感染者,易感者有1-α的概率保持健康,故有![]() 概率保持健康
概率保持健康
三、Python实现SIR模型
1.导入数据
这里我导入的是一个节点表和一个边表,边表用来构建网络图,节点表用来选择初始感染者:
import pandas
import csv
import random
node_df = pandas.read_csv('E:/data/节点.csv')
all_nodes_list = node_df.values.tolist() #获取文件中所有节点
edge = [] #获取所有边
with open('E:/data/边.csv','r',encoding='utf-8-sig') as f:
data = f.readlines()
for line in data:
line = list(line.replace('\r','').replace('\n','').replace('\t','').split(','))
single_edge = tuple([line[1],line[4]])
edge.append(single_edge)2.绘制初始网络
所有节点都是健康的
import networkx as nx
ba = nx.Graph()
ba.add_edges_from(edge)
for node in ba.nodes():
ba.nodes[node]["state"] = "S"3. 更新网络节点状态
先考虑单个节点的更新
我们使用一个简单的函数来实现一个节点的状态的更新。
首先,如果一个节点是恢复者,那么下一步还是恢复者,其节点状态保持不变。 如果一个节点是感染者,那么其恢复的概率是 β。用程序实现的方法为,先均匀生成一个0到1的随机数 p,如果 p < β,则节点恢复,否则节点依然处于感染状态。
如一个节点是易感者,先要去其邻居节点中看看一共有多少个邻居是感染者,有 k 个邻居是感染者,那么当前节点被感染的概率是 1 - (1 - α)k。我们生成一个0到1的随机数 p,如果 p < 1 - (1 - α)k,则节点被感染,否则不被感染。
import random
# 根据 SIR 模型,更新单一节点的状态
def updateNodeState(G,node, alpha, beta):
if G.nodes[node]["state"] == "I": #感染者
p = random.random() # 生成一个0到1的随机数
if p < beta: # gamma的概率恢复
G.nodes[node]["state"] = "R" #将节点状态设置成“R”
elif G.nodes[node]["state"] == "S": #易感者
p = random.random() # 生成一个0到1的随机数
k = 0 # 计算邻居中的感染者数量
for neibor in G.adj[node]: # 查看所有邻居状态,遍历邻居用 G.adj[node]
if G.nodes[neibor]["state"] == "I": #如果这个邻居是感染者,则k加1
k = k + 1
if p < 1 - (1 - alpha)**k: # 易感者被感染
G.nodes[node]["state"] = "I" 再对网络中所有节点进行更新
def updateNetworkState(G, alpha, beta):
for node in G: #遍历图中节点,每一个节点状态进行更新
updateNodeState(G,node, alpha, beta)用函数countSIR统计三类人群数量
# 计算三类人群的数量
def countSIR(G):
S = 0;I = 0
for node in G:
if G.nodes[node]["state"] == "S":
S = S + 1
elif G.nodes[node]["state"] == "I":
I = I + 1
return S,I, len(G.nodes) - S - I 所有函数都准备好后,我们进行网络和参数初始化设置
#随机选取一个节点为初始感染者
ba.nodes["123549878"]["state"] = "I"
days = 50 #设置模拟的天数
alpha = 0.0003 #感染率
beta = 0.10 #恢复率
#设置不同人群的显示颜色,易感者为橘色,感染者为红色,恢复者为绿色
color_dict = {"S":"orange","I":"red","R":"green"} 在图中开始SIR模型的模拟,设置模拟天数,开始执行模拟过程
# 模拟天数为days,更新节点状态
import matplotlib.pyplot as plt
#fig,ax = plt.subplots(111)
%matplotlib inline
import time
SIR_list = []
for t in range(0,days):
updateNetworkState(ba,alpha,beta) #对网络状态进行模拟更新
SIR_list.append(list(countSIR(ba))) #计算更新后三种节点的数量模型结果可视化
df = pd.DataFrame(SIR_list,columns=["S","I","R"])
df.plot(figsize=(9,6),color=[color_dict.get(x) for x in df.columns])我的数据得到的结果如下
我的网络结果中分为几个隔离比较大的社区,所以有一部分人群始终没有被感染。
4.探究不同节点作为初始感染者传播网络的区别
比如我想知道在一个网络中的谣言传播情况,想看看随机选一个节点,通过这个节点看看最后有多少个人会接触到这个谣言(其实就是传播停止时的恢复者)
import matplotlib.pyplot as plt
import time
import pandas as pd
#fig,ax = plt.subplots(111)
%matplotlib inline
import time
# 获得所有节点的属性
def get_last_node_state(all_nodes_list,G):
SIR_result = []
for item in all_nodes_list:
node_attri = []
node_attri.extend(item)
try:
for node in G.nodes():
G.nodes[node]["state"] = "S" #将网络中所有节点设为健康者
G.nodes[str(item[0])]["state"] = "I" #将某一节点设为感染者
for t in range(0,days):
updateNetworkState(G,beta,gamma) #对网络状态进行模拟更新
node_result = pd.DataFrame([i[1] for i in G.nodes(data=True)], index=[i[0] for i in G.nodes(data=True)])
state_count = node_result.groupby('state')['label'].count().to_dict() #按感染情况分组
R_count = state_count['R'] #计算恢复者数量
node_attri.append(R_count)
SIR_result.append(node_attri)
except Exception as e:
print(e)
return SIR_result #返回结果形式为[节点id,节点接触到的人数(恢复者人数)]
#执行函数
SIR_result = get_last_node_state(all_nodes_list,ba)