sklearn基础篇(十)-- 非负矩阵分解与t-SNE
1 非负矩阵分解(NFM)
NMF(Non-negative matrix factorization),即对于任意给定的一个非负矩阵 V \pmb{V} VVV,其能够寻找到一个非负矩阵 W \pmb{W} WWW和一个非负矩阵 H \pmb{H} HHH,满足条件 V = W ∗ H \pmb{V=W*H} V=W∗HV=W∗HV=W∗H,从而将一个非负的矩阵分解为左右两个非负矩阵的乘积。其中, V \pmb{V} VVV矩阵中每一列代表一个观测(observation),每一行代表一个特征(feature); W \pmb{W} WWW矩阵称为基矩阵, H \pmb{H} HHH矩阵称为系数矩阵或权重矩阵。这时用系数矩阵 H \pmb{H} HHH代替原始矩阵,就可以实现对原始矩阵进行降维,得到数据特征的降维矩阵,从而减少存储空间。 过程如下图所示:
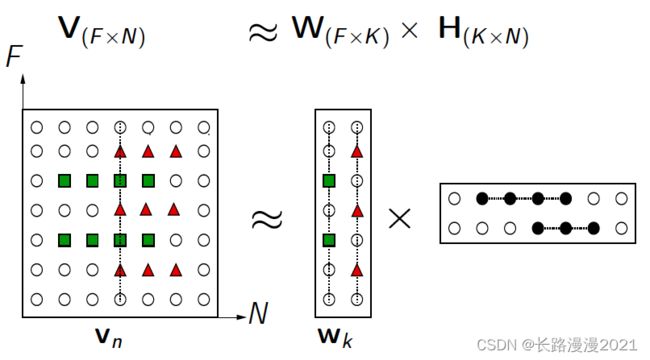
NMF本质上说是一种矩阵分解的方法,它的特点是可以将一个大的非负矩阵分解为两个小的非负矩阵,又因为分解后的矩阵也是非负的,所以也可以继续分解。NMF的应用包括但不限于提取特征、快速识别、基因和语音的检测等等。
从矩阵空间的角度分析,NMF的意义在于在原空间中寻找一组新基底并将原数据投影到该基底上去。原非负矩阵 V \pmb{V} VVV对应原空间中的原数据,分解之后的两个非负矩阵 W \pmb{W} WWW和 H \pmb{H} HHH分别对应寻找得到的新基底和投影在新基底上的数值。
下面用数学语言对NMF进行描述,NMF原理很简单,与SVD将矩阵分解为三个矩阵类似,NMF将矩阵分解为两个小矩阵,比如原始矩阵 V m × n \pmb{V}_{m\times n} VVVm×n分解为 W m × k \pmb{W}_{m\times k} WWWm×k与 H k × n \pmb{H}_{k\times n} HHHk×n的乘积,即:
V m × n ≈ W m × k H k × n (1-1) \pmb{V}_{m\times n}\approx \pmb{W}_{m\times k}\pmb{H}_{k\times n}\tag{1-1} VVVm×n≈WWWm×kHHHk×n(1-1)
这里,要求 A , W , H A,W,H A,W,H中的元素都非负,而参数估计也很简单,最小化如下的平方损失即可:
L ( V , W , H ) = 1 2 ∑ i = 1 m ∑ j = 1 n ( V i j − ( W H ) i j ) 2 = 1 2 ∑ i = 1 m ∑ j = 1 n ( V i j − ∑ l = 1 k W i l H l j ) 2 (1-2) L(V,W,H)=\frac{1}{2}\sum_{i=1}^m\sum_{j=1}^n(V_{ij}-(WH)_{ij})^2=\frac{1}{2}\sum_{i=1}^m\sum_{j=1}^n(V_{ij}-\sum_{l=1}^kW_{il}H_{lj})^2\tag{1-2} L(V,W,H)=21i=1∑mj=1∑n(Vij−(WH)ij)2=21i=1∑mj=1∑n(Vij−l=1∑kWilHlj)2(1-2)
所以:
W ∗ , H ∗ = a r g min W , H L ( V , W , H ) (1-3) W^*,H^*=arg\min_{W,H}L(V,W,H)\tag{1-3} W∗,H∗=argW,HminL(V,W,H)(1-3)
对于参数估计,采用梯度下降即可,下面推导一下:
∂ L ∂ W i k = ∑ j ( V i j − ( W H ) i j ) ⋅ ∂ ( W H ) i j ∂ W i k = ∑ j ( V i j − ( W H ) i j ) ⋅ ( − H k j ) = − ∑ j V i j H k j + ∑ j ( W H ) i j H k j = ( W H H T ) i k − ( V H T ) i k (1-4) \begin{aligned} \frac{\partial L}{\partial W_{ik}}&=\sum_j(V_{ij}-(WH)_{ij})\cdot \frac{\partial (WH)_{ij}}{\partial W_{ik}}\\ &=\sum_j(V_{ij}-(WH)_{ij})\cdot (-H_{kj})\\ &=-\sum_j V_{ij}H_{kj}+\sum_j(WH)_{ij}H_{kj}\\ &=(WHH^T)_{ik}-(VH^T)_{ik} \end{aligned}\tag{1-4} ∂Wik∂L=j∑(Vij−(WH)ij)⋅∂Wik∂(WH)ij=j∑(Vij−(WH)ij)⋅(−Hkj)=−j∑VijHkj+j∑(WH)ijHkj=(WHHT)ik−(VHT)ik(1-4)
类似地:
∂ L ∂ H k j = ( W T W H ) k j − ( W T V ) k j (1-5) \frac{\partial L}{\partial H_{kj}}=(W^TWH)_{kj}-(W^TV)_{kj}\tag{1-5} ∂Hkj∂L=(WTWH)kj−(WTV)kj(1-5)
所以,梯度下降的更新公式可以表示如下:
W i k ← W i k + α 1 [ ( V H T ) i k − ( W H H T ) i k ] H k j ← H k j + α 2 [ ( W T V ) k j − ( W T W H ) k j ] (1-6) W_{ik}\leftarrow W_{ik}+\alpha_1[(VH^T)_{ik}-(WHH^T)_{ik}]\\ H_{kj}\leftarrow H_{kj}+\alpha_2[(W^TV)_{kj}-(W^TWH)_{kj}]\tag{1-6} Wik←Wik+α1[(VHT)ik−(WHHT)ik]Hkj←Hkj+α2[(WTV)kj−(WTWH)kj](1-6)
这里, α 1 > 0 , α 2 > 0 \alpha_1>0,\alpha_2>0 α1>0,α2>0为学习率,如果我们巧妙的设置:
α 1 = W i k ( W H H T ) i k α 2 = H k j ( W T W H ) k j (1-7) \alpha_1=\frac{W_{ik}}{(WHH^T)_{ik}}\\ \alpha_2=\frac{H_{kj}}{(W^TWH)_{kj}}\tag{1-7} α1=(WHHT)ikWikα2=(WTWH)kjHkj(1-7)
那么,迭代公式为:
W i k ← W i k ⋅ ( V H T ) i k ( W H H T ) i k H k j ← H k j ⋅ ( W T V ) k j ( W T W H ) k j (1-8) W_{ik}\leftarrow W_{ik}\cdot\frac{(VH^T)_{ik}}{(WHH^T)_{ik}}\\ H_{kj}\leftarrow H_{kj}\cdot\frac{(W^TV)_{kj}}{(W^TWH)_{kj}}\tag{1-8} Wik←Wik⋅(WHHT)ik(VHT)ikHkj←Hkj⋅(WTWH)kj(WTV)kj(1-8)
可以发现该迭代公式也很好的满足了我们的约束条件, W , H \pmb{W},\pmb{H} WWW,HHH在迭代过程中始终非负。
上面采用的是迭代法,一步步逼近最终的结果,当计算得到的两个矩阵 W \pmb{W} WWW和 H \pmb{H} HHH收敛时,就说明分解成功。需要注意的是,原矩阵和分解之后两个矩阵的乘积并不要求完全相等,可以存在一定程度上的误差。如果要在计算机中实现NMF,则可以根据如下图所示的步骤进行

详细求解过程可以参考:非负矩阵分解(NMF)迭代公式推导证明
1.1 将 NMF 应用于人脸图像
与使用PCA不同,我们需要保证数据是正的。这说明数据相对于原点(0, 0)的位置实际上对NMF很重要。因此,你可以将提取出来的非负分量看作是从(0, 0)到数据的方向。下面的例子给出了NMF 在二维玩具数据上的结果:
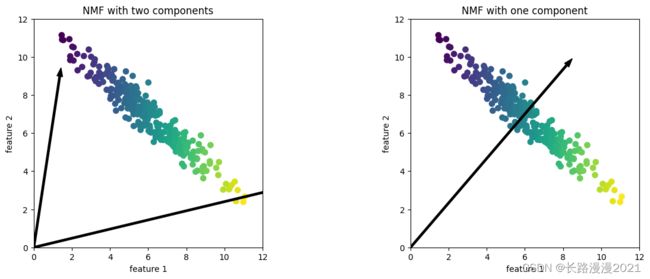
首先,我们来观察分量个数如何影响NMF 重建数据的好坏:
from sklearn.datasets import fetch_lfw_people
from sklearn.model_selection import train_test_split
# 导入人脸图像
people = fetch_lfw_people(min_faces_per_person=20, resize=0.7)
image_shape = people.images[0].shape
# 每类最多取50张
mask = np.zeros(people.target.shape, dtype=np.bool)
for target in np.unique(people.target):
mask[np.where(people.target == target)[0][:50]] = 1
X_people = people.data[mask]
y_people = people.target[mask]
# 将灰度值缩放到0到1之间,而不是在0到255之间,以得到更好的数据稳定性
X_people = X_people / 255
# 将数据集分为训练集和测试集
X_train, X_test, y_train, y_test = train_test_split(X_people, y_people, stratify=y_people, random_state=0)
mglearn.plots.plot_nmf_faces(X_train, X_test, image_shape)

反向变换的数据质量与使用PCA时类似,但要稍差一些。这是符合预期的,因为PCA找到的是重建的最佳方向。NMF通常并不用于对数据进行重建或编码,而是用于在数据中寻找有趣的模式。
我们尝试仅提取一部分分量(比如15个),初步观察一下数据。
from sklearn.decomposition import NMF
nmf = NMF(n_components=15, random_state=0)
nmf.fit(X_train)
X_train_nmf = nmf.transform(X_train)
X_test_nmf = nmf.transform(X_test)
fig, axes = plt.subplots(3, 5, figsize=(15, 12),
subplot_kw={'xticks': (), 'yticks': ()})
for i, (component, ax) in enumerate(zip(nmf.components_, axes.ravel())):
ax.imshow(component.reshape(image_shape))
ax.set_title("{}. component".format(i))

你可以清楚地看到,分量3(component 3)显示了稍微向右转动的人脸,而分量7(component 7)则显示了稍微向左转动的人脸。我们来看一下这两个分量特别大的那些图像,
compn = 3
# 第3个分量的系数较大的人脸
inds = np.argsort(X_train_nmf[:, compn])[::-1]
fig, axes = plt.subplots(2, 5, figsize=(15, 8),
subplot_kw={'xticks': (), 'yticks': ()})
fig.suptitle("Large component 3")
for i, (ind, ax) in enumerate(zip(inds, axes.ravel())):
ax.imshow(X_train[ind].reshape(image_shape))

compn = 7
# 第7个分量的系数较大的人脸
inds = np.argsort(X_train_nmf[:, compn])[::-1]
fig.suptitle("Large component 7")
fig, axes = plt.subplots(2, 5, figsize=(15, 8),
subplot_kw={'xticks': (), 'yticks': ()})
for i, (ind, ax) in enumerate(zip(inds, axes.ravel())):
ax.imshow(X_train[ind].reshape(image_shape))

正如所料,分量3系数较大的人脸都是向右看的人脸(图4),而分量7系数较大的人脸都向左看(图5)。如前所述,提取这样的模式最适合于具有叠加结构的数据,包括音频、基因表达和文本数据。我们通过一个模拟数据的例子来看一下这种用法。
1.2 盲源信号分离
假设我们对一个信号感兴趣,它是三个不同信号源合成的:
S = mglearn.datasets.make_signals()
plt.figure(figsize=(6, 1))
plt.plot(S, '-')
plt.xlabel('Time')
plt.ylabel('Signal')

不幸的是,我们无法观测到原始信号,只能观测到三个信号的叠加混合。我们想要将混合信号分解为原始分量。假设我们有许多种不同的方法来观测混合信号(比如有100台测量装置),每种方法都为我们提供了一系列测量结果。
# 将数据混合成100维的状态
A = np.random.RandomState(0).uniform(size=(100, 3))
X = np.dot(S, A.T)
print("Shape of measurements: {}".format(X.shape)) # Shape of measurements: (2000, 100)
我们可以用NMF和PCA来还原这三个信号:
from sklearn.decomposition import PCA
pca = PCA(n_components=3)
H = pca.fit_transform(X)
给出了NMF和PCA发现的信号活动:
models = [X, S, S_, H]
names = ['Observations (first three measurements)',
'True sources',
'NMF recovered signals',
'PCA recovered signals']
fig, axes = plt.subplots(4, figsize=(8, 4), gridspec_kw={'hspace': .5}, subplot_kw={'xticks': (), 'yticks': ()})
for model, name, ax in zip(models, names, axes):
ax.set_title(name)
ax.plot(model[:, :3], '-')
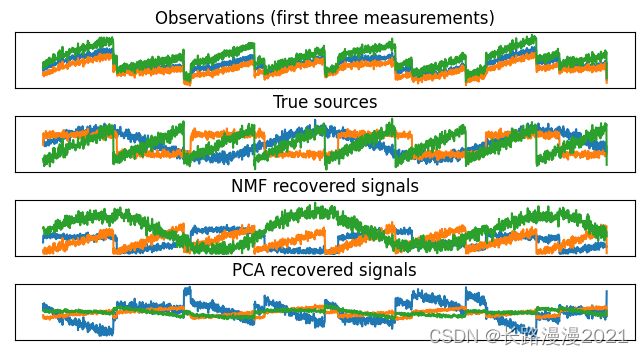
图中包含来自X的100次测量中的3次,用于参考。可以看到,NMF在发现原始信号源时得到了不错的结果,而PCA则失败了,仅使用第一个成分来解释数据中的大部分变化。要记住,NMF生成的分量是没有顺序的。在这个例子中,NMF分量的顺序与原始信号完全相同(参见三条曲线的颜色),但这纯属偶然。
2 用t-SNE进行流形学习
流形学习算法主要用于可视化,因此很少用来生成两个以上的新特征。其中一些算法(包括t-SNE)计算训练数据的一种新表示,但不允许变换新数据。这意味着这些算法不能用于测试集:更确切地说,它们只能变换用于训练的数据。流形学习对探索性数据分析是很有用的,但如果最终目标是监督学习的话,则很少使用。t-SNE背后的思想是找到数据的一个二维表示,尽可能地保持数据点之间的距离。t-SNE首先给出每个数据点的随机二维表示,然后尝试让在原始特征空间中距离较近的点更加靠近,原始特征空间中相距较远的点更加远离。t-SNE重点关注距离较近的点,而不是保持距离较远的点之间的距离。换句话说,它试图保存那些表示哪些点比较靠近的信息。
t-SNE全称为 t-distributed Stochastic Neighbor Embedding,翻译为 t分布-随机邻近嵌入。首先,t-分布是关于样本(而非总体)的t 变换值的分布,它是对u 变换变量值的标准正态分布的估计分布,是一位学生首先提出的,所以 t-分布全称:学生t-分布。其次,t-SNE本质是一种嵌入模型,能够将高维空间中的数据映射到低维空间中,并保留数据集的局部特性。t-SNE 可以算是目前效果很好的数据降维和可视化方法之一。缺点主要是占用内存较多、运行时间长。
t-SNE变换后,如果在低维空间中具有可分性,则数据是可分的;如果在低维空间中不可分,则可能是因为数据集本身不可分,或者数据集中的数据不适合投影到低维空间。该算法在论文中非常常见,主要用于高维数据的降维和可视化。Visualizing Data using t-SNE,2008年发表在Journal of Machine Learning Research。
我们将对scikit-learn包含的一个手写数字数据集2应用t-SNE流形学习算法。在这个数据集中,每个数据点都是0到9之间手写数字的一张8×8灰度图像。
from sklearn.datasets import load_digits
digits = load_digits()
print("手写数字数据集的形状= {}".format(digits.data.shape)) # (1797, 64)
print("手写数字数据集中图片的形状= {}".format(digits.images.shape)) # (1797, 8, 8)
fig, axes = plt.subplots(2, 5, figsize=(10, 5),subplot_kw={'xticks':(), 'yticks': ()})
for ax, img in zip(axes.ravel(), digits.images):
ax.imshow(img)

我们用PCA将降到二维的数据可视化。我们对前两个主成分作图,并按类别对数据点着色。
# 构建一个 PCA 模型
pca = PCA(n_components=2)
pca.fit(digits.data)
# 将 digits 数据变换到前两个主成分的方向上
digits_pca = pca.transform(digits.data)
colors = ["#476A2A", "#7851B8", "#BD3430", "#4A2D4E", "#875525",
"#A83683", "#4E655E", "#853541", "#3A3120", "#535D8E"]
plt.figure(figsize=(10, 10))
plt.xlim(digits_pca[:, 0].min(), digits_pca[:, 0].max())
plt.ylim(digits_pca[:, 1].min(), digits_pca[:, 1].max())
for i in range(len(digits.data)):
# 将数据绘制成文本图(代替散点图)
plt.text(digits_pca[i, 0], digits_pca[i, 1], str(digits.target[i]),
color = colors[digits.target[i]],
fontdict={'weight': 'bold', 'size': 9})
plt.xlabel("第一个主成分")
plt.ylabel("第二个主成分")

实际上,这里我们用每个类别对应的数字作为符号来显示每个类别的位置。利用前两个主成分可以将数字0、6和4相对较好地分开,尽管仍有重叠。大部分其他数字都大量重叠在一起。
我们将t-SNE应用于同一个数据集,并对结果进行比较。由于t-SNE不支持变换新数据,所以TSNE类没有transform方法。我们可以调用fit_transform方法来代替,它会构建模型并立刻返回变换后的数据:
from sklearn.manifold import TSNE
tsne = TSNE(random_state=42)
# 使用 fit_transform() 代替 fit(), 因为 TSNE 没有 transform()
digits_tsne = tsne.fit_transform(digits.data)
plt.figure(figsize=(10, 10))
plt.xlim(digits_tsne[:, 0].min(), digits_tsne[:, 0].max() + 1)
plt.ylim(digits_tsne[:, 1].min(), digits_tsne[:, 1].max() + 1)
for i in range(len(digits.data)):
# 将数据绘制成文本图(代替散点图)
plt.text(digits_tsne[i, 0], digits_tsne[i, 1], str(digits.target[i]),
color = colors[digits.target[i]],
fontdict={'weight': 'bold', 'size': 9})
plt.xlabel("t-SNE feature 0")
plt.ylabel("t-SNE feature 1")

t-SNE的结果非常棒。所有类别都被明确分开。数字1和9被分成几块,但大多数类别都形成一个密集的组。要记住,这种方法并不知道类别标签:它完全是无监督的。但它能够找到数据的一种二维表示,仅根据原始空间中数据点之间的靠近程度就能够将各个类别明确分开。
参考
- Nimfa is a Python library for nonnegative matrix factorization:http://nimfa.biolab.si/
- Non-negative matrix factorization:https://en.wikipedia.org/wiki/Non-negative_matrix_factorization
- NMF 非负矩阵分解 – 原理与应用:https://blog.csdn.net/qq_26225295/article/details/51211529
- 非负矩阵分解NMF:https://blog.csdn.net/pipisorry/article/details/52098864
- 非负矩阵分解(NMF)迭代公式推导证明:https://zhuanlan.zhihu.com/p/340774022
- 非负矩阵分解(NMF)浅析:https://cloud.tencent.com/developer/article/2104422
- t-SNE:最好的降维方法之一:https://zhuanlan.zhihu.com/p/64664346