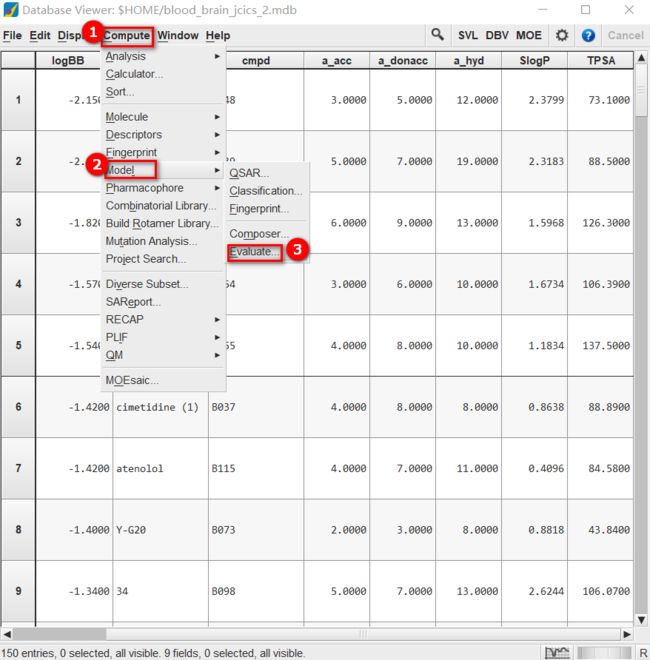
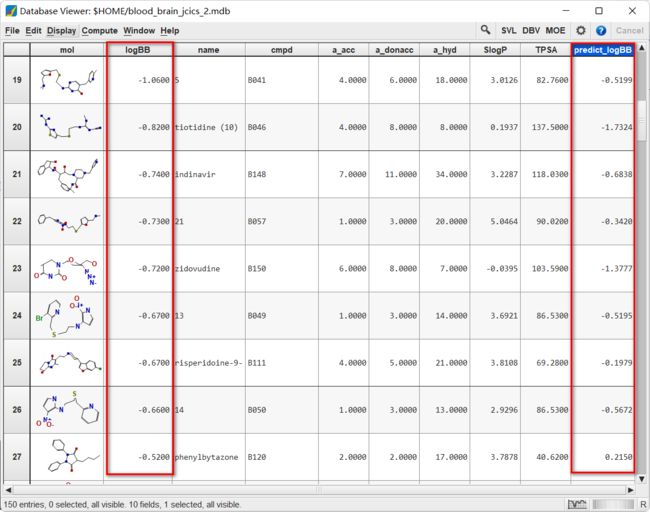
- 程序员的技术栈及学习路径
Honeysea_70
基础知识学习经验分享笔记
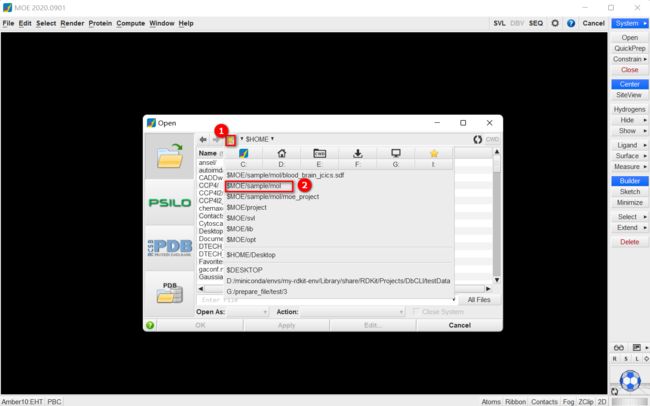
程序员的技术栈是非常多元的,通常涵盖了多个领域和技术。程序员的技术栈通常根据工作需求、项目类型以及个人兴趣的不同而有所不同,但通常会有一定的共性。下面是一个较为典型的程序员的技术栈,以及如何从入门到进阶地学习这些技术。1.编程语言掌握多种编程语言一个资深程序员通常会熟练掌握至少两到三种编程语言,每种语言的侧重点不同,适用于不同的开发场景。主流编程语言:JavaScript:前端开发的核心语言,Re
- 读《认知天性》(1)
云城梦天
认知是对天性是挑战认知可以用数学统计与实践客观来评价,而我感觉是一种自我感知。当未知时,感知痛苦然而这是个时习之中乐的过程。也可以通过rain和轻疗的方法安抚情绪编码,可以以好奇心与视觉画面联动来做记忆编码的过程,因人是视觉性爬行动物,且好奇心也是人的天性好奇时会主动探索算是翻转式学习的一种,编码是记忆过程。另外你可能对记忆中某一刻的感觉记得很清楚,然而忘记了内容,人或许也是感觉爬行动物。巩固,可
- WSL介绍
bigleek
树莓派pico开发linux
WSL(WindowsSubsystemforLinux)是微软为Windows系统开发的一项功能,允许用户在Windows上直接运行原生的Linux环境,而无需传统的虚拟机或双系统。它通过轻量化技术将Linux工具、命令行和应用程序无缝集成到Windows中,是开发者、运维人员和Linux学习者的高效工具。一、WSL的核心特点1.无需虚拟机:直接在Windows中运行Linux二进制文件(如Ba
- 操作系统试验三:观察Linux进程/线程的异步并发执行
只会打孔的磁带
linuxc++c语言
一、实验目的通过本实验学习如何创建Linux进程及线程,通过实验,观察Linux进程及线程的异步执行。理解进程及线程的区别及特性,进一步理解进程是资源分配单位,线程是独立调度单位。二、实验环境硬件环境:计算机一台,局域网环境;软件环境:LinuxUbuntu操作系统,gcc编译器。三、实验内容和步骤1、进程异步并发执行(1)编写一个C语言程序,该程序首先初始化一个count变量为1,然后使用for
- 【王老师每日家庭教育分享】8种方法清除孩子内心的自卑感——印刷阴影法
王继轩24
失败的阴影是产生自卑的温床。有自卑心理的孩子遇到挫折与失败比一般孩子要多得多,及时洗刷失败的阴影是克服自卑、保持自信的重要手段。洗刷失败阴影的方法很多,较为常见的有两种是家长要帮助孩子将失败当作学习的机遇,认真分析失败的原因,从失败中学习和吸取教训,总结经验;二是彻底遗忘,家长要帮助孩子有意将那些不愉快的、痛苦的事彻底地忘记,或是用成功的经历去抵消失败的阴影。
- 网课感想
我的耗子跑掉了ei
这是我大学的第一个寒假,本来有好多计划的,没想到一场灾难从天而降,打乱了所有人的计划,开学也因此延期了。而我也实现了我曾经最想实现的梦想—在床上上课。高中时不知道说了多少次想把床搬到教室里上,没想到还真能在床上上课。说起网课我还是蛮期待的,从来没有上过,也很好奇,我朋友高三,她早就开始上课了,感觉挺轻松的,她们是在腾讯课堂里上,是直播,我们是在学习通里看老师们安排的课程,这和我想的有点出入,不过仔
- 开始写亲子日记了
闻韶三三中队亲子日记
图片发自App今天我下班回家,硕说就特别兴奋的和我说:“妈妈今天老师表扬我了,说我的作业写的比以前好了,我一定要再接再厉,写的更好”,听到他这么说,瞬间特别欣慰,孩子也需要不断的鼓励,不能总是批评,这是我需要改正的错误。今天看到班里的几个家长去参加了学校的活动,可惜没有时间没能到场,下次再有这样的机会,一定参加。互相学习成长。
- 自然的启示
陌上繁花江南落
文/杜丽敏自然神秘莫测,一花一草,一树一叶总代给人们无限遐想,引发无数文人热议;陶渊明说:木欣欣以向荣,泉涓涓而始流.,他从自然繁荣滋长的景象中感叹时光的流逝,人生的短暂.总之,人们从大自然中获得了许许多多的真理,给予人们生活,学习以重大的影响.从屋檐下滴落的,经过长年累月的拍打,能够穿破坚硬的石头;古人从这简单的自然现象中感悟到了一个道理:做事要有持之以恒,坚持不懈的精神,不要轻易放弃;从月的圆
- 知行格英雄之旅57天
好的_6022
阅读:《彷徨之刃》健身:散步日收获与内省:今天听了一个学长的分享,自己也产生了一些想法。我还是想在新媒体运营编辑方面更加深入的了解,希望自己能找一个这方面的实习先了解一下看自己是不是想以后做此类工作。对于考研真的下不定决心,觉得自己不能认真的学习,但是又对自己的学历为以后就业而感到担心。明日三件事:1.百词斩打卡2.学六级3.学韩语知行格,培育独立之人格,自由之精神
- 2018-05-23
poppy彭
六项精进2018-5-23姓名:彭雪公司:广汉油脂354期学员379期志工【日精进打卡第106天】【知~学习】《六项精进》0遍共154遍《大学》0遍共134遍《领导者的十项资质》遍【经典句分享】【行~实践】一、修身:1,第8天二、齐家:1,三、建功:1,配合检查{积善}:发愿从2018年1月28日起两年内日行一善事。今日1善,总计142善。【省~觉悟】1,态度,格局2,【感谢】1,谢谢妈妈的大度,
- 犀水家族办公室理财入门课程七:为什么我的基金一买进去就亏?
犀水家族办公室
嗨大家好,老宋又来了,今天我们聊得深入一点,我们只聊一个问题,为什么你的基金一买就亏。很多小伙伴很爱学习,又是买书又是百度又是看视频,学习了很多筛选基金的办法,这指标那指标,什么三个月、半年、三年表现,阿尔法、贝塔一起上,自己在电脑面前精挑细选了一只基金,满怀希望买进去,结果一买进去就蹭蹭蹭下跌?是我智商有问题?其实乱买基金就是自己往火坑里面跳!什么?个股到处是坑,基金也有坑?大了去了,以前没听说
- 0基础学画画(瓢虫),连载82/100天
大鱼漫说
大家好,我是大鱼漫说,你们可以叫我大鱼,我现在是一位零基础学习绘画的程序员。每日一问~小鱼问:我每次用procreate临摹的时候,颜色用不准,感觉和作者的还是有区别,请问您是怎么临摹颜色的?大鱼答:这有两种方法,第一种,也是不是很了解软件人的用法,右上角小扳手工具,添加你要临摹的图片,每次涂颜色的时候都去原图上吸取颜色,大鱼刚开始也这样。第二种,就是新建调色板,具体步骤是,打开色盘,选中最右边的
- VSCode使用Jupyter完整指南配置机器学习环境
z日火
校招学习日记vscodejupyter机器学习
接下来开始机器学习部分第一步配置环境:VSCode使用Jupyter完整指南1.安装必要的扩展打开VSCode,按Ctrl+Shift+X打开扩展市场,搜索并安装以下扩展:必装扩展:Python(Microsoft官方)-Python语言支持Jupyter(Microsoft官方)-Jupyternotebook支持Pylance(Microsoft官方)-Python智能提示和语法检查推荐扩展:
- 阅读法记录《如何给孩子读绘本》
Vigar0510
今天体检排队等做彩超的超长“间隙”正好适合读这样小巧的一本书,一打开书发现作者松居直先生竟然是位1926年的长者,感慨一下日本绘本阅读真是领先中国好多年。开始吸引我买这本书的是封面的设计,没有很特别,但就是第一眼就喜欢了,结果读到结尾发现是自己喜欢的书籍装帧设计师杉浦康平先生设计的,是哒,眼缘这个事儿,就是没办法解释。这里就这本书试着学习老师的读书方法进行分析。从目录看,第一章讲述“绘本为何重要”
- 好的判断力
半糖私语
今天天气很好,,可以说是超级棒哦⊙∀⊙!,,奈何在家里,不过,也没什么关系,我今天,不,还有昨天,,还有前天,感觉自己啥也没做,,就是一味的追剧,,药理,病生留了好多白,,还是要好好学习,,不能荒废学业呀!不管怎么样,不学习,,没有起来,今天听了一句话,,知识就是力量,,学知识,,是为了更好的判断,有自己的判断力,所以要努力学习。
- 25数据库三级备考自整理笔记
备考策略:博主是边做题边学习知识点的,从每个章节->每套真题的流程,知识点清晰详细,喜欢的请点个关注和收藏,祝大家考试顺利,必过必过必过!一、数据库应用系统开发方法1.数据库的三级模式:外模式、模式、内模式。外->是数据库用户(包括应用程序员和最终用户看见和使用的局部数据的逻辑结构和特征的描述,是数据库用户的视图,是某一应用有关的数据的逻辑表示;外模式是模式的子集,一个数据库可以有多个外模式);(
- 如何调整优化器的参数来优化神经网络性能?
Idividuals
深度学习神经网络机器学习pythonscikit-learn
不同优化器有不同的可调整参数,下面以常见的优化器为例,讲解如何调整其参数来优化神经网络性能:Adam优化器Adam优化器有几个关键参数:learning_rate(学习率)、beta_1、beta_2和epsilon。1.学习率(learning_rate)-作用:控制每次参数更新的步长。学习率过大,模型可能无法收敛,在最优解附近振荡甚至发散;学习率过小,训练速度会非常缓慢。-调整方法:通常初始值
- 2018-08-18
子分小
姓名:张颖公司:青岛博厚医疗管理股份有限公司【反省总结第40天,始于20180709今天是201808018】【知~学习】六项精背诵3遍大学背诵1遍【行~实践】一、修身:(对自己个人)早起做了颈椎运动二、齐家:(对家庭和家人)暂无三、建功:(对工作)石老人门店证照问题{积善}:发愿从2018年7月9日起1年内龙365善事。今日0善,累计29善。【省~觉悟】找准方向,做自己喜欢的事,并坚持到底,努力
- 养老院管理系统基于SpringBoot的养老院管理系统系统设计与实现(源码+论文+部署讲解等)
博主介绍:✌全网粉丝60W+,csdn特邀作者、Java领域优质创作者、csdn/掘金/哔哩哔哩/知乎/道客/小红书等平台优质作者,计算机毕设实战导师,目前专注于大学生项目实战开发,讲解,毕业答疑辅导,欢迎高校老师/同行前辈交流合作✌技术栈范围:SpringBoot、Vue、SSM、Jsp、HLMT、Nodejs、Python、爬虫、数据可视化、小程序、安卓app、大数据、物联网、机器学习、单片机
- 《学习之道》读后感
薯片_chips
说起这本书,就不得不说下这本书的作者,乔希维茨金,他少年时曾8次在全国象棋冠军赛中夺魁,13岁获得象棋大师头衔,在纵横西方棋坛十年后,那时他才22岁,在那一年他开始研习太极拳,并连续21次赢得全美太极拳冠军及世界冠军头衔。上面的一段话我基本就是照搬书本扉页的介绍的,书中我看到的是作者对于他象棋和太极生涯中无数个困难的思考和解答,以及他是怎么克服障碍的,不论这个障碍是他的内心,外物干扰,周围的人,亦
- TCP 粘包/拆包的原因及解决方法
青城楼主
avaNIO学习时,发现,如果客户端连续不断的向服务端发送数据包时,服务端接收的数据会出现两个数据包粘在一起的情况,这就是TCP协议中经常会遇到的粘包以及拆包的问题。发生TCP粘包或拆包有很多原因,现列出常见的几点,可能不全面,欢迎补充,1、要发送的数据大于TCP发送缓冲区剩余空间大小,将会发生拆包。2、待发送数据大于MSS(最大报文长度),TCP在传输前将进行拆包。3、要发送的数据小于TCP发送
- 解决引入TransXNet模块后显存爆炸问题的全面指南
pk_xz123456
算法大数据python机器人数据挖掘深度学习
解决引入TransXNet模块后显存爆炸问题的全面指南前些天发现了一个巨牛的人工智能学习网站,通俗易懂,风趣幽默,忍不住分享一下给大家。点击跳转到网站。1.问题背景与现状分析1.1MF-PSN和TransXNet项目概述MF-PSN(Multi-FeaturePyramidStereoNetwork)是一个基于金字塔特征的多特征立体匹配网络,它通过构建多层次的特征金字塔来处理不同尺度的立体匹配问题
- 探索名门望族背后的秘密
期待遇见最好的自己
20200824读书笔记(十七)朱熹家族186节朱熹家族。一个人是如何成人,成才,成事的?想搞懂自己与他人。有一天,我发现搞懂自己与他人还是不够的,还要搞懂人生,当我去探索人生的时候,我是否进入了一个全新的世界,不是为了学习历史,而是需要探索东方智慧。不是为了探索东方智慧,而是为了寻找人生答案。朱熹是一个从天理之人,从小想搞懂天下万物。把道和理都要研究透。因此成为了我要重点研究的对象之一。带着朱熹
- 【有所思】凡天下事,三规定天下
金钟山人
2022年6月30日周四曲靖晴终于下定决心学习书法了,这是认真的,交了费,拜了师,摆好桌,选好贴,准备至少半年的纸和笔,做足功夫。刘老师帮我分析,确定从魏碑练习开始,要求放空忘记过去,照着写,放空自己,不发挥不走样。老师还告诉我“凡天下事,三规定天下”,我也觉得持有道理。何为三规,曰规律,曰规则,曰规划。试分析如下:一、找规律世间万事万物大到宇宙运行的规律,中到人性的规律,小到孩子不同心理发展阶段
- 《洞见》读后感
杨慧裕
《洞见》把认知升级的过程总结为“认知闭环”:选择学习认知——践行验证认知——调整迭代认知:第一步,选择已被验证的认知,学习之。这就是一个学习知识的过程,关键要分清的是,哪些是值得学习的认知。将这个点应用在习惯养成上面,在养成习惯之前,一定要选择适合自己的方法,不要盲目跟风,就像别人四点起床阅读,呢没分清自己的学习体制,就跟风四点起来阅读,最后早起毁一天,书也没读好,人还迷迷糊糊一天什么也做不好。第
- Java高并发编程核心:并发集合与原子类详解
msbQQ
java开发语言后端并发编程
在当今高并发、高吞吐的分布式系统中,Java并发编程已成为开发者必备的核心能力。当线程如潮水般涌来,如何确保数据安全?如何避免死锁陷阱?如何实现无阻塞的高效运算?答案就隐藏在并发集合与原子类这两大基石之中。1.并发集合:线程安全的容器1.1ConcurrentHashMap我在最开始学习这个容器的时候当时会记住它的特点是:线程安全,允许多个线程进行读和写。null值和键:ConcurrentHas
- 2018-03-29
XuelinW
今天去cambrils上课,孩子们都很可爱。可是不怎么喜欢学习中文。不过感谢他们好几年的坚持,尽管自己不想牺牲玩儿的时间来学习现在看似没用的汉语。尽管他们都是被家长逼着过来的,只要来,就有进步!感谢家长提供上课场所。感谢奶奶邀请儿子去她家吃饭,感谢她对我的帮助。感谢老公每天记得打电话给我。感谢他让我超越过去的混沌。感谢儿子的自立,感谢儿子的自律,他在运动,做人家妈妈的总不好坐着看电视,总不好无节制
- 【大语言模型基础】GPT(Generative Pre-training )生成式无监督预训练模型原理
前言ELMo:将上下文当作特征,但是无监督的语料和我们真实的语料还是有区别的,不一定符合我们特定的任务,是一种双向的特征提取。OpenAIGPT:通过transformerdecoder学习出来一个语言模型,不是固定的,通过任务fine-tuning,用transfomer代替ELMo的LSTM。OpenAIGPT其实就是缺少了encoder的transformer:当然也没了encoder与de
- GPT-4o mini TTS:领先的文本转语音技术
桂花饼
AIGCGPT-4oo4-mini语音识别人工智能GPT-4o
什么是GPT-4ominiTTS?GPT-4ominiTTS是OpenAI推出的全新一代文本转语音(TTS)技术,能够以自然、流畅的方式将普通文本转换为语音。依托先进的神经网络架构,GPT-4ominiTTS在语音合成中避免了传统TTS的生硬与机械感,能够生成富有情感和个性化表达的高质量语音。该技术支持多语言与多口音,是视频、播客、电子学习等场景的理想选择。核心特点自然流畅,接近真人GPT-4om
- AI产品经理成长记《零号列车》第一集 邂逅0XAI列车
黑客思维者
AI产品经理养成人工智能AI产品经理大模型智能体
《零号列车》绝非传统意义上的AI产品经理教程——它是我沉淀二十多年跨行业数字化转型与工业4.0实战经验后,首创的100集大型小说体培养指南。那些曾在千行百业验证过的知识与经验,不再是枯燥的文字堆砌,而是化作一场沉浸式的学习旅程。这里没有生硬的理论灌输,而是用跌宕起伏的故事情节,串联起AI技术的底层逻辑。你会跟着角色的脚步推进剧情,在不知不觉中吃透机器学习、大模型应用等专业概念;更有深入浅出的技术拆
- 插入表主键冲突做更新
a-john
有以下场景:
用户下了一个订单,订单内的内容较多,且来自多表,首次下单的时候,内容可能会不全(部分内容不是必须,出现有些表根本就没有没有该订单的值)。在以后更改订单时,有些内容会更改,有些内容会新增。
问题:
如果在sql语句中执行update操作,在没有数据的表中会出错。如果在逻辑代码中先做查询,查询结果有做更新,没有做插入,这样会将代码复杂化。
解决:
mysql中提供了一个sql语
- Android xml资源文件中@、@android:type、@*、?、@+含义和区别
Cb123456
@+@?@*
一.@代表引用资源
1.引用自定义资源。格式:@[package:]type/name
android:text="@string/hello"
2.引用系统资源。格式:@android:type/name
android:textColor="@android:color/opaque_red"
- 数据结构的基本介绍
天子之骄
数据结构散列表树、图线性结构价格标签
数据结构的基本介绍
数据结构就是数据的组织形式,用一种提前设计好的框架去存取数据,以便更方便,高效的对数据进行增删查改。正确选择合适的数据结构,对软件程序的高效执行的影响作用不亚于算法的设计。此外,在计算机系统中数据结构的作用也是非同小可。例如常常在编程语言中听到的栈,堆等,就是经典的数据结构。
经典的数据结构大致如下:
一:线性数据结构
(1):列表
a
- 通过二维码开放平台的API快速生成二维码
一炮送你回车库
api
现在很多网站都有通过扫二维码用手机连接的功能,联图网(http://www.liantu.com/pingtai/)的二维码开放平台开放了一个生成二维码图片的Api,挺方便使用的。闲着无聊,写了个前台快速生成二维码的方法。
html代码如下:(二维码将生成在这div下)
? 1
&nbs
- ImageIO读取一张图片改变大小
3213213333332132
javaIOimageBufferedImage
package com.demo;
import java.awt.image.BufferedImage;
import java.io.File;
import java.io.IOException;
import javax.imageio.ImageIO;
/**
* @Description 读取一张图片改变大小
* @author FuJianyon
- myeclipse集成svn(一针见血)
7454103
eclipseSVNMyEclipse
&n
- 装箱与拆箱----autoboxing和unboxing
darkranger
J2SE
4.2 自动装箱和拆箱
基本数据(Primitive)类型的自动装箱(autoboxing)、拆箱(unboxing)是自J2SE 5.0开始提供的功能。虽然为您打包基本数据类型提供了方便,但提供方便的同时表示隐藏了细节,建议在能够区分基本数据类型与对象的差别时再使用。
4.2.1 autoboxing和unboxing
在Java中,所有要处理的东西几乎都是对象(Object)
- ajax传统的方式制作ajax
aijuans
Ajax
//这是前台的代码
<%@ page language="java" import="java.util.*" pageEncoding="UTF-8"%> <% String path = request.getContextPath(); String basePath = request.getScheme()+
- 只用jre的eclipse是怎么编译java源文件的?
avords
javaeclipsejdktomcat
eclipse只需要jre就可以运行开发java程序了,也能自动 编译java源代码,但是jre不是java的运行环境么,难道jre中也带有编译工具? 还是eclipse自己实现的?谁能给解释一下呢问题补充:假设系统中没有安装jdk or jre,只在eclipse的目录中有一个jre,那么eclipse会采用该jre,问题是eclipse照样可以编译java源文件,为什么呢?
&nb
- 前端模块化
bee1314
模块化
背景: 前端JavaScript模块化,其实已经不是什么新鲜事了。但是很多的项目还没有真正的使用起来,还处于刀耕火种的野蛮生长阶段。 JavaScript一直缺乏有效的包管理机制,造成了大量的全局变量,大量的方法冲突。我们多么渴望有天能像Java(import),Python (import),Ruby(require)那样写代码。在没有包管理机制的年代,我们是怎么避免所
- 处理百万级以上的数据处理
bijian1013
oraclesql数据库大数据查询
一.处理百万级以上的数据提高查询速度的方法: 1.应尽量避免在 where 子句中使用!=或<>操作符,否则将引擎放弃使用索引而进行全表扫描。
2.对查询进行优化,应尽量避免全表扫描,首先应考虑在 where 及 o
- mac 卸载 java 1.7 或更高版本
征客丶
javaOS
卸载 java 1.7 或更高
sudo rm -rf /Library/Internet\ Plug-Ins/JavaAppletPlugin.plugin
成功执行此命令后,还可以执行 java 与 javac 命令
sudo rm -rf /Library/PreferencePanes/JavaControlPanel.prefPane
成功执行此命令后,还可以执行 java
- 【Spark六十一】Spark Streaming结合Flume、Kafka进行日志分析
bit1129
Stream
第一步,Flume和Kakfa对接,Flume抓取日志,写到Kafka中
第二部,Spark Streaming读取Kafka中的数据,进行实时分析
本文首先使用Kakfa自带的消息处理(脚本)来获取消息,走通Flume和Kafka的对接 1. Flume配置
1. 下载Flume和Kafka集成的插件,下载地址:https://github.com/beyondj2ee/f
- Erlang vs TNSDL
bookjovi
erlang
TNSDL是Nokia内部用于开发电信交换软件的私有语言,是在SDL语言的基础上加以修改而成,TNSDL需翻译成C语言得以编译执行,TNSDL语言中实现了异步并行的特点,当然要完整实现异步并行还需要运行时动态库的支持,异步并行类似于Erlang的process(轻量级进程),TNSDL中则称之为hand,Erlang是基于vm(beam)开发,
- 非常希望有一个预防疲劳的java软件, 预防过劳死和眼睛疲劳,大家一起努力搞一个
ljy325
企业应用
非常希望有一个预防疲劳的java软件,我看新闻和网站,国防科技大学的科学家累死了,太疲劳,老是加班,不休息,经常吃药,吃药根本就没用,根本原因是疲劳过度。我以前做java,那会公司垃圾,老想赶快学习到东西跳槽离开,搞得超负荷,不明理。深圳做软件开发经常累死人,总有不明理的人,有个软件提醒限制很好,可以挽救很多人的生命。
相关新闻:
(1)IT行业成五大疾病重灾区:过劳死平均37.9岁
- 读《研磨设计模式》-代码笔记-原型模式
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
/**
* Effective Java 建议使用copy constructor or copy factory来代替clone()方法:
* 1.public Product copy(Product p){}
* 2.publi
- 配置管理---svn工具之权限配置
chenyu19891124
SVN
今天花了大半天的功夫,终于弄懂svn权限配置。下面是今天收获的战绩。
安装完svn后就是在svn中建立版本库,比如我本地的是版本库路径是C:\Repositories\pepos。pepos是我的版本库。在pepos的目录结构
pepos
component
webapps
在conf里面的auth里赋予的权限配置为
[groups]
- 浅谈程序员的数学修养
comsci
设计模式编程算法面试招聘
浅谈程序员的数学修养
- 批量执行 bulk collect与forall用法
daizj
oraclesqlbulk collectforall
BULK COLLECT 子句会批量检索结果,即一次性将结果集绑定到一个集合变量中,并从SQL引擎发送到PL/SQL引擎。通常可以在SELECT INTO、
FETCH INTO以及RETURNING INTO子句中使用BULK COLLECT。本文将逐一描述BULK COLLECT在这几种情形下的用法。
有关FORALL语句的用法请参考:批量SQL之 F
- Linux下使用rsync最快速删除海量文件的方法
dongwei_6688
OS
1、先安装rsync:yum install rsync
2、建立一个空的文件夹:mkdir /tmp/test
3、用rsync删除目标目录:rsync --delete-before -a -H -v --progress --stats /tmp/test/ log/这样我们要删除的log目录就会被清空了,删除的速度会非常快。rsync实际上用的是替换原理,处理数十万个文件也是秒删。
- Yii CModel中rules验证规格
dcj3sjt126com
rulesyiivalidate
Yii cValidator主要用法分析:
yii验证rulesit 分类: Yii yii的rules验证 cValidator主要属性 attributes ,builtInValidators,enableClientValidation,message,on,safe,skipOnError
- 基于vagrant的redis主从实验
dcj3sjt126com
vagrant
平台: Mac
工具: Vagrant
系统: Centos6.5
实验目的: Redis主从
实现思路
制作一个基于sentos6.5, 已经安装好reids的box, 添加一个脚本配置从机, 然后作为后面主机从机的基础box
制作sentos6.5+redis的box
mkdir vagrant_redis
cd vagrant_
- Memcached(二)、Centos安装Memcached服务器
frank1234
centosmemcached
一、安装gcc
rpm和yum安装memcached服务器连接没有找到,所以我使用的是make的方式安装,由于make依赖于gcc,所以要先安装gcc
开始安装,命令如下,[color=red][b]顺序一定不能出错[/b][/color]:
建议可以先切换到root用户,不然可能会遇到权限问题:su root 输入密码......
rpm -ivh kernel-head
- Remove Duplicates from Sorted List
hcx2013
remove
Given a sorted linked list, delete all duplicates such that each element appear only once.
For example,Given 1->1->2, return 1->2.Given 1->1->2->3->3, return&
- Spring4新特性——JSR310日期时间API的支持
jinnianshilongnian
spring4
Spring4新特性——泛型限定式依赖注入
Spring4新特性——核心容器的其他改进
Spring4新特性——Web开发的增强
Spring4新特性——集成Bean Validation 1.1(JSR-349)到SpringMVC
Spring4新特性——Groovy Bean定义DSL
Spring4新特性——更好的Java泛型操作API
Spring4新
- 浅谈enum与单例设计模式
247687009
java单例
在JDK1.5之前的单例实现方式有两种(懒汉式和饿汉式并无设计上的区别故看做一种),两者同是私有构
造器,导出静态成员变量,以便调用者访问。
第一种
package singleton;
public class Singleton {
//导出全局成员
public final static Singleton INSTANCE = new S
- 使用switch条件语句需要注意的几点
openwrt
cbreakswitch
1. 当满足条件的case中没有break,程序将依次执行其后的每种条件(包括default)直到遇到break跳出
int main()
{
int n = 1;
switch(n) {
case 1:
printf("--1--\n");
default:
printf("defa
- 配置Spring Mybatis JUnit测试环境的应用上下文
schnell18
springmybatisJUnit
Spring-test模块中的应用上下文和web及spring boot的有很大差异。主要试下来差异有:
单元测试的app context不支持从外部properties文件注入属性
@Value注解不能解析带通配符的路径字符串
解决第一个问题可以配置一个PropertyPlaceholderConfigurer的bean。
第二个问题的具体实例是:
- Java 定时任务总结一
tuoni
javaspringtimerquartztimertask
Java定时任务总结 一.从技术上分类大概分为以下三种方式: 1.Java自带的java.util.Timer类,这个类允许你调度一个java.util.TimerTask任务; 说明: java.util.Timer定时器,实际上是个线程,定时执行TimerTask类 &
- 一种防止用户生成内容站点出现商业广告以及非法有害等垃圾信息的方法
yangshangchuan
rank相似度计算文本相似度词袋模型余弦相似度
本文描述了一种在ITEYE博客频道上面出现的新型的商业广告形式及其应对方法,对于其他的用户生成内容站点类型也具有同样的适用性。
最近在ITEYE博客频道上面出现了一种新型的商业广告形式,方法如下:
1、注册多个账号(一般10个以上)。
2、从多个账号中选择一个账号,发表1-2篇博文