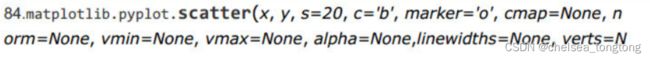机器学习Sklearn——核函数、核函数在不同数据集上的表现、核函数的优势和缺点
目录
1 核函数
2 Sklearn中理解核函数
2.1 重要参数kernel
2.2 探索核函数在不同数据集上的表现
2.2.1 导入所需要的库和模块
2.2.2 创建数据集,定义核函数的选择
2.2.3构建一行4列子图
2.2.4 构建子图
2.2.5 进行子图循环
3、探索核函数的优势和缺陷
1 核函数
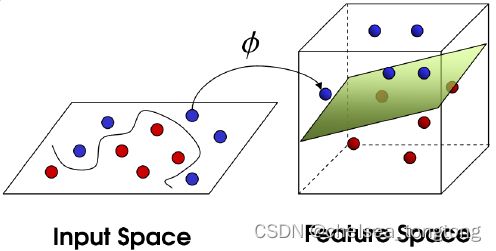
核函数的功能就是从低维度的空间向高维度空间转换的函数, 因为在低维空间不可以线性分类的问题到高维度空间就可以线性分类的。 其求法为 低维空间的点映射到高维空间的点后,两个点的内积 就是核函数。
核函数粗浅的理解 - 知乎
核函数的入门理解还是要从,将二维非线性问题转化为三维线性问题。
小结:通过增加维度的方法来实现从非线性可分到线性可分。
2 Sklearn中理解核函数
2.1 重要参数kernel
在sklearn中参数kernel有以下几种选项
| 输入 | 含义 | 解决问题 | 核函数表达式 | 参数 gamma |
参数 degree |
参数 coef0 |
| ‘linear’ | 线性核 | 线性 | K(x,y)=xTy=x⋅y | NO | NO | NO |
| ‘poly’ | 多项式核 | 偏线性 | K(x,y)=(γ(x⋅y)+r)d | YES | YES | YES |
| "sigmoid" | 双曲正切核 | 非线性 | K(x,y)=tanh(γ(x⋅y)+r) | YES | NO | YES |
| ‘rbf | 高斯径向基 | 偏非线性 | K(x,y)=e−γ(x−y)2,γ>0 | NO | NO | NO |
可以看出,除了选项"linear"之外,其他核函数都可以处理非线性问题。多项式核函数有次数d,当d为1的时候它就是再处理线性问题,当d为更高次项的时候它就是在处理非线性问题。
2.2 探索核函数在不同数据集上的表现
我们可以通过在不同的核函数中循环去找寻最佳的核函数来对核函数进行一个选取。
接下来通过一个例子,来探索一下不同数据集上核函数的表现。我们现在有一系列线性或非线性可分的数据,我们希望通过绘制SVC在不同核函数下的决策边界并计算SVC在不同核函数下分类准确率来观察核函数的效用。
2.2.1 导入所需要的库和模块
- make_bolbs生成聚类点,make_circles生成环形数据,make_moons生成月牙形数据,make_classification生成分类数据
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs,make_circles,make_moons,make_classification
from sklearn.svm import SVC2.2.2 创建数据集,定义核函数的选择
- make_classification参数:
n_informative=, 有效特征个数
n_redundant=, 冗余特征个数(有效特征的随机组合)
n_repeated=, 重复特征个数(有效特征和冗余特征的随机组合)
n_classes=, 样本类别
n_clusters_per_class=, 簇的个数
n_samples = 300
datasets = [
make_blobs(n_samples = n_samples,centers = 2,random_state =1)
,make_circles(n_samples = n_samples,noise = 0.2,factor = 0.5,random_state=1)
,make_moons(n_samples = n_samples,noise = 0.2,random_state=0)
,make_classification(n_samples = n_samples,n_features=2,n_informative=2,n_redundant=0,random_state=1)
]
Kernel=["linear","poly","rbf","sigmoid"]2.2.3构建一行4列子图
#创建一行四列子图
fig,axes = plt.subplots(nrows=1,ncols=4,figsize=(20,4))
for i,(X,Y) in enumerate(datasets):
ax = axes[i]
ax.scatter(X[:,0],X[:,1],cmap='rainbow',c=Y)
ax.set_xticks([])
ax.set_yticks([])
2.2.4 构建子图
- plt.subplot()用来构建子图
nrows=len(datasets)
ncols=len(Kernel)+1
fig,axes=plt.subplots(nrows,ncols,figsize=(20,16))
注:为了好截图我将figsize调整成了(15,4),不然太大大了。
2.2.5 进行子图循环
- list(enumerate(datasets))==[*enumerate(datasets)]
- [*enumerate(datasets)]#map映射,enumerate遍历索引及列表元素,zip列表合并
#第一层循环:在不同的数据集中循环
for ds_cnt, (X,Y) in enumerate(datasets):
#在图像中的第一列,第一个,放置原数据的分布
#zorder=10表示画布的层级,散点图最优先,edgecolors表示边缘额颜色
ax = axes[ds_cnt, 0]
if ds_cnt == 0:
ax.set_title("Input data")
ax.scatter(X[:, 0], X[:, 1], c=Y, zorder=10, cmap=plt.cm.Paired,edgecolors='k')
ax.set_xticks(())
ax.set_yticks(())#输入空的元组和空的列表是一样的
#第二层循环:在不同的核函数中循环
#从图像的第二列开始,一个个填充分类结果
for est_idx, kernel in enumerate(Kernel):
#定义子图位置,从第一列,第二个开始
ax = axes[ds_cnt, est_idx + 1]
#建模
clf = SVC(kernel=kernel, gamma=2).fit(X, Y)
score = clf.score(X, Y)
#绘制图像本身分布的散点图
ax.scatter(X[:, 0], X[:, 1], c=Y
,zorder=10
,cmap=plt.cm.Paired,edgecolors='k')
#绘制支持向量
ax.scatter(clf.support_vectors_[:, 0], clf.support_vectors_[:, 1], s=50,
facecolors='none', zorder=10, edgecolors='k')
#绘制决策边界
x_min, x_max = X[:, 0].min() - .5, X[:, 0].max() + .5
y_min, y_max = X[:, 1].min() - .5, X[:, 1].max() + .5
#np.mgrid,合并了我们之前使用的np.linspace和np.meshgrid的用法
#一次性使用最大值和最小值来生成网格
#表示为[起始值:结束值:步长]
#如果步长是复数,则其整数部分就是起始值和结束值之间创建的点的数量,并且结束值被包含在内
XX, YY = np.mgrid[x_min:x_max:200j, y_min:y_max:200j]
#np.c_,类似于np.vstack的功能
Z = clf.decision_function(np.c_[XX.ravel(), YY.ravel()]).reshape(XX.shape)
#填充等高线不同区域的颜色
ax.pcolormesh(XX, YY, Z > 0, cmap=plt.cm.Paired)
#绘制等高线
ax.contour(XX, YY, Z, colors=['k', 'k', 'k'], linestyles=['--', '-', '--'],
levels=[-1, 0, 1])
#设定坐标轴为不显示
ax.set_xticks(())
ax.set_yticks(())
#将标题放在第一行的顶上
if ds_cnt == 0:
ax.set_title(kernel)
#为每张图添加分类的分数
ax.text(0.95, 0.06, ('%.2f' % score).lstrip('0')
, size=15
, bbox=dict(boxstyle='round', alpha=0.8, facecolor='white')
#为分数添加一个白色的格子作为底色
, transform=ax.transAxes #确定文字所对应的坐标轴,就是ax子图的坐标轴本身
, horizontalalignment='right' #位于坐标轴的什么方向
)
plt.tight_layout()
plt.show()环形数据和月牙形适合用rbf进行分类(决策树能达到接近rbf),混合的数据每个分类都不是很好。
总结:高斯径向基rbf核函数效果特别好,线性和非线性都适用,最常用。
3、探索核函数的优势和缺陷
看起来,除了Sigmoid核函数,其他核函数效果都不错。但其实rbf和poly也有自己的弊端没我们用乳腺癌数据集作为例子展示一下。
- from time import time
import datetime 时间模块,将时间戳转化为现在的时间 - 导入时间戳timestamp,循环的开端time0 = time(),循环的末尾datetime.datetime.fromtimestamp可以计算运行的时间。
- degree在poly里面默认是3,degree=1说明是线性的
- SVC参数解释_Pg-Man的博客-CSDN博客_svc参数
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
from sklearn.svm import SVC
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from time import time
import datetime
data = load_breast_cancer()
kernel = ['linear','poly','sigmoid','rbf']
X = data.data
y = data.target
Xtrain,Xtest,Ytrain,Ytest = train_test_split(X,y,test_size = 0.3)
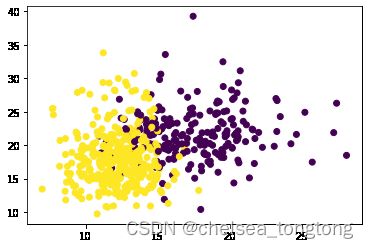
plt.scatter(data.data[:,0],data.data[:,1],c=data.target)
for kernel in kernel:
time0 = time()
clf = SVC(kernel = kernel
,gamma = 'auto'
,degree = 1
,cache_size = 500 #MB,可以使用的计算内存
).fit(Xtrain,Ytrain)
score = clf.score(Xtest,Ytest)
print('The accuracy under kernel {} is {:.2f}%'.format(kernel,score*100))#{}表示字符串, {:.2f}表示浮点数
print(datetime.datetime.fromtimestamp(time()-time0).strftime("%M:%S:%f"))
The accuracy under kernel linear is 94.74%
00:00:803546
The accuracy under kernel poly is 95.91%
00:00:052964
The accuracy under kernel sigmoid is 60.82%
00:00:007996
The accuracy under kernel rbf is 60.82%
00:00:026003
乳腺癌数据集有30各特征,取出前两列的特征可以看到这是一个相互交叠但线性可分的数据。线性核函数跑出来的效果很好。rbf和sigmoid两个擅长非线性的数据从效果上来看完全不可用。其次,线性核函数的运行速度远远不如非线性的两个核函数。
但是,之前我们了解说,rbf在线性数据上也可以表现得非常好,那在这里,为什么跑出来的结果如此糟糕呢?其实,这里真正的问题是数据的量纲问题。回忆一下我们如何求解决策边界,如何判断点是否在决策边界的一边?是靠计算”距离“,虽然我们不能说SVM是完全的距离类模型,但是它严重受到数据量纲的影响。
乳腺癌数据存在严重的量纲不一的问题。我们来使用数据预处理中的标准化的类,对数据进行标准化,然后再看看模型的结果:
from sklearn.preprocessing import StandardScaler
X = StandardScaler().fit_transform(X)
kernel = ['linear','poly','sigmoid','rbf']
Xtrain,Xtest,Ytrain,Ytest = train_test_split(X,y,test_size = 0.3)
for kernel in kernel:
time0 = time()
clf = SVC(kernel = kernel
,gamma = 'auto'
,degree = 1
,cache_size = 2000 #MB,可以使用的计算内存
).fit(Xtrain,Ytrain)
score = clf.score(Xtest,Ytest)
print('The accuracy under kernel {} is {:.2f}%'.format(kernel,score*100))
print(datetime.datetime.fromtimestamp(time()-time0).strftime("%M:%S:%f"))
The accuracy under kernel linear is 97.08%
00:00:004997
The accuracy under kernel poly is 96.49%
00:00:004998
The accuracy under kernel sigmoid is 95.91%
00:00:004995
The accuracy under kernel rbf is 97.08%
00:00:006995
量纲统一之后,可以观察到,所有核函数的运算时间都大大地减少了,尤其是对于线性核来说,而多项式核函数居然变成了计算最快的。其次,rbf表现出了非常优秀的结果。经过我们的探索,我们可以得到的结论是:
- 线性核,尤其是多项式核函数在高次项时计算非常缓慢
- rbf和多项式核函数都不擅长处理量纲不统一的数据集
幸运的是,这两个缺点都可以由数据无量纲化来解决。因此,SVM执行之前,非常推荐先进行数据的无量纲化!