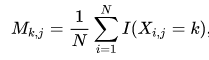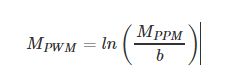- 生物信息学工作流(Bioinformatics Workflow):概念、历史、现状与展望?
lisw05
生物信息学生物信息学工作流
李升伟整理1.引言生物信息学工作流是指通过一系列计算步骤和工具,对生物学数据进行处理、分析和解释的系统化流程。随着高通量测序技术的普及和生物数据的爆炸式增长,生物信息学工作流在基因组学、转录组学、蛋白质组学等领域中扮演着至关重要的角色。它不仅提高了数据分析的效率,还为生命科学研究提供了新的视角和方法。2.生物信息学工作流的概念生物信息学工作流的核心是将复杂的生物学数据分析任务分解为多个可管理的步骤
- LM_Funny-2-01 递推算法:从数学基础到跨学科应用
王旭·wangxu_a
算法
目录第一章递推算法的数学本质1.1形式化定义与公理化体系定理1.1(完备性条件)1.2高阶递推的特征分析案例:Gauss同余递推4第二章工程实现优化技术2.1内存压缩的革新方法滚动窗口策略分块存储技术2.2异构计算加速方案GPU并行递推量子计算原型第三章跨学科应用案例3.1密码学中的递推构造混沌流密码系统3.2生物信息学的序列分析DNA甲基化预测第一章递推算法的数学本质1.1形式化定义与公理化体系
- R语言安装生物信息数据库包
Bio Coder
R语言r语言数据库
R语言安装生物信息数据库包在生物信息学领域,R语言是重要的数据分析工具。今天,我们就来聊聊在R语言环境下,安装生物信息数据库包(org.*.*.db)的步骤。为什么要安装org.*.*.db系列包生物信息学分析中,我们常处理基因相关数据,比如基因功能注释、位置、参与的生物学通路等。org.*.*.db系列包就像基因百科全书,提供不同物种的基因注释信息。比如研究人类基因时,能帮我们快速获取基因别名、
- 基于python使用scanpy分析单细胞转录组数据
探序基因
单细胞分析python开发语言
探序基因肿瘤研究院整理相关后缀的格式介绍:.h5ad:是一种用于存储单细胞数据的文件格式,可以通过anndata库在Python中处理.loom:高效的数据存储格式(.loom文件),使得用户可以轻松地存储、查询和分析大规模的单细胞数据集。Loompy的设计目标是提供一个快速、灵活且易于使用的工具,以支持生物信息学家和研究人员在单细胞水平上进行数据分析。python的单细胞转录组数据结构说明:da
- Perl 语言入门学习指南:探索高效脚本编程的奥秘
我的运维人生
简约运维perlPerl编程脚本语言文本处理Perl基础语法
引言Perl,全称PracticalExtractionandReportLanguage,是一种功能强大的编程语言,特别擅长于文本处理、报告生成以及系统自动化管理任务。自1987年诞生以来,Perl凭借其灵活性、强大的内置功能库和广泛的社区支持,在Web开发、生物信息学、网络管理等多个领域发挥着重要作用。本文旨在为初学者提供一份Perl语言入门学习指南,帮助大家快速掌握这门强大的脚本语言。一、P
- gseapy python包GO、KEGG富集(注释)分析
loong_XL
生信pythongolang开发语言
文档案例参考:https://gseapy.readthedocs.io/en/latest/gseapy_example.html#Over-representation-analysis-by-Enrichr-web-services简介:富集分析是一种常见的生物信息学分析方法,通过比较一个给定的基因集(如一组显著差异表达基因)与已知的生物过程、通路或功能的数据库,来发现哪些过程、通路或功能与
- Java 大视界 -- Java 大数据在生物信息学中的应用与挑战(67)
青云交
大数据新视界Java大视界大数据生物信息学基因序列分析蛋白质结构预测数据质量计算资源机器学习
亲爱的朋友们,热烈欢迎来到青云交的博客!能与诸位在此相逢,我倍感荣幸。在这飞速更迭的时代,我们都渴望一方心灵净土,而我的博客正是这样温暖的所在。这里为你呈上趣味与实用兼具的知识,也期待你毫无保留地分享独特见解,愿我们于此携手成长,共赴新程!一、欢迎加入【福利社群】点击快速加入:青云交灵犀技韵交响盛汇福利社群点击快速加入2:2024CSDN博客之星创作交流营(NEW)二、本博客的精华专栏:大数据新视
- 三甲医院大型生信服务器多配置方案剖析与应用(2024版)
Allen_LVyingbo
数智化医院2024服务器数据库运维
一、引言1.1研究背景与意义在当今数智化时代,生物信息学作为一门融合生物学、计算机科学和信息技术的交叉学科,在三甲医院的科研和临床应用中占据着举足轻重的地位。随着高通量测序技术、医学影像技术等的飞速发展,生物医学数据呈爆发式增长,这些数据涵盖了基因组、蛋白质组、代谢组等多个层面的信息,为医学研究和临床诊断提供了前所未有的机遇与挑战。从科研角度来看,生物信息学助力三甲医院开展前沿性的医学研究。通过对
- Python3 【集合】项目实战:3 个新颖的学习案例
李智 - 重庆
Python精讲精练-从入门到实战python经验分享案例学习编程技巧
Python3【集合】项目实战:3个新颖的学习案例以下是3个应用“Python集合”知识的综合应用项目,这些项目具有新颖性、前瞻性和实用性,每个项目都包含完整的代码、解释说明、测试案例和执行结果。基因序列比对文章推荐系统运行日志分析项目1:基因序列比对(集合运算与去重)项目描述在生物信息学中,比对两个基因序列的相似性。使用集合的交集和并集计算相似度。代码实现#基因序列(简化为字符串集合)seque
- AI人工智能深度学习算法:在生物信息学中的应用
AI大模型应用之禅
AI大模型与大数据计算科学神经计算深度学习神经网络大数据人工智能大型语言模型AIAGILLMJavaPython架构设计AgentRPA
AI人工智能深度学习算法:在生物信息学中的应用关键词:人工智能、深度学习、生物信息学、基因组学、蛋白质结构预测、药物发现、个性化医疗文章目录AI人工智能深度学习算法:在生物信息学中的应用1.背景介绍2.核心概念与联系2.1人工智能(AI)2.2机器学习(ML)2.3深度学习(DL)2.4生物信息学2.5应用领域3.核心算法原理&具体操作步骤3.1算法原理概述3.1.1卷积神经网络(CNN)3.1.
- Spark GraphX原理与代码实例讲解
AI大模型应用之禅
AI大模型与大数据计算科学神经计算深度学习神经网络大数据人工智能大型语言模型AIAGILLMJavaPython架构设计AgentRPA
SparkGraphX原理与代码实例讲解作者:禅与计算机程序设计艺术/ZenandtheArtofComputerProgramming1.背景介绍1.1问题的由来随着互联网和大数据技术的迅猛发展,社交网络、推荐系统、生物信息学、图分析等领域对图计算的需求日益增长。传统的图处理技术如GraphLab、Neo4j等,虽然功能强大,但往往存在扩展性差、易用性低、计算效率不足等问题。为了解决这些问题,A
- R语言的计算机基础
java熊猫
包罗万象golang开发语言后端
R语言计算机基础引言R语言是一种用于数据分析、统计计算和图形显示的编程语言。它被广泛应用于统计学、数据科学、生态学、生物信息学等多个领域。由于其强大的功能和灵活性,R语言在学术界和工业界都得到了广泛的认可和应用。本文将从R语言的基本概念、数据类型、数据结构、函数、控制结构、图形绘制等方面进行介绍,帮助读者掌握R语言的基础知识。一、R语言的基本概念R语言源于新西兰的维特利大学,最初由RobertGe
- Web APP 阶段性综述
预测模型的开发与应用研究
APPconstructionwebapp
WebAPP阶段性综述当前,WebAPP主要应用于电脑端,常被用于部署数据分析、机器学习及深度学习等高算力需求的任务。在医学与生物信息学领域,WebAPP扮演着重要角色。在生物信息学领域,诸多工具以WebAPP的形式呈现,相较之下,医学领域的此类应用数量相对较少。在医学和生物信息学的学术论文中,WebAPP是展示研究成果的有效工具,并且还能部署到网络上,服务于实际应用场景。ShinyAPP平台特性
- 推荐一份生物信息学入门很好的参考材料
小明的数据分析笔记本
链接是https://bioinformatics.uconn.edu/resources-and-events/tutorials-2/这个是康涅狄格大学(UniversityofConnecticut)提供的一份教程,主要的内容包括1、生物信息学中经常用到的文件格式image.png2、linux操作系统和R语言的基础知识image.png3、转录组数据的处理流程image.png这里包括有参
- 【机器学习】朴素贝叶斯方法的概率图表示以及贝叶斯统计中的共轭先验方法
Lossya
机器学习概率论人工智能朴素贝叶斯共轭先验
引言朴素贝叶斯方法是一种基于贝叶斯定理的简单概率模型,它假设特征之间相互独立。文章目录引言一、朴素贝叶斯方法的概率图表示1.1节点表示1.2边表示1.3无其他连接1.4总结二、朴素贝叶斯的应用场景2.1文本分类2.2推荐系统2.3医疗诊断2.4欺诈检测2.5情感分析2.6邮件过滤2.7信息检索2.8生物信息学三、朴素贝叶斯的优点四、朴素贝叶斯的局限性4.1特征独立性假设4.2敏感于输入数据的表示4
- 零基础入门生信数据分析——导读
呆猪儿
生信之转录组——上游分析生信之转录组——下游分析学习方法r语言数据分析数据库数据挖掘需求分析大数据
零基础入门生信数据分析——导读生信数据分析,即生物信息学数据分析,是一个涵盖了生物学、计算机科学、数学和统计学等多个领域的交叉学科。它主要利用计算机算法和统计方法对生物学数据进行处理、分析和解释,以揭示生物分子、细胞、组织和生物体等各个层次的生物学规律和机制。本帖主要是为生信数据分析的各个分析点提供跳转链接(简单说就是提供了一个目录供大家选择自己想要的知识点可以直接跳转)关联的生信数据分析的分析点
- NCBI BLAST+:分析生物内在编码的工具
belldeep
生物信息学Blast生物数据分析
在生物信息学的广阔领域中,NCBI(NationalCenterforBiotechnologyInformation,美国国立生物技术信息中心)开发的BLAST(BasicLocalAlignmentSearchTool,基本局部比对搜索工具)无疑是一把不可或缺的分析工具。NCBIBLAST+,作为其最新版本2.16.0+,为科研工作者提供了一套强大的序列比对和搜索功能,帮助解析生命现象背后的遗
- 【图论简介】
WA-自动机
图论深度优先算法架构后端前端面试
图论简介图论是一门数学分支,主要研究图(Graph)的性质、结构和应用。图论在计算机科学、网络理论、优化问题、生物信息学等多个领域都有广泛的应用。本文将简要介绍图论的基本概念、常见算法及其在实际中的应用。一、图的基本概念图(Graph):图是由一组顶点(Vertices)和连接顶点的边(Edges)组成的结构。可以表示为(G=(V,E)),其中(V)是顶点的集合,(E)是边的集合。根据边的不同属性
- 生信圆桌:专业生信服务器与平台服务的提供者
生信圆桌x生信云服务器
服务器人工智能运维
生信圆桌是一个专注于提供生物信息学(生信)服务器和平台服务的领先企业,致力于为全球科研机构、企业和独立研究者提供高性能的生信分析解决方案。随着生物信息学研究对计算资源的需求日益增加,生信圆桌凭借其先进的服务器技术和专业的服务团队,成为了生信领域中不可或缺的合作伙伴。访问生信圆桌,使用生信云。高效分析少走弯路www.tebteb.cc生信圆桌的核心服务高性能生信服务器定制:生信圆桌为客户提供定制化的
- 用Python实现生信分析——基序(Motif)识别详解
写代码的M教授
生信分析python开发语言
1.什么是基序(Motif)?在生物信息学中,基序(Motif)是指在生物序列(如DNA、RNA或蛋白质序列)中具有特定功能或结构的短序列片段。基序通常在生物进化中得到保留,因为它们在生物学功能中起着重要作用。例如,在DNA序列中,基序可能是一个转录因子结合位点;在蛋白质序列中,基序可能是一个具有特定功能的结构域。基序识别是指从一组生物序列中识别出保守的短序列片段,这对于功能预测、基因调控网络分析
- 数据结构与算法——动态规划
passion更好
数据结构C++动态规划算法
目录引言最优子结构重叠子问题打家劫舍(LeetCode198题)经典例题1.爬楼梯(LeetCode70题)2.斐波那契数列(LeetCode126题)3.最长公共子序列(LeetCode95题)引言动态规划(DynamicProgramming,简称DP)是一种在数学、计算机科学、经济学和生物信息学等领域广泛使用的算法设计技术。它通过把原问题分解为相对简单的子问题的方式,来求解复杂问题。动态规划
- 深度学习——概念引入
韶光流年都束之高阁
深度学习日记深度学习人工智能职场和发展
深度学习深度学习简介深度学习分类根据网络结构划分:循环神经网络卷积神经网络根据学习方式划分:监督学习无监督学习半监督学习根据应用领域划分:计算机视觉自然语言处理语音识别生物信息学深度学习简介深度学习(DeepLearning,DL)是机器学习领域中的一个新的研究方向,主要是通过学习样本数据的内在规律和表示层次,让机器能够具有类似于人类的分析学习能力。深度学习的最终目标是让机器能够识别和解释各种数据
- 考研调剂:中医生命科学
菌心说双脑论
科学网—考研调剂——欢迎研究生调剂到我们的招生专业方向“中西医结合基础”:中医药与肠道菌群、生物信息学等交叉学科-张成岗的博文http://blog.sciencenet.cn/home.php?mod=space&uid=40692&do=blog&id=1281078欢迎各位有志于从事中医生命科学、解码中医、中西医结合以及医学与数学、计算机科学等交叉学科研究的青年才俊加入我们的研究团队,共同见
- 2020-04-07
liuyang2020
学习小组Day2笔记--linux入门(刘阳)1.为什么学习linux大多数人用的是可视化界面,便捷的windows,linux用户量比较少,但是需要知道,linux的功能相当的强大,对于数据处理、程序运行方面的优势,那是其它的系统无法比拟的,生物信息学数据处理对电脑要求较高,因此学习linux,,嘿嘿,大势所趋。2.linux操作2.1登录远程登录linux服务器,好像有很多连接软件,今天尝试应
- Python在生物信息学中的应用:有序字典
简说基因-专业生信合作伙伴
python开发语言
我们知道,通过{}创建的字典是无序的。如何创建有序字典呢?解决方案可以使用collections模块中的OrderedDict类。当对字典做迭代时,它会严格按照元素添加的顺序进行。例如:from collection import OrderedDictd=OrderedDict()d['1st'] = 1d['2nd'] = 2d['3rd'] = 3d['4th']=4forkeyind:
- Python在生物信息学中的应用:同时对数据做转换和换算
简说基因-专业生信合作伙伴
python开发语言
我们需要调用一个换算(reduction)函数,例如sum()、min()、max()等,但首先得对数据做转换或筛选。解决方案一种优雅的方式能将数据换算和转换结合在一起,即在函数中使用生成器表达式。例如,要计算平方和,可以这样:nums=[1,2,3,4,5]s=sum(x*xforxinnums)更多的例子:#Determineifany.pyfilesexistinadirectoryimpo
- Python在生物信息学中的应用:列表推导式
简说基因-专业生信合作伙伴
pythonwindows开发语言
列表中有一些数据,我们想提取或删除某些值,该怎么办?解决方案最简单的方法是使用列表推导式(listcomprehension)。例如:>>>mylist=[1,4,-5,10,-7,2,3,-1]>>>[nforninmylistifn>0][1,4,10,2,3]>>>[nforninmylistifn>>列表推导式的使用需要注意其内存占用,当原始列表比较大时,其内存占用较高,可以使用生成器表达
- 最长公共子序列(LCS)
算法
定义(维基百科)在一个序列集合中(通常为两个序列)查找所有序列中最长的子序列。这与查找最长公共子串的问题不同的地方是:子序列不需要在原序列中占用连续的位置。最长公共子序列问题是一个经典的计算机科学问题,也是数据比较程序,比如Diff工具和生物信息学应用的基础。它也被广泛地应用在版本控制,比如Git用来调和文件之间的改变解决方案这类问题通常都是采用动态规划的思想来解决,核心就是构造出动态解决方程。以
- 自学生物信息学
gtt儿_生物信息学习
我是生物工程专业出身,在大三保研时选择了生物信息的道路,到现在为止已经在行业里摸爬滚打了6年的时间,在这6年的学习之路上疑惑过,也迷茫过,特此把我学习的过程以及遇到的问题总结出来以让大家避免出现同样的问题。在我学习生物信息过程的基础上带着大家顺畅的走一遍。在学习生物信息学之前,我们先来了解一下什么是生物信息学。生物信息学,顾名思义,生物学和信息学的结合。生物学,这个对大家比较简单,基本入生信行的同
- 我们能成为孩子的上帝吗—— 谁来管理非法行医的贺建奎
闲月农
贺建奎,原南方科技大学副教授,毕业于美国斯坦福大学,拥有多学科交叉的背景,并在基因测序仪研究,CRISPR基因编辑,生物信息学等多个领域取得研究突破。2018年11月26日,贺建奎“基因编辑婴儿”事件引发轩然大波。2018年12月19日,贺建奎入选《Nature》年度十大科学人物。2019年4月18日,上榜美国《时代》杂志(Time)2019年度全球百位最具影响力人物榜单。2019年12月30日,
- LeetCode[Math] - #66 Plus One
Cwind
javaLeetCode题解AlgorithmMath
原题链接:#66 Plus One
要求:
给定一个用数字数组表示的非负整数,如num1 = {1, 2, 3, 9}, num2 = {9, 9}等,给这个数加上1。
注意:
1. 数字的较高位存在数组的头上,即num1表示数字1239
2. 每一位(数组中的每个元素)的取值范围为0~9
难度:简单
分析:
题目比较简单,只须从数组
- JQuery中$.ajax()方法参数详解
AILIKES
JavaScriptjsonpjqueryAjaxjson
url: 要求为String类型的参数,(默认为当前页地址)发送请求的地址。
type: 要求为String类型的参数,请求方式(post或get)默认为get。注意其他http请求方法,例如put和 delete也可以使用,但仅部分浏览器支持。
timeout: 要求为Number类型的参数,设置请求超时时间(毫秒)。此设置将覆盖$.ajaxSetup()方法的全局
- JConsole & JVisualVM远程监视Webphere服务器JVM
Kai_Ge
JVisualVMJConsoleWebphere
JConsole是JDK里自带的一个工具,可以监测Java程序运行时所有对象的申请、释放等动作,将内存管理的所有信息进行统计、分析、可视化。我们可以根据这些信息判断程序是否有内存泄漏问题。
使用JConsole工具来分析WAS的JVM问题,需要进行相关的配置。
首先我们看WAS服务器端的配置.
1、登录was控制台https://10.4.119.18
- 自定义annotation
120153216
annotation
Java annotation 自定义注释@interface的用法 一、什么是注释
说起注释,得先提一提什么是元数据(metadata)。所谓元数据就是数据的数据。也就是说,元数据是描述数据的。就象数据表中的字段一样,每个字段描述了这个字段下的数据的含义。而J2SE5.0中提供的注释就是java源代码的元数据,也就是说注释是描述java源
- CentOS 5/6.X 使用 EPEL YUM源
2002wmj
centos
CentOS 6.X 安装使用EPEL YUM源1. 查看操作系统版本[root@node1 ~]# uname -a Linux node1.test.com 2.6.32-358.el6.x86_64 #1 SMP Fri Feb 22 00:31:26 UTC 2013 x86_64 x86_64 x86_64 GNU/Linux [root@node1 ~]#
- 在SQLSERVER中查找缺失和无用的索引SQL
357029540
SQL Server
--缺失的索引
SELECT avg_total_user_cost * avg_user_impact * ( user_scans + user_seeks ) AS PossibleImprovement ,
last_user_seek ,
- Spring3 MVC 笔记(二) —json+rest优化
7454103
Spring3 MVC
接上次的 spring mvc 注解的一些详细信息!
其实也是一些个人的学习笔记 呵呵!
- 替换“\”的时候报错Unexpected internal error near index 1 \ ^
adminjun
java“\替换”
发现还是有些东西没有刻子脑子里,,过段时间就没什么概念了,所以贴出来...以免再忘...
在拆分字符串时遇到通过 \ 来拆分,可是用所以想通过转义 \\ 来拆分的时候会报异常
public class Main {
/*
- POJ 1035 Spell checker(哈希表)
aijuans
暴力求解--哈希表
/*
题意:输入字典,然后输入单词,判断字典中是否出现过该单词,或者是否进行删除、添加、替换操作,如果是,则输出对应的字典中的单词
要求按照输入时候的排名输出
题解:建立两个哈希表。一个存储字典和输入字典中单词的排名,一个进行最后输出的判重
*/
#include <iostream>
//#define
using namespace std;
const int HASH =
- 通过原型实现javascript Array的去重、最大值和最小值
ayaoxinchao
JavaScriptarrayprototype
用原型函数(prototype)可以定义一些很方便的自定义函数,实现各种自定义功能。本次主要是实现了Array的去重、获取最大值和最小值。
实现代码如下:
<script type="text/javascript">
Array.prototype.unique = function() {
var a = {};
var le
- UIWebView实现https双向认证请求
bewithme
UIWebViewhttpsObjective-C
什么是HTTPS双向认证我已在先前的博文 ASIHTTPRequest实现https双向认证请求
中有讲述,不理解的读者可以先复习一下。本文是用UIWebView来实现对需要客户端证书验证的服务请求,网上有些文章中有涉及到此内容,但都只言片语,没有讲完全,更没有完整的代码,让人困扰不已。但是此知
- NoSQL数据库之Redis数据库管理(Redis高级应用之事务处理、持久化操作、pub_sub、虚拟内存)
bijian1013
redis数据库NoSQL
3.事务处理
Redis对事务的支持目前不比较简单。Redis只能保证一个client发起的事务中的命令可以连续的执行,而中间不会插入其他client的命令。当一个client在一个连接中发出multi命令时,这个连接会进入一个事务上下文,该连接后续的命令不会立即执行,而是先放到一个队列中,当执行exec命令时,redis会顺序的执行队列中
- 各数据库分页sql备忘
bingyingao
oraclesql分页
ORACLE
下面这个效率很低
SELECT * FROM ( SELECT A.*, ROWNUM RN FROM (SELECT * FROM IPAY_RCD_FS_RETURN order by id desc) A ) WHERE RN <20;
下面这个效率很高
SELECT A.*, ROWNUM RN FROM (SELECT * FROM IPAY_RCD_
- 【Scala七】Scala核心一:函数
bit1129
scala
1. 如果函数体只有一行代码,则可以不用写{},比如
def print(x: Int) = println(x)
一行上的多条语句用分号隔开,则只有第一句属于方法体,例如
def printWithValue(x: Int) : String= println(x); "ABC"
上面的代码报错,因为,printWithValue的方法
- 了解GHC的factorial编译过程
bookjovi
haskell
GHC相对其他主流语言的编译器或解释器还是比较复杂的,一部分原因是haskell本身的设计就不易于实现compiler,如lazy特性,static typed,类型推导等。
关于GHC的内部实现有篇文章说的挺好,这里,文中在RTS一节中详细说了haskell的concurrent实现,里面提到了green thread,如果熟悉Go语言的话就会发现,ghc的concurrent实现和Go有点类
- Java-Collections Framework学习与总结-LinkedHashMap
BrokenDreams
LinkedHashMap
前面总结了java.util.HashMap,了解了其内部由散列表实现,每个桶内是一个单向链表。那有没有双向链表的实现呢?双向链表的实现会具备什么特性呢?来看一下HashMap的一个子类——java.util.LinkedHashMap。
- 读《研磨设计模式》-代码笔记-抽象工厂模式-Abstract Factory
bylijinnan
abstract
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
package design.pattern;
/*
* Abstract Factory Pattern
* 抽象工厂模式的目的是:
* 通过在抽象工厂里面定义一组产品接口,方便地切换“产品簇”
* 这些接口是相关或者相依赖的
- 压暗面部高光
cherishLC
PS
方法一、压暗高光&重新着色
当皮肤很油又使用闪光灯时,很容易在面部形成高光区域。
下面讲一下我今天处理高光区域的心得:
皮肤可以分为纹理和色彩两个属性。其中纹理主要由亮度通道(Lab模式的L通道)决定,色彩则由a、b通道确定。
处理思路为在保持高光区域纹理的情况下,对高光区域着色。具体步骤为:降低高光区域的整体的亮度,再进行着色。
如果想简化步骤,可以只进行着色(参看下面的步骤1
- Java VisualVM监控远程JVM
crabdave
visualvm
Java VisualVM监控远程JVM
JDK1.6开始自带的VisualVM就是不错的监控工具.
这个工具就在JAVA_HOME\bin\目录下的jvisualvm.exe, 双击这个文件就能看到界面
通过JMX连接远程机器, 需要经过下面的配置:
1. 修改远程机器JDK配置文件 (我这里远程机器是linux).
- Saiku去掉登录模块
daizj
saiku登录olapBI
1、修改applicationContext-saiku-webapp.xml
<security:intercept-url pattern="/rest/**" access="IS_AUTHENTICATED_ANONYMOUSLY" />
<security:intercept-url pattern=&qu
- 浅析 Flex中的Focus
dsjt
htmlFlexFlash
关键字:focus、 setFocus、 IFocusManager、KeyboardEvent
焦点、设置焦点、获得焦点、键盘事件
一、无焦点的困扰——组件监听不到键盘事件
原因:只有获得焦点的组件(确切说是InteractiveObject)才能监听到键盘事件的目标阶段;键盘事件(flash.events.KeyboardEvent)参与冒泡阶段,所以焦点组件的父项(以及它爸
- Yii全局函数使用
dcj3sjt126com
yii
由于YII致力于完美的整合第三方库,它并没有定义任何全局函数。yii中的每一个应用都需要全类别和对象范围。例如,Yii::app()->user;Yii::app()->params['name'];等等。我们可以自行设定全局函数,使得代码看起来更加简洁易用。(原文地址)
我们可以保存在globals.php在protected目录下。然后,在入口脚本index.php的,我们包括在
- 设计模式之单例模式二(解决无序写入的问题)
come_for_dream
单例模式volatile乱序执行双重检验锁
在上篇文章中我们使用了双重检验锁的方式避免懒汉式单例模式下由于多线程造成的实例被多次创建的问题,但是因为由于JVM为了使得处理器内部的运算单元能充分利用,处理器可能会对输入代码进行乱序执行(Out Of Order Execute)优化,处理器会在计算之后将乱序执行的结果进行重组,保证该
- 程序员从初级到高级的蜕变
gcq511120594
框架工作PHPandroidhtml5
软件开发是一个奇怪的行业,市场远远供不应求。这是一个已经存在多年的问题,而且随着时间的流逝,愈演愈烈。
我们严重缺乏能够满足需求的人才。这个行业相当年轻。大多数软件项目是失败的。几乎所有的项目都会超出预算。我们解决问题的最佳指导方针可以归结为——“用一些通用方法去解决问题,当然这些方法常常不管用,于是,唯一能做的就是不断地尝试,逐个看看是否奏效”。
现在我们把淫浸代码时间超过3年的开发人员称为
- Reverse Linked List
hcx2013
list
Reverse a singly linked list.
/**
* Definition for singly-linked list.
* public class ListNode {
* int val;
* ListNode next;
* ListNode(int x) { val = x; }
* }
*/
p
- Spring4.1新特性——数据库集成测试
jinnianshilongnian
spring 4.1
目录
Spring4.1新特性——综述
Spring4.1新特性——Spring核心部分及其他
Spring4.1新特性——Spring缓存框架增强
Spring4.1新特性——异步调用和事件机制的异常处理
Spring4.1新特性——数据库集成测试脚本初始化
Spring4.1新特性——Spring MVC增强
Spring4.1新特性——页面自动化测试框架Spring MVC T
- C# Ajax上传图片同时生成微缩图(附Demo)
liyonghui160com
1.Ajax无刷新上传图片,详情请阅我的这篇文章。(jquery + c# ashx)
2.C#位图处理 System.Drawing。
3.最新demo支持IE7,IE8,Fir
- Java list三种遍历方法性能比较
pda158
java
从c/c++语言转向java开发,学习java语言list遍历的三种方法,顺便测试各种遍历方法的性能,测试方法为在ArrayList中插入1千万条记录,然后遍历ArrayList,发现了一个奇怪的现象,测试代码例如以下:
package com.hisense.tiger.list;
import java.util.ArrayList;
import java.util.Iterator;
- 300个涵盖IT各方面的免费资源(上)——商业与市场篇
shoothao
seo商业与市场IT资源免费资源
A.网站模板+logo+服务器主机+发票生成
HTML5 UP:响应式的HTML5和CSS3网站模板。
Bootswatch:免费的Bootstrap主题。
Templated:收集了845个免费的CSS和HTML5网站模板。
Wordpress.org|Wordpress.com:可免费创建你的新网站。
Strikingly:关注领域中免费无限的移动优
- localStorage、sessionStorage
uule
localStorage
W3School 例子
HTML5 提供了两种在客户端存储数据的新方法:
localStorage - 没有时间限制的数据存储
sessionStorage - 针对一个 session 的数据存储
之前,这些都是由 cookie 完成的。但是 cookie 不适合大量数据的存储,因为它们由每个对服务器的请求来传递,这使得 cookie 速度很慢而且效率也不