lasso回归结果一行代码提取及美化
获取更多R语言知识,请关注公众号:医学和生信笔记
医学和生信笔记,专注R语言在临床医学中的使用,R语言数据分析和可视化。主要分享R语言做医学统计学、meta分析、网络药理学、临床预测模型、机器学习、生物信息学等。
文章目录
-
- 加载R包
- 默认画图
- 提取数据
- 自定义绘图
lasso回归非常常用,默认的图不丑,但是总有人想要自定义,想要更好看。
其实画图很简单,难的是提取数据。
说到提取数据,说难不难,说简单不简单,如果你会用搜索,就非常简单,根本不用自己写,一般来说,你遇到过的问题,本人肯定早就遇到过且解决了!
下面就给大家演示下怎么用1行代码提取数据!包你满意!包治百病!
加载R包
library(glmnet)
## 载入需要的程辑包:Matrix
## Loaded glmnet 4.1-3
library(survival)
自带的生存分析数据,方便学习。
data(CoxExample)
x <- CoxExample$x
y <- CoxExample$y
head(y)
## time status
## [1,] 1.76877757 1
## [2,] 0.54528404 1
## [3,] 0.04485918 0
## [4,] 0.85032298 0
## [5,] 0.61488426 1
## [6,] 0.29860939 0
默认画图
mod <- glmnet(x,y,family = "cox")
cvmod <- cv.glmnet(x,y,family="cox") # 交叉验证
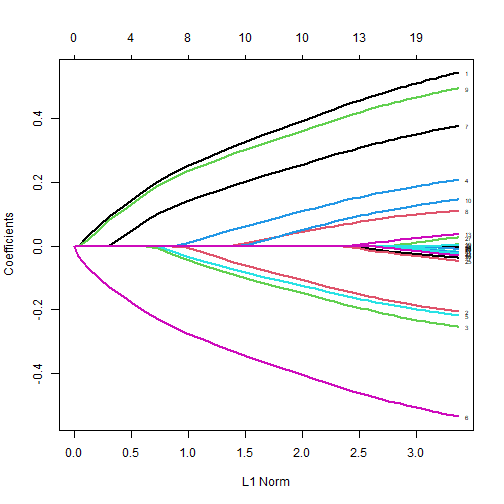
下面3个都是默认的函数画的图,真不能说丑,你看超多SCI里都是直接用的原图。
plot(mod,label = T,lwd=2)
plot(mod,xvar = "lambda",label = T,lwd=2)
plot(cvmod)
提取数据
如果你想提取数据,也没什么难度,就是慢慢找,肯定能找到。
library(tidyverse)
## -- Attaching packages ----------------------------- tidyverse 1.3.1 --
## v ggplot2 3.3.5 v purrr 0.3.4
## v tibble 3.1.6 v dplyr 1.0.8
## v tidyr 1.2.0 v stringr 1.4.0
## v readr 2.1.1 v forcats 0.5.1
## -- Conflicts -------------------------------- tidyverse_conflicts() --
## x tidyr::expand() masks Matrix::expand()
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()
## x tidyr::pack() masks Matrix::pack()
## x tidyr::unpack() masks Matrix::unpack()
tmp <- as_tibble(as.matrix(coef(mod)), rownames = "coef") %>%
pivot_longer(cols = -coef,
names_to = "variable",
names_transform = list(variable = parse_number),
values_to = "value") %>%
group_by(variable) %>%
mutate(lambda = mod$lambda[variable + 1],
norm = sum(if_else(coef == "(Intercept)", 0, abs(value))))
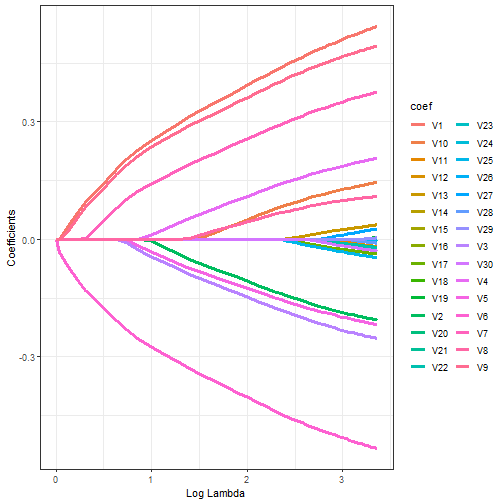
接下来画图就非常简单。
ggplot(tmp, aes(norm,value,color=coef,group=coef))+
geom_line(size=1.2)+
labs(x="Log Lambda",y="Coefficients")+
theme_bw()
当然,肯定也有简便方法!
俗话说得好:
包治百病,包你满意。
下面就是我的表演,用包搞定这个看似复杂的问题!
这个包很神奇,大家有空可以学习下,没空的话等我更新。
# 就是这个包,没装的自己安装下!
library(broom)
# 提取数据,就是这么简单!
tidy_df <- broom::tidy(mod)
tidy_cvdf <- broom::tidy(cvmod)
够简单吗?我觉得比自己撸代码简单。
# 给大家看看数据结构
head(tidy_df)
## # A tibble: 6 x 5
## term step estimate lambda dev.ratio
##
## 1 V1 3 0.0312 0.197 0.00596
## 2 V1 4 0.0637 0.179 0.0105
## 3 V1 5 0.0939 0.163 0.0144
## 4 V1 6 0.125 0.149 0.0188
## 5 V1 7 0.153 0.135 0.0226
## 6 V1 8 0.179 0.123 0.0256
head(tidy_cvdf)
## # A tibble: 6 x 6
## lambda estimate std.error conf.low conf.high nzero
##
## 1 0.237 13.7 0.0429 13.7 13.7 0
## 2 0.216 13.7 0.0427 13.6 13.7 1
## 3 0.197 13.6 0.0421 13.6 13.7 3
## 4 0.179 13.6 0.0419 13.5 13.6 3
## 5 0.163 13.5 0.0422 13.5 13.6 4
## 6 0.149 13.5 0.0429 13.4 13.5 4
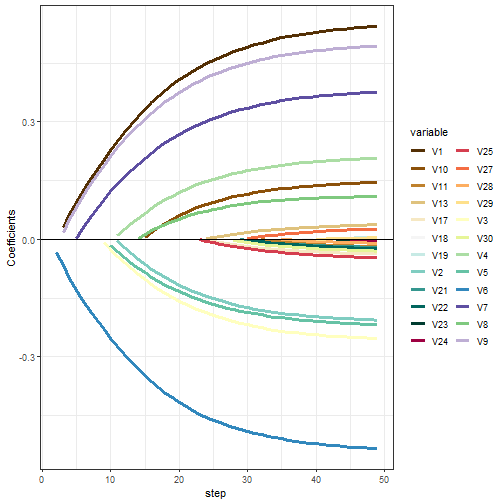
自定义绘图
有了数据,画图就很简单了。
library(ggplot2)
library(RColorBrewer)
#随便定义几个颜色,多找几个,防止不够用
mypalette <- c(brewer.pal(11,"BrBG"),brewer.pal(11,"Spectral"),brewer.pal(5,"Accent"))
ggplot(tidy_df, aes(step, estimate, group = term,color=term)) +
geom_line(size=1.2)+
geom_hline(yintercept = 0)+
ylab("Coefficients")+
scale_color_manual(name="variable",values = mypalette)+
theme_bw()
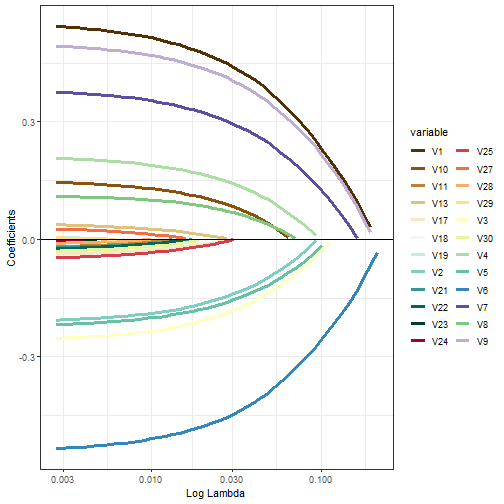
p2 <- ggplot(tidy_df, aes(lambda, estimate, group = term, color = term)) +
geom_line(size=1.2)+
geom_hline(yintercept = 0)+
scale_x_log10(name = "Log Lambda")+
ylab("Coefficients")+
scale_color_manual(name="variable",values = mypalette)+
theme_bw()
p2
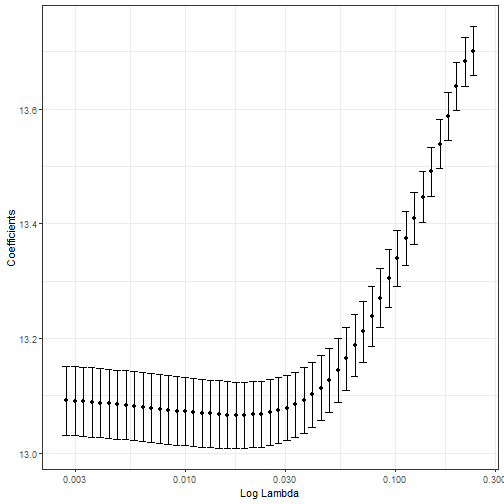
p3 <- ggplot()+
geom_point(data=tidy_cvdf, aes(lambda,estimate))+
geom_errorbar(data = tidy_cvdf, aes(x=lambda,ymin=conf.low,ymax=conf.high))+
scale_x_log10(name = "Log Lambda")+
ylab("Coefficients")+
theme_bw()
p3
如果你发文章用的话,横坐标多数都是log lambda,所以你可以把第2张和第3张拼一起,完全不影响使用。
library(patchwork)
p2 / p3
获取更多R语言知识,请关注公众号:医学和生信笔记
医学和生信笔记,专注R语言在临床医学中的使用,R语言数据分析和可视化。主要分享R语言做医学统计学、meta分析、网络药理学、临床预测模型、机器学习、生物信息学等。