【Bioinfo Blog 012】【R Code 010】——生存分析(Kaplan-Meier & Cox)
目录
- 一、生存分析(Survival Analysis)基本概念
-
- 1.1 随访研究
- 1.2 起始事件与终点事件
- 1.3 生存时间
- 1.4 生存函数(Survival Function)
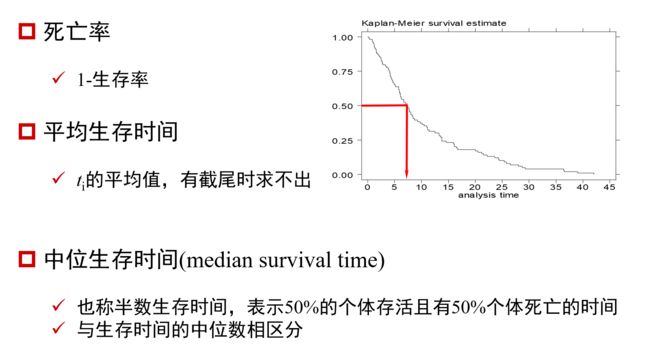
- 1.5 常用指标
- 1.2 主要内容及研究方法
- 二、Kaplan-Meier 生存曲线
-
- 2.1 生存率估计的概率乘法原理
- 2.2 R语言实现
-
- 2.2.1 环境搭建
- 2.2.2 生存函数构建
- 2.2.3 可视化
- 三、Cox比例风险模型
-
- 3.1 Cox比例风险模型的概念
- 3.2 R语言实现
- 四、log-rank检验
- 五、参考
- 作业1
一、生存分析(Survival Analysis)基本概念
1.1 随访研究
随访研究(follow-up study)是一种前瞻性(试验or调查)研究。

1.2 起始事件与终点事件
起始事件(initial event):反应生存时间起始特征的事件,如疾病确诊、某种疾病治疗开始等。
失效事件(failure event):在生存分析随访研究过程中,一部分研究对象可观察到死亡,可以得到准确的生存时间,它提供的信息是完全的,这种事件称为失效事件,也称之为死亡事件、终点事件。
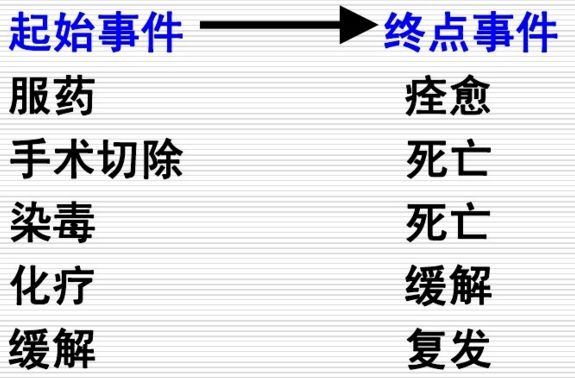
起始事件和失效事件是相对而言的,它们都由研究目的决定,在设计时须明确规定,并在研究期间严格遵守,不能随意改变。
1.3 生存时间
广义上指某个起点事件开始到某个终点事件发生所经历的时间,度量单位可以是年、月、日、小时等,常用符号t所示。

生存时间类型
- 完全数据(complete data)
观察对象在观察期内出现终点事件,记录到的时间信息是完整的,这种生存时间数据称为完全数据。 - 截尾数据(censored data)
尚未观察到研究对象出现终点事件时,即由于某种原因停止了随访,这时记录到的时间信息是不完整的,这种生存时间数据称为不完全数据或截尾值。常用符号“+”表示。
截尾的原因
主要有3种:
①失访(lost of follow up):中途失访:包括拒绝访问、失去联系等。
②退出(quit of experiment):中途退出试验、改变治疗方案。死于其它与研究无关的原因:如肺癌患者死于心机梗塞、自杀或因车祸死亡,终止随访时间为死亡时间。
③终止(terminated):指观察期结束时仍未出现结局。
生存时间资料的整理
- 对于随访资料,需记录的原始数据包括
开始观察的时点、终止观察的时点、研究对象的结局、考虑的影响因素。 - 生存时间为反映时间长短的指标,属数值变量:
生存时间(t)= 终止观察的时点–开始观察的时点; - 结局变量(δ)反映终点事件是否发生,为二分类的变量。
- 通常用(t, δ)完整地表示一个观察对象的随访结果。
1.4 生存函数(Survival Function)
1.5 常用指标
1.2 主要内容及研究方法
二、Kaplan-Meier 生存曲线
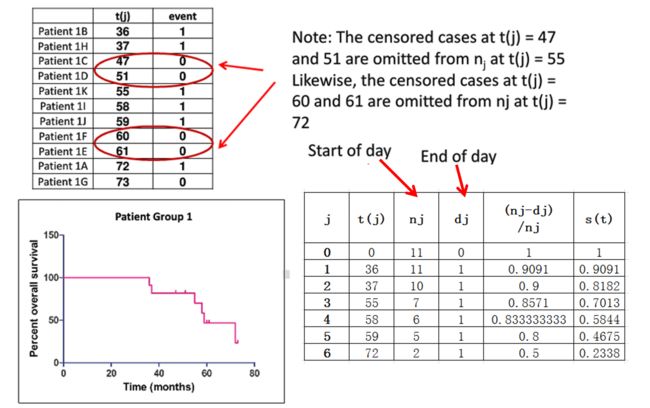
2.1 生存率估计的概率乘法原理
2.2 R语言实现
2.2.1 环境搭建
# install.packages("survminer")
# install.packages("survival")
library(survival)
library(ggplot2)
library(ggpubr)
library(survminer)
library(dplyr)
cliDat <- read.table("tcga-clinical data.txt",sep = "\t",header = T)
seqDat <- read.table("tcga-01-seq.txt",sep = "\t",header = T)
cliDat:
seqDat:

可以看到seqDat中barcode的一列为大写,因为后续需要合并这两个数据,需要用tolower()将其转为小写~
seqDat$patient.bcr_patient_barcode <- tolower(seqDat$patient.bcr_patient_barcode)
然后将两个数据集合并,利用dplyr包的inner_join()函数
mydat <- inner_join(cliDat,seqDat, by = "patient.bcr_patient_barcode")
2.2.2 生存函数构建
在survival包中先使用Surv()函数创建生存对象,生存对象是将事件时间和删失信息合并在一起的数据结构。使用survfit()函数来拟合生存曲线。
fit<- survfit(Surv(survival.time, patient.vital_status) ~ SAV1_exp, data = mydat)
## 查看看完整的生存表格
summary(fit_hw)
summary(fit)$table
info <- data.frame(time = fit$time,
n.risk = fit$n.risk,
n.event = fit$n.event,
n.censor = fit$n.censor,
surv = fit$surv,
upper = fit$upper,
lower = fit$lower
)
head(info)

time:曲线上的时间点。
n.riks:在时间t处有风险的受试者人数
n.event:在时间t发生的事件数
n.censor:在时间t退出事件而不发生风险的删失者的数量
lower,upper:曲线的置信度上限和下限
2.2.3 可视化
conf.int = TRUE 显示生存函数的95%置信区间。
risk.table =TRUE 显示按时间划分的处于风险中的人的数量和/或百分比。
pval = TRUE 显示对数秩检验(Log-Rank test)的p值。
surv.median.line 显示中位生存时间点水平/垂直线。允许的值包括c(“ none”,“ hv”,“ h”,“ v”)
ggsurvplot(fit_hw,
pval = TRUE, conf.int = TRUE,
risk.table = TRUE, # 添加风险表
risk.table.col = "strata",
surv.median.line = "hv", # 显示中位生存时间
xlab = "Follow up time(d)", # 指定x轴标签
legend.title = "SAV1_exp", # 设置图例标题
legend.labs = c("low", "high"), # 指定图例分组标签
palette = c("#E7B800", "#2E9FDF"))#定义颜色
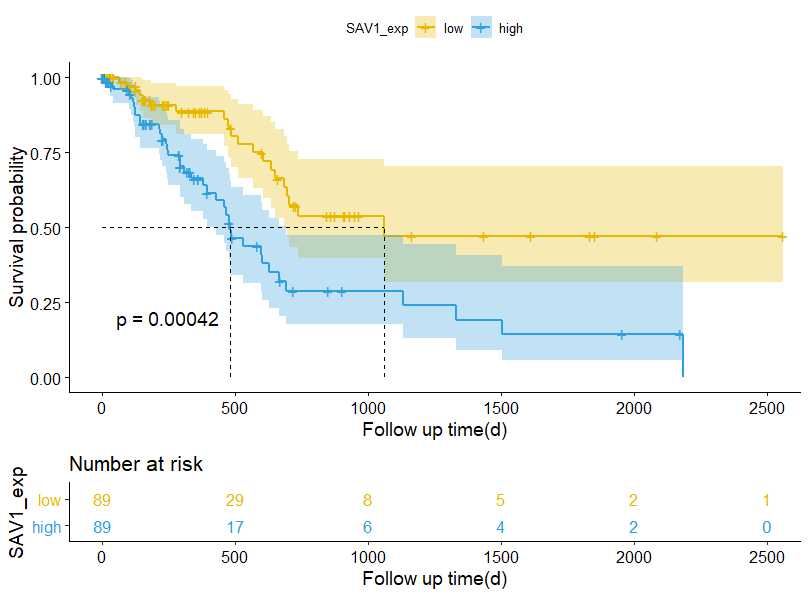
横轴(x轴)表示以天为单位的时间;
纵轴(y轴)表示生存的可能性或生存的人口比例;
线代表两组/层的存活曲线,曲线中的垂直下降表示事件,曲线上的十字叉表示此时患者删失。
在起点时,生存概率为1.0(或100%的参与者还活着)。
在时间500时,SAV1高表达人群的存活概率约为0.45,SAV1低表达人群的存活概率约为0.80。
SAV1高表达人群的中位生存时间约为490天,SAV1低表达人群的中位生存期约为1050天,这表明SAV1低表达人群的生存期高于SAV1高表达人群
三、Cox比例风险模型
KM生存分析模型是单变量分析,模型只描述了该单变量和生存之间的关系而忽略其他变量的影响。同时,Kaplan-Meier方法只能针对分类变量(治疗A vs 治疗B,男 vs 女),不能分析连续变量对生存造成的影响。
在实际临床研究中,影响事件发生的因素往往不止一个,它是多个因素综合作用的结果。Cox比例风险模型,它既适用于连续型变量也适用于类别变量。
3.1 Cox比例风险模型的概念
t 表示生存时间
h(t) 是由一组数量为p的协变量 (x1,x2,…,xpx1,x2,…,xp)
系数 (b1,b2,…,bpb1,b2,…,bp)可以衡量协变量的影响(即影响大小)。
h0 被称为基准风险,即当所有xi等于零(exp(0)数值等于1)时的事件风险。
H(t)中的“t”表示该风险是随时间变化的。
Exp(bi)称为风险比(Hazard ratios,HR),风险比大于1表示协变量与事件概率正相关,与生存时间负相关 。
HR = 1:无效
HR < 1:减少风险
HR > 1:增加风险
在癌症研究中:
HR > 1(即:b > 0)的协变量称为不良预后因素
HR < 1(即:b < 0)的协变量被称为良好预后因素
3.2 R语言实现
由于数据限制,这里只展示单因素Cox分析
fit_cox <- coxph(Surv(survival.time, patient.vital_status) ~ SAV1_exp, data = mydat)
summary(fit_cox)
四、log-rank检验
非参数假设检验:用来检验两组生存数据是否有显著差异
H0:两组个体在任意时间点,生存率无显著差异
H1:两组个体在任意时间点,生存率有显著差异
survDiff <- survdiff(Surv(survival.time, patient.vital_status) ~ SAV1_exp, data = mydat)
五、参考
R语言统计与绘图:ggsurvplot()函数绘制Kaplan-Meier生存曲线
R语言统计与绘图:Kaplan-Meier生存曲线绘制
生存分析(survival analysis)
第二十六讲 R-生存分析:绘制KM生存曲线
第二十八讲 R语言-Cox比例风险模型1
作业1
求出例1的累积生存率,并画出例1的KM曲线
根据例1给出的数据,可以推断出样本的状态,读入R进行绘制

绘制累积概率曲线:
a <- read.table("clipboard",header = T)
ggsurvplot(survfit(Surv(time,status)~1,data=a),
fun = "event", # l绘制1-S(t)曲线
conf.int = TRUE, # 输出曲线置信区间
conf.int.alpha=0.45, # 设置置信区间透明区
xlab="Follow up time(d)",
ylab="Cumulative Event Rate (%)", # 或者直接写“1-S(t)”
palette = "npg"
)