ccc-sklearn-4-特征降维
1.维度的概念
对于数组和Series,维度就是shape返回的数字个数。其中一行和一列都称为1维
对于一个特征矩阵或者DateFrame,维度指样本的数量或者特征的数量,一般都是特征的数量
对于图像来说,维度就是图像中特征向量的数量。各个特征向量之间相互垂直
降维算法的目的
- 降低特征矩阵中特征的数量,让算法运算更快,效果更好
- 对于三维以上的矩阵无法可视化,降维可以帮助可视化的实现,利于对于数据的直观理解
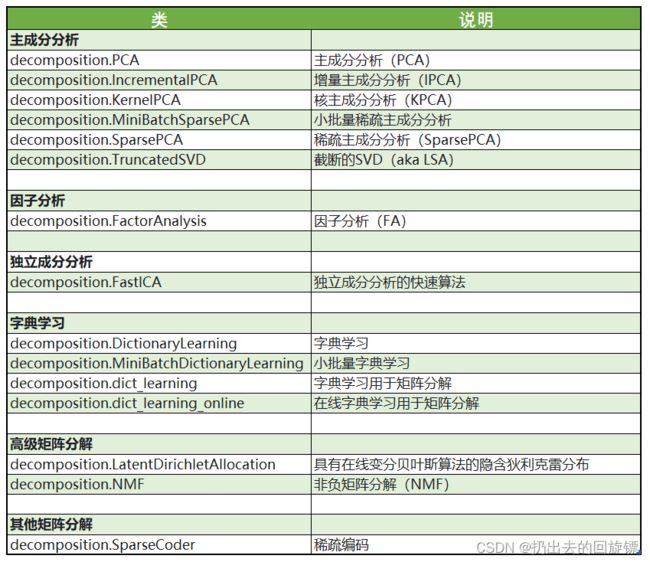
2.sklearn中的降维算法
sklearn的降维算法都在模块decomposition中,本质是一个矩阵分解模块。在降维、深度学习、聚类分析、数据预处理、低纬度特征、大数据分析等领域都广泛使用
PCA与SVD
在降维的过程中,希望减少特征数量的同时保留大部分有效信息,删除带无效的信息的特征的新的特征矩阵
PCA使用信息量衡量指标是样本方差,即可解释性方差,越大说明特征携带的信息量越多
V a r = 1 n − 1 ∑ i = 1 n ( x i − x ^ ) 2 Var = \frac{1}{n-1}\sum_{i=1}^{n}(x_i - \hat{x})^2 Var=n−11i=1∑n(xi−x^)2
Var代表一个特征的方差,n代表样本量, x i x_i xi表示一个特征中的每个样本取值, x ^ \hat{x} x^表示样本均值
说明:计算样本方差时为什么是除以(n-1)?
原则就是得到样本方差的无偏估计。所谓“无偏”,就是说,样本值应该是围绕总体值上下波动的,它不能总在总体值的上面,或者总在总体值下面。数学上可以证明:

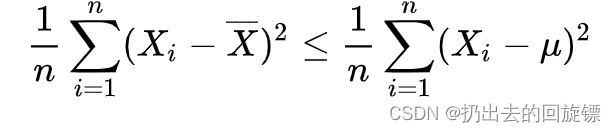
这样总是小于就不是无偏估计了,所以需要进行修正。至于修正的过程这里暂时不做解释。
降维是如何实现的
一组简单的二维数据降维

从二维到n维降维的基本过程:
| 过程 | 二维特征矩阵 | n维特征矩阵 |
|---|---|---|
| 1 | 输入原数据,结构(3,2) 找出原本2个特征对应的直角坐标系 |
输入原数据,结构(m,n) 找出原本n个特征构成的n维空间V |
| 2 | 决定降维后的特征数量:1 | 决定降维后的特征数量:k |
| 3 | 旋转,找出新的坐标系,将新特征向量被压缩并不损失太多总信息量 | 通过某种变化,找出n个新的特征向量以及这些向量构成的n维空间V |
| 4 | 找出数据在新的坐标系上,2个坐标轴上的坐标 | 找出原数据在新特征空间V中的n个新特征对应的值 |
| 5 | 选第一个方差最大的特征向量,删掉没有被选中的特征 | 选前k个信息量最大的特征,删除没有被选中的特征 |
说明:
- 不通降维算法都遵循这上面的步骤来进行降维,不同的是它们的第三部矩阵分解的算法不同,信息量的衡量指标也不同
- 特征选择是选择已有特征携带有信息最多的特征,选择后特征依然可以找到其在原数据的位置,代表的含义;PCA这种属于特征创造,即新特征举证生成前,无法知晓PCA建立了怎样的新特征向量,新特征向量生成后也不具有可读性,虽然带有原始数据的信息,但已经不是原始数据携带的含义了,所以PAC一般不适用于探索特征和标签之间的关系的模型
重要参数n_components与实际可视化案例
n_components即降维后需要保留的特征数量
步骤一:调库和数据
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
from sklearn.decomposition import PCA
iris = load_iris()
y = iris.target
X = iris.data
X.shape
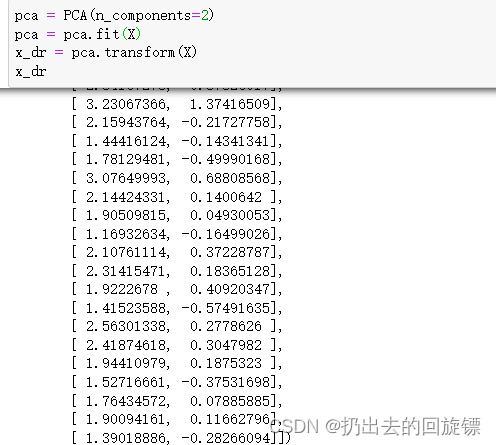
pca = PCA(n_components=2)
pca = pca.fit(X)
x_dr = pca.transform(X)
x_dr
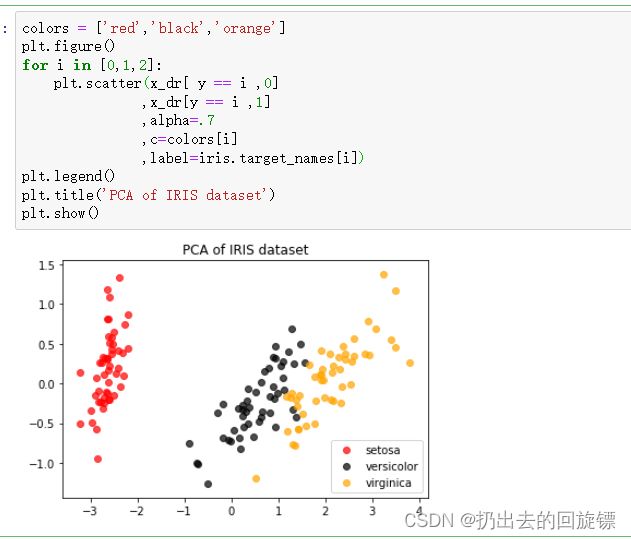
colors = ['red','black','orange']
plt.figure()
for i in [0,1,2]:
plt.scatter(x_dr[ y == i ,0]
,x_dr[y == i ,1]
,alpha=.7
,c=colors[i]
,label=iris.target_names[i])
plt.legend()
plt.title('PCA of IRIS dataset')
plt.show()
#查看降维后每个特征携带的信息量的大小
pca.explained_variance_
#查看降维后每个新特征所占信息量站原数据的百分比
pca.explained_variance_ratio_
pca.explained_variance_ratio_.sum()
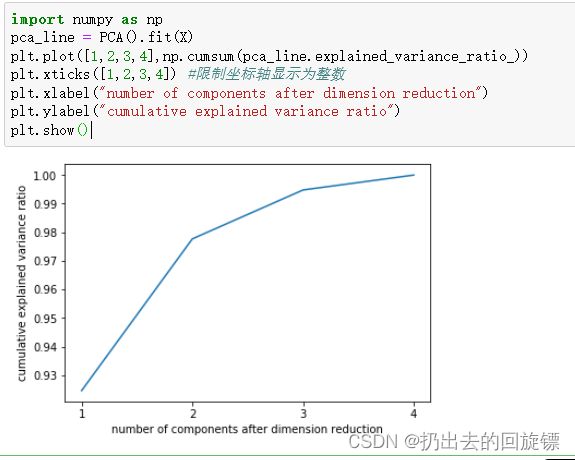
用于帮助我们决定n_components中最好的取值
import numpy as np
pca_line = PCA().fit(X)
plt.plot([1,2,3,4],np.cumsum(pca_line.explained_variance_ratio_))
plt.xticks([1,2,3,4]) #限制坐标轴显示为整数
plt.xlabel("number of components after dimension reduction")
plt.ylabel("cumulative explained variance ratio")
plt.show()
pca_mle = PCA(n_components="mle")
pca_mle = pca_mle.fit(X)
X_mle = pca_mle.transform(X)
X_mle
pca_mle.explained_variance_ratio_.sum()
步骤七:按照信息占比选超参数
pca_f = PCA(n_components=0.95,svd_solver="full")
pac_f = pca_f.fit(X)
X_f = pca_f.transform(X)
pca_f.explained_variance_ratio_.sum()

PCA中的SVD是什么?
svd_solver:奇异值分解器,可以不结算协方差等结构复杂、计算冗长的矩阵,直接求出新特征空间和降维后的特征矩阵
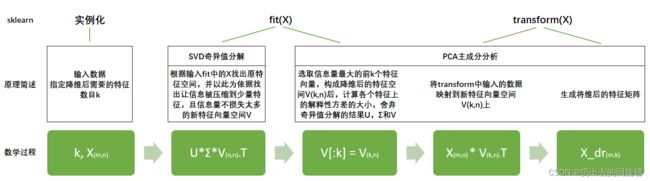
从流程中看到,sklearn中通过SVD求出特征空间,这样减少了计算量。可以认为fit中的矩阵U和Σ之后被舍弃,奇异值分解追求仅仅是V,保存在components_中
说明:右奇异矩阵 V T V^T VT具有以下特征:
X d r = X ∗ V [ : k ] T X_{dr}=X*V[:k]^T Xdr=X∗V[:k]T
k就是n_components,是我们降维后希望得到的维度。若X为(m,n)的特征矩阵, 就是结构为(n,n)的矩阵,取这个矩阵的前k行(进行切片),即将V转换为结构为(k,n)的矩阵。而 V ( k , n ) T V_{(k,n)}^T V(k,n)T与原特征矩阵X相乘,即可得到降维后的特征矩阵X_dr
svd_solver可选参数:默认auto
- auto:基于X.shape和n_components的默认策略来选择分解器:如果输入数据的尺寸大于500x500且要提取的特征数小于数据最小维度min(X.shape)的80%,就启用效率更高的”randomized“方法。否则,精确完整的SVD将被计算,截断将会在矩阵被分解完成后有选择地发生
- full:从scipy.linalg.svd中调用标准的LAPACK分解器来生成精确完整的SVD,适合数据量比较适中,计算时间充足的情况,生成的精确完整的SVD的结构为: U ( m , m ) , ∑ ( m , n ) , V ( n , n ) T U_(m,m),\sum_{}{}_{(m,n)},V^T_{(n,n)} U(m,m),∑(m,n),V(n,n)T
- arpack:从scipy.sparse.linalg.svds调用ARPACK分解器来运行截断奇异值分解(SVD truncated),分解时就将特征数量降到n_components中输入的数值k,可以加快运算速度,适合特征矩阵很大的时候,但一般用于特征矩阵为稀疏矩阵的情况,包含一定的随机性。截断后的SVD分解出的结构为: U ( m , k ) , ∑ ( k , k ) , V ( n , n ) T U_(m,k),\sum_{}{}_{(k,k)},V^T_{(n,n)} U(m,k),∑(k,k),V(n,n)T
- randomized:根据原始数据和输入的n_components值去计算和寻找符合需求的新特征向量,但是在"randomized"方法中,分解器会先生成多个随机向量,然后去检测这些随机向量中是否有任何一个符合我们的分解需求,如果符合,就保留这个随机向量,并基于这个随机向量来构建后续的向量空间。比"full"模式下计算快很多,并且还能够保证模型运行效果。适合特征矩阵巨大,计算量庞大的情况(svd_solver的值为"arpack" or "randomized"生效)
3.人脸识别案例与手写字识别噪音过滤
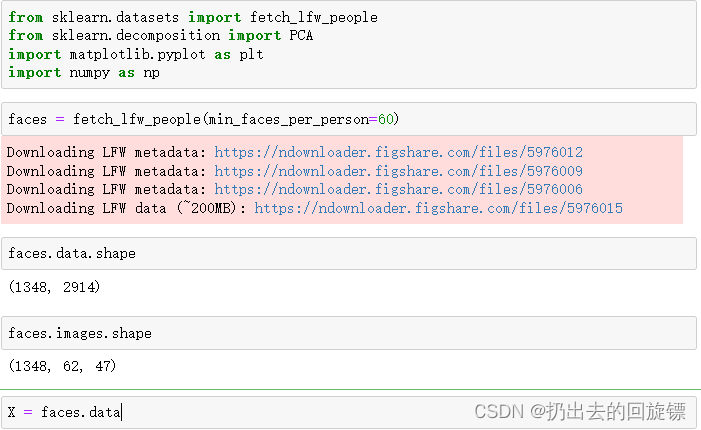
步骤一:导入库和数据
from sklearn.datasets import fetch_lfw_people
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
import numpy as np
faces = fetch_lfw_people(min_faces_per_person=60)
X = faces.data
fig, axes = plt.subplots(4,5
,figsize=(8,4)
,subplot_kw = {"xticks":[],"yticks":[]})
#subplot_kw控制坐标轴为空
#fig 画布
#axes 生成的子图对象
fig, axes = plt.subplots(3,8
,figsize=(8,4)
,subplot_kw = {"xticks":[],"yticks":[]})
for i, ax in enumerate(axes.flat):
ax.imshow(faces.images[i,:,:],cmap="gray")
pca = PCA(150).fit(X)
V = pca.components_
V.shape
fig, axes = plt.subplots(3,8
,figsize=(8,4)
,subplot_kw = {"xticks":[],"yticks":[]})
for i, ax in enumerate(axes.flat):
ax.imshow(V[i,:].reshape(62,47),cmap="gray")
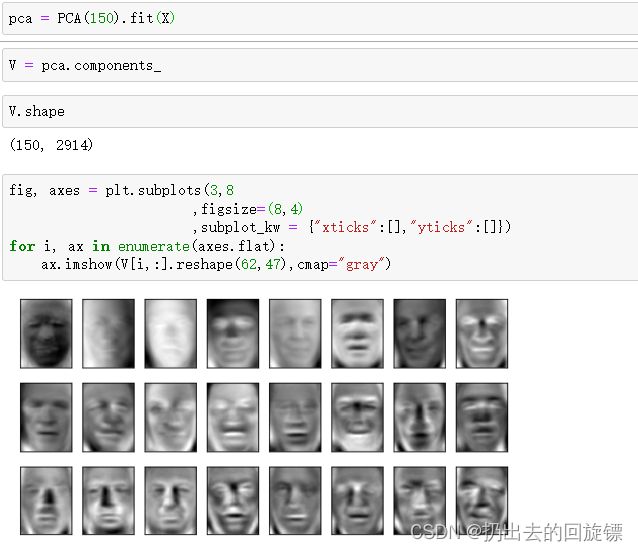
相对来说,眼睛,鼻子,嘴巴比较清晰,然而脸的轮廓和头发就变的模糊了。说明性特征的特征向量大多根据“五官”和“亮度”相关的特征中提取出来
步骤四:将降维后的矩阵还原图像查看信息保存量
x_dr = pca.fit_transform(X)
X_inverse = pca.inverse_transform(x_dr)
fig, ax = plt.subplots(2,10,figsize=(10,2.5)
,subplot_kw={"xticks":[],"yticks":[]}
)
for i in range(10):
ax[0,i].imshow(faces.images[i,:,:],cmap="binary_r")
ax[1,i].imshow(X_inverse[i].reshape(62,47),cmap="binary_r")
虽然大部分相似,但是明显舍弃了部分信息。即降维不是完全可逆,但是也的确保留了原数据的大部分信息
PCA手写字噪音过滤:
步骤一:导入所需库和数据
from sklearn.datasets import load_digits
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
import numpy as np
digits = load_digits()
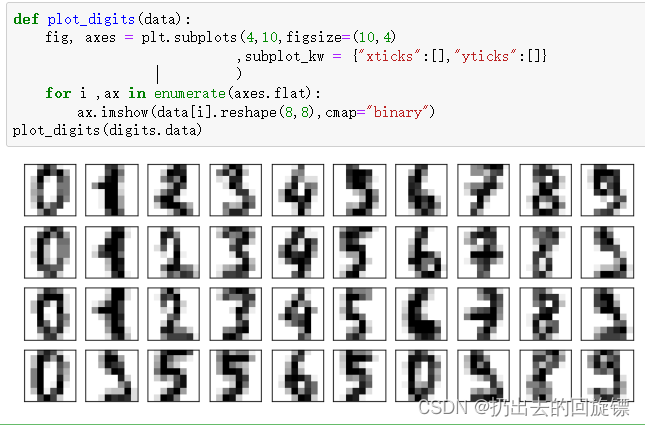
def plot_digits(data):
fig, axes = plt.subplots(4,10,figsize=(10,4)
,subplot_kw = {"xticks":[],"yticks":[]}
)
for i ,ax in enumerate(axes.flat):
ax.imshow(data[i].reshape(8,8),cmap="binary")
plot_digits(digits.data)
np.random.RandomState(42)
noisy = np.random.normal(digits.data,2)
plot_digits(noisy)
pca = PCA(0.5).fit(noisy)
X_dr = pca.transform(noisy)
X_dr.shape
without_noise = pca.inverse_transform(X_dr)
plot_digits(without_noise)
4.PCA实现784个特征的手写字降维
步骤一:导入库和数据
from sklearn.decomposition import PCA
from sklearn.ensemble import RandomForestClassifier as RFC
from sklearn.model_selection import cross_val_score
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
data = pd.read_csv(r".\digit recognizor.csv")
X = data.iloc[:,1:]
y = data.iloc[:,0]
pca_line = PCA().fit(X)
plt.figure(figsize=[20,5])
plt.plot(np.cumsum(pca_line.explained_variance_ratio_))
plt.xlabel("number of components after dimension reduction")
plt.ylabel("cumulative explained variance ratio")
plt.show()
#======【TIME WARNING:2mins 30s】======#
score = []
for i in range(1,101,10):
X_dr = PCA(i).fit_transform(X)
once = cross_val_score(RFC(n_estimators=10,random_state=0)
,X_dr,y,cv=5).mean()
score.append(once)
plt.figure(figsize=[20,5])
plt.plot(range(1,101,10),score)
plt.show()
score = []
for i in range(10,25):
X_dr = PCA(i).fit_transform(X)
once = cross_val_score(RFC(n_estimators=10,random_state=0),X_dr,y,cv=5).mean()
score.append(once)
plt.figure(figsize=[20,5])
plt.plot(range(10,25),score)
plt.show()
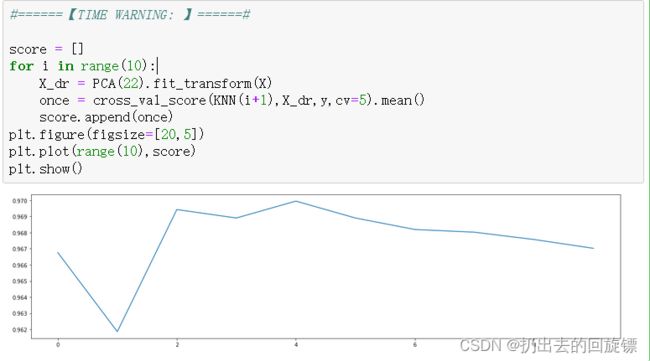
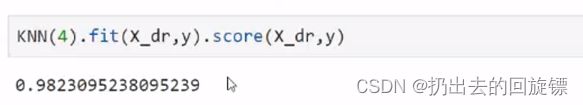
步骤四:验证22维的准确性,以及其他算法
X_dr = PCA(22).fit_transform(X)
#======【TIME WARNING:1mins 30s】======#
cross_val_score(RFC(n_estimators=100,random_state=0),X_dr,y,cv=5).mean()
from sklearn.neighbors import KNeighborsClassifier as KNN
cross_val_score(KNN(),X_dr,y,cv=5).mean()#KNN()的值不填写默认=5
#======【TIME WARNING: 】======#
score = []
for i in range(10):
X_dr = PCA(22).fit_transform(X)
once = cross_val_score(KNN(i+1),X_dr,y,cv=5).mean()
score.append(once)
plt.figure(figsize=[20,5])
plt.plot(range(10),score)
plt.show()
KNN(4).fit(X_dr,y).score(X_dr,y)