脏数据-异常值处理

点击上方蓝色字体,关注我们

作者:勾蒙蒙,R语言资深爱好者。
个人公众号: R语言及生态系统服务。
异常值(outlier)是指一组测定值中与平均值的偏差超过两倍标准差的测定值,与平均值的偏差超过三倍标准差的测定值,称为高度异常的异常值。
科学数据中异常值总是无处不在,可能是仪器造成的异常,亦或是观测的异常。暂且不管异常值从哪来,我们今天探讨异常值的识别以及异常值该往哪去?难道异常值就只有删除这种简单而粗暴的方法吗?其实不然!
—探寻异常值—
1、箱型图检验异常值
x<-c(1,4,2,-13,5,6,1,2,8,10,22)
boxplot(x,col="yellow")
boxplot.stats(x)$out
输出结果为-13、22为异常值。用如上单变量的异常值监测办法,稍作延伸,就可以把多个变量共同异常值的样本找出:
##生成数据
x<-c(1,4,2,-13,5,6,1,2,8,10,22)
y<-c(2,9,3,4,6,2,8,11,13,15,-9)
Data<-cbind(x,y)
##找出x,y异常值所在的行
a <- which(x %in%boxplot.stats(x)$out)
b <- which(y %in%boxplot.stats(y)$out)
##将ab进行相交,找出x,y均为异常值的行
outlier.line<-intersect(a,b)
##进行制图直观感觉
plot(Data,pch=16,cex=1.5)
##标注异常点(x,y均为异常,图中以红色加号表示)
points(Data[outlier.line,],col="red",pch="+",cex=3.5)
box(lwd=2)
##标注异常点(x异常或者y异常,图中以蓝色加号表示)
##将ab进行联合
outlier.line1<-union(a,b)
points(Data[outlier.line1,],col="blue",pch="+",cex=3.5)
box(lwd=2)
2、局部异常因子(LOF)法
Local Outlier Factor(LOF)算法是基于密度的经典算法,其核心的部分是关于数据点密度的刻画,相对于其他的聚类分析等方法,其更简单、直观,且不需要太多的数据的分布有太多的要求,其最大的特点是可以量化每个数据的异常程度。
如下图所示,对于C1的数据集合,整体间距、密度及分散程度等均相对均匀一致,可以将其认为是一簇;对于C2集合的点,同样可认为是一簇;那么O1和O2点相对独立,其可以被认定为异常值或离散值,现在的问题是,如何实现算法的通用性,可以满足C1和C2这种密度分散情况迥异的集合的异常点识别。
(摘自Wangyibo0201的博客,https://blog.csdn.net/wangyibo0201/article/details/51705966)
##生成随机数据
a<-set.seed(665544)
n<-100
x <- cbind( x=runif(10, 0,5) + rnorm(n, sd=0.4),y=runif(10, 0, 5) + rnorm(n, sd=0.4))
x
##作图直观看
plot(x,pch=16,col="blue",cex=2)
box(lwd=2)
##LOF局部分析
install.packages("DMwR")
library(DMwR)
##计算每个样本的LOF值,其中k值指的是计算LOF值时使用的周围邻居点的数量
out.score<-lofactor(x,k=5)
##制作LOF值概率分布图
plot(density(out.score))
box(lwd=2)
##将LOF值排在前五位的作为异常值,提取其样方号
outlier<-order(out.score,decreasing=TRUE)[1:5]
outlier
##由此可得出异常值数据
x[outlier,]
3、K-means算法
通过聚类分析进行异常值的检测通过把数据聚成类,将不属于任务一类的数据当作为异常值。使用K-mean算法,数据共被分为K组,通过把它们分配到最近的聚类中心。然后,我们能够计算每个对象到聚类中心的距离(或相似性),并且选择最大的距离作为异常值。
##生成数据
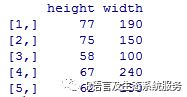
height<-c(65,73,59,61,75,67,68,70,62,66,77,75,74,70,61,58,66,59,68,61)
width<-c(220,160,110,120,150,240,230,220,130,210,190,180,170,210,110,100,230,120,210,130)
Data<-cbind(height,width)
##查看分布情况
plot(Data,cex=1.5,pch=16)
从图中的数据分布来看,数据大致可以分为3个类别
kmeans.result<-kmeans(Data,centers=3)
kmeans.result
##输出聚类中心
kmeans.result$centers
##输出聚类结果
kmeans.result$cluster
![]()
##计算每个样本对应的聚类中心的样本,即对聚类中心进行样本间的对应
centers<-kmeans.result$centers[kmeans.result$cluster,]
##计算每个样本到其聚类中心的距离
distances<-sqrt(rowSums(Data-centers)^2)
这一步的操作不懂的也可参考下图:
(摘自Wangyibo0201的博客,https://blog.csdn.net/wangyibo0201/article/details/51705966)
##同LOF方法一样,也是找到排在前五位的将其作为异常值
outlier<-(order(distances,decreasing=TRUE))[1:5]
outlier
![]()
##输出异常值
Data[outlier,]
##绘制聚类的结果
plot(Data[-outlier,],cex=3,pch=16)
#聚类中心
points(kmeans.result$centers,pch=8,col="red",cex=3)
#异常值
points(Data[outlier,],pch=4,cex=3,col="red")
box(lwd=2)
4、稳健马氏距离法
将所有的数据不分类别的当成样本的总体,计算每个样本点与样本中心的马氏距离,以马氏距离的大小衡量某个样本的异常程度。马氏距离可不受量纲的影响,而且在多元的条件下,马氏距离将各变量的相关性考虑在内,因此,比欧式距离受欢迎些。
但是一般马氏距离检测方法也是不稳定的,个别异常值会把均值向量和协方差矩阵向自己方向吸引,这样算出来的样本马氏距离起不了检测异常值的所用。因此,稳健马氏距离的首要目的是利用迭代和一般马氏距离的思想构造一个稳健的协方差矩阵估计量和稳健的均值向量。
R语言的“mvoutlier”程序包在卡方分布的基础上对基于稳健马氏距离判定异常值的算法做了进一步的修正,同时其可支持多种计算和画图功能,在多元异常值检验中地位凸显。
数据来源:全国主要流域重点断面水质自动检测周报(2018.04.19-2018.05.10)
http://datacenter.mep.gov.cn/websjzx/queryIndex.vm
数据来源描述:
其中Ph为酸碱度
DO为溶解氧浓度
MnO4为高锰酸盐指数
N为氨氮浓度
##数据读取
Data<-read.csv("Environmentdataset.csv",head=TRUE)
newdata<-Data[,c(9:12)]
View(newdata)
##一维上直观观测数据
library(mvoutlier)
uni.plot(newdata)
##返回异常值的编号
which(try1$outliers==T)
图中的红点表示为疑似异常值,其偏离均值的距离相对较大,此例子中我们更侧重于多元异常值的检验,用aq.plot()函数进行基于稳健马氏距离的异常值监测。
##基于稳健马氏距离的多元异常值检测过程
try2<-aq.plot(newdata)
##返回异常值的编号
which(try2$outliers==T)
在此图中可以看出,绿色颜色的样本为正常值,而红色颜色的样本为异常值,但是从图上也可以看出,也有一些正常值被误判为异常值了,因此需要调整aq.plot()
函数中的参数,quan:表示用于预测MAC值的数据的比例,默认值为0.5,最大值为1;alpha:表示最大阈值比例,默认值为0.025。
上述用到的例子仅仅是对少数变量进行多元异常值检验,但往往在实验或观测中的变量有非常多,导致数据稀疏,导致距离的意义不大。“mvoutlier”程序包提供了一种类似于主成分分析将维的思路进行多元异常值检验,其函数为pcout()。
—处理异常值—
1、均值替换法
2、回归法插补
3、多重插补法
上篇文章对缺失值均值替换、回归插补已经做了详细的介绍,此章着重对多重插补法处理异常值进行详细说明,当然了其也可以用到缺失值处理中。
多重插补的思路:
mice包多重插补:
对数值型数据默认使用随机回归添补法;
对二元因子数据,默认使用Logistic回归添补法;
对多元因子默认使用分类回归添补法;
详情可参考:
https://cran.r-project.org/web/packages/mice/mice.pdf
往期回顾:
地形图绘制
R语言——初步认识
脏数据-数据量纲差异
脏数据-缺失值处理
公众号后台回复关键字即可学习
回复 爬虫 爬虫三大案例实战
回复 Python 1小时破冰入门
回复 数据挖掘 R语言入门及数据挖掘
回复 人工智能 三个月入门人工智能
回复 数据分析师 数据分析师成长之路
回复 机器学习 机器学习的商业应用
回复 数据科学 数据科学实战
回复 常用算法 常用数据挖掘算法