Python数据分析案例18——化学分子数据模型(机器学习分类问题全流程)
1. 引言
1.1设计背景
对分子进行分类,对于筛选特定疾病的候选药物是至关重要的。传统的机器学习算法可以对分子进行分类,但是分子不能直接作为机器学习模型的输入,需要进行大量的实验从分子中得到一系列的分子特性。将分子特征使用数字化进行处理,挖掘出数据中的特征信息,从而对分子不同类别进行一个很好的区分。
1.2设计目的和意义
本设计利用大量的分子的不同特征变量数据,进行有监督的机器学习模型构建,结合训练集上有类别标签的数据进行模型的迭代拟合,训练出可以智能识别分类别的机器学习模型。本设计主要的三点意义如下:
1.对分子特征数据进行探索,需要大量的分子的特征分布特点。
2.利用K折交叉验证、超参数搜索寻找最优的适合分子类别预测的机器学习和其超参数。
3.训练构建最优的分子类别预测的机器学习模型,并在验证集上进行预测。
2. 设计思路与流程
一般机器学习的数据科学项目流程主要分为数据的预处理,初步分析探索和可视化,特征工程的构建,异常值处理,之后进行模型选择和超参数调整。本设计的整体思路流程如下:
后续代码实现和论文撰写都按照这个思路来。
3. 数据探索与分析
3.1数据读取和预处理
导入数据分析常用的包
#导入数据分析常用包
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
plt.rcParams['font.sans-serif'] = ['KaiTi'] #中文
plt.rcParams['axes.unicode_minus'] = False #负号读取数据
data=pd.read_excel('分子类型预测 for students.xlsx',sheet_name=0)
data2=pd.read_excel('分子类型预测 for students.xlsx',sheet_name=1)查看数据前五行
data.head()查看训练集和验证集数据基础信息
data=data.infer_objects()
data2=data2.infer_objects()
data.info() ,data2.info()上述图左边是训练集信息,可以看到数据总共有17999条样本,变量一共20个,其中Type是响应变量,其余19个为特征变量。变量类型有整数型和浮点数,也有分类的文本型。由于模型不能直接计算文本数据,所有对于文本分类变量需要进一步处理。整体数据所有变量都没有缺失值,不用进行填充处理。
上述图右边是需要预测的验证集信息,可以看到数据总共有3735条样本,变量一共也是20个,其中Type是响应变量是全部空值,因为这是需要我们进行预测的。验证集的数据也没有缺失值,并且特征变量和训练集是一一对应的。
整体的所有变量信息总结如下表:
| 变量名称 |
含义 |
变量类型 |
| formula |
分子化学式 |
文本变量(唯一取值) |
| C |
碳元素个数 |
数值变量 |
| H |
氢元素个数 |
数值变量 |
| O: |
氧元素个数 |
数值变量 |
| N |
氮元素个数 |
数值变量 |
| S |
硫元素个数 |
数值变量 |
| Group |
分子所在的组别 |
分类变量 |
| AImod |
修正后的芳香指数 |
数值变量 |
| DBE |
不饱和双键当量 |
数值变量 |
| MZ |
质荷比 |
数值变量 |
| OC |
氧碳比 |
数值变量 |
| HC |
氢碳比 |
数值变量 |
| SC |
硫碳比 |
数值变量 |
| NC |
氮碳比 |
数值变量 |
| NOSC |
氧化态指数 |
数值变量 |
| DBEC |
不饱和双键当量与碳的差值 |
数值变量 |
| DBEO |
不饱和双键当量与氧的差值 |
数值变量 |
| Location |
位置 |
分类变量 |
| Sample |
所样品名称 |
分类变量 |
| Type |
分子类型 |
分类变量 |
根据上面变量信息可以了解每个变量的特点。其中formula是分子表达式,对于预测没有帮助,要删除。验证集的formula要保留,用于提交。
变量group,location,sample是分类型变量,需要做独立热编码处理。其余变量是数值型变量,可以直接使用。
Type是响应变量,需要作为y单独取出。由于y是分类变量,所以这是一个分类问题。
#验证集ID留着后面提交
formula=data2['formula']
y=data['Type']
data.drop(['formula','Type'],axis=1,inplace=True)
data2.drop(['formula','Type'],axis=1,inplace=True)分类变量进行独立热编码
#生成虚拟变量
data=pd.get_dummies(data)
data2=pd.get_dummies(data2)将上述特征变量处理完后,下面分别对特征向量X和响应变量y进行数据的初步探索和可视化分析。
3.2数据探索和可视化
3.2.1特征变量的描述性统计
利用descirbe方法能很快的计算所有特征变量的基础统计量,均值,方差,最小值,分位数等。
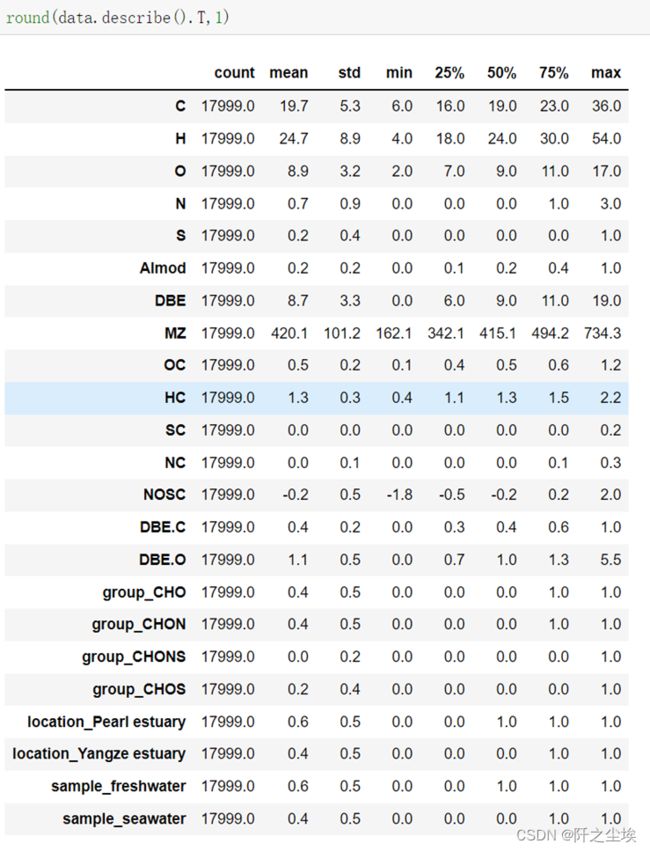
round(data.describe().T,1)我们可以从上图看出每个特征变量的取值范围,均值和方差的大小。其中观察分类变量,看到group独立热编码出来了4个哑变量,location和sample独热出来了2个哑变量。哑变量的取值只有0和1两个数值。验证集的描述性统计也和上图相似。
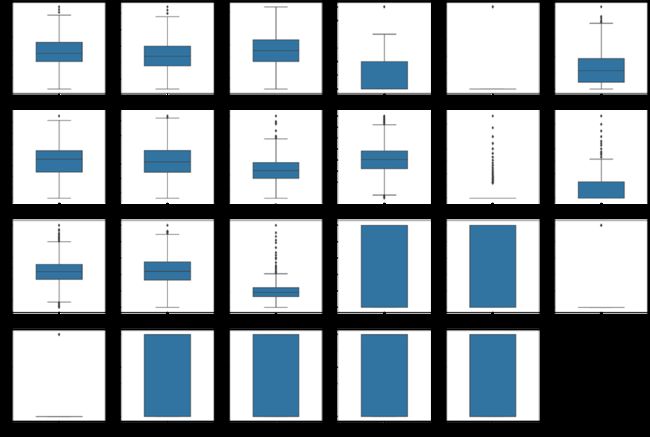
由于数据不是很直观,进一步对特征变量数据可视化,采用箱线图进行分析:
#查看特征变量的箱线图分布
columns = data.columns.tolist() # 列表头
dis_cols = 6 #一行几个
dis_rows = len(columns)
plt.figure(figsize=(4 * dis_cols, 4 * dis_rows),dpi=256)
for i in range(len(columns)):
plt.subplot(dis_rows,dis_cols,i+1)
sns.boxplot(data=data[columns[i]], orient="v",width=0.5)
plt.xlabel(columns[i],fontsize = 20)
plt.tight_layout()
#plt.savefig('特征变量箱线图.jpg',dpi=512)
plt.show()从上图可以看到大多数变量分布都还是服从正态分布的,比如C,H,O,DBE,MZ这几个变量分布的箱线图没有异常值,箱体均值都在中央。少数变量如S,SC,NC,DBE.O具有较多的异常值,S和SC最为严重,箱体变成了一条线,说明少数分子里面的硫分子和硫碳比很高。 分类变量里面group变量里面类型为CHONS和CHOS的分子较少,所以基本看不到箱体。
进一步,为了对比训练集和验证集的特征变量分布是否相同,我们画出两者的核密度图进行对比:
#画密度图,训练集和测试集对比
dis_cols = 6 #一行几个
dis_rows = len(columns)
plt.figure(figsize=(4 * dis_cols, 4 * dis_rows),dpi=256)
for i in range(len(columns)):
ax = plt.subplot(dis_rows, dis_cols, i+1)
ax = sns.kdeplot(data[columns[i]], color="Red" ,shade=True)
ax = sns.kdeplot(data2[columns[i]], color="Blue",warn_singular=False,shade=True)
ax.set_xlabel(columns[i],fontsize = 20)
ax.set_ylabel("Frequency",fontsize = 18)
ax = ax.legend(["train", "test"])
plt.tight_layout()
#plt.savefig('训练测试特征变量核密度图.jpg',dpi=500)
plt.show()可以看到训练集和验证集的数据的分布基本都是一致的,不需要进行处理。
其中C,H,O,DBE,MZ,OC,HC,NOSC,DBE.C这几个变量和前面的结论一致,从分布形状上来看服从正态分布。S,SC,NC,DBE.O是具有明显的右偏分布,说明数据存在较多的异常值。下面呈现两峰分布的变量都是分类的哑变量。
3.2.2响应变量的描述性统计
首先计算响应变量y的每个类别在训练集的占比数量:
y.value_counts(normalize=True)| 类别 |
占比 |
| resistant |
0.456248 |
| labile |
0.312628 |
| product |
0.231124 |
画出其柱状图,饼图和核密度图:
# 查看y的分布
#分类问题
plt.figure(figsize=(8,3),dpi=128)
plt.subplot(1,3,1)
y.value_counts().plot.bar(title='响应变量柱状图图')
plt.subplot(1,3,2)
y.value_counts().plot.pie(title='响应变量饼图')
plt.subplot(1,3,3)
y.value_counts().plot.kde(title='响应变量核密度图')
#plt.savefig('响应变量.png')
plt.tight_layout()
plt.show()可以看到在训练集17999条样本中,类别为resistant的分子占比最多,占比达到45%,总共有八千条左右,labile将近六千条,product最少将近四千条。类别比例较为平衡。
从上述分析中我们得知特征向量里面存在着异常值,训练数据中的异常值会影响模型的学习表示和泛化能力。所以下面我们对异常值要进行处理。
3.2.3相关性分析
画出所有特征变量的皮尔逊相关系数的热力图如上图所示,我们可以清楚的看到每个变量之间的相关性大小。C和H两个变量的性关系将近0.8,说明分子里面的碳氢构成的结构较多,MZ质荷比和C,H,O三种原子的相关性很高,说明分子里面的C,H,O分子较多,则质荷比也会很高。NC和N相关性高达0.95,SC和S的相关性也高达0.96。DBE.C和HC的相关性是负数,为-0.97,呈现严格的负相关关系。HC和AImod——修正后的芳香指数也是高度的负相关。其他存在-1相关系数的是一个分类变量的不同的哑变量。验证集的特征变量相关性和训练集也差不多,不展示了。
可以看到相关性高的还是C,H,O这些变量,数据的分布和相关性是一样的,模型能很好的具有泛化能力。
下面加入y来研究特征向量的区别,即在响应变量不同的时候的X分布的差异。我们对不同类别的分子画出他们的所有特征变量的小提琴图如下:
#查看特征变量的箱线图分布
columns = data.columns.tolist() # 列表头
dis_cols = 5 #一行几个
dis_rows = len(columns)
plt.figure(figsize=(4 * dis_cols, 4 * dis_rows),dpi=256)
for i in range(len(columns)):
plt.subplot(dis_rows,dis_cols,i+1)
sns.violinplot(x='Y',y=columns[i],width=0.8,saturation=0.9,lw=0.8,palette="Set2",orient="v",inner="box",data=data.assign(Y=y))
#plt.xlabel((y.unique().tolist()),fontsize=12)
plt.ylabel(columns[i], fontsize=18)
plt.tight_layout()
plt.show()绿色的小提琴图代表resistant类别的分子,红色的代表product类别分子, labile为蓝色的小提琴图。从上图可以看到,不同的分子的C,H,O,N,S的数量分布具有明显的不同。例如product类别分子的C含量明显要低于其他两中分子。resistant分子的S和N含量会较低,labile的Almod的分布较为分散,而product类别的分子的Almod的数值主要是0。labile的DBE明显高于其他两中分子,OC含量低于其他两种分子。并且labile的DBE.C和DBE.O两个特征比起其他两种分子都较为分散,而且均值数值要略微大一点。
综上所述,这些特征变量对于不同类别的分子还是具有一定的区分度的,模型一个能从不同的特征里面学到不同的分子的特点,从而进行较好的分类效果。
3.3异常值处理
X=data.copy()
X2=data2.copy()由于数据的每一个特征变量单位不一样,为了方便对比,将所有的数据进行标准化,减去均值除以标准差,转化为标准正态分布,然后画图展示如下:
#X异常值处理,先标准化
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
X_s = scaler.fit_transform(X)
X2_s = scaler.fit_transform(X2)#然后画图查看
plt.figure(figsize=(20,8))
plt.boxplot(x=X_s,labels=data.columns)
plt.hlines([-6,6],0,len(columns))
plt.xticks(rotation=40)
#plt.savefig('特征变量标准化箱线图.png',dpi=256)
plt.show() 可以看到较多特征变量的离散值较多,需要对这些有异常值的样本进行筛选,最好进行删除。
可以看到较多特征变量的离散值较多,需要对这些有异常值的样本进行筛选,最好进行删除。
本设计根据三西格玛原则,选择那些值在三倍标准差之外的样本进行删除处理。
#异常值多的列进行处理
def deal_outline(data,col,n): #数据,要处理的列名,几倍的方差
for c in col:
mean=data[c].mean()
std=data[c].std()
data=data[(data[c]>mean-n*std)&(data[c]#超过3倍方差进行删除
X=deal_outline(X,X.columns,3)
y=y[X.index]
X.shape,y.shape然后将筛选出来的样本的索引赋值给y,最终留下的样本量为17996条。
最后由于y是文本变量,模型不能直接进行运算,需要进行映射处理,我们将响应变量的三类['resistant', 'product', 'labile']对应因子化,映射为[0,1,2]。
#最后为了让模型进行运算,将y变为数值型的分类变量,进行映射
codes,uniques=pd.factorize(y)
print(uniques)
y=codes到这里所有的数据预处理,特征工程都构建完毕,可以进行下面的建模实现过程。
4. 模型实现过程
4.1模型或方法介绍
4.1.1模型介绍
由于需要筛选出最优的机器学习模型,所以首先我们要初步探索不同模型在这个数据集上的表现能力。选择随机将训练数据化为训练集和测试集数据,训练集数据对模型进行训练,测试集数据来验证模型的准确率。
本设计共选择了10种机器学习常见的分类模型,如下表:
| 模型种类 |
||||
| '逻辑回归' |
'线性判别' |
'K近邻' |
'决策树' |
'随机森林' |
| '梯度提升' |
'极端梯度提升' |
'轻量梯度提升' |
'支持向量机' |
'神经网络' |
4.1.2评价指标介绍
本文是一个分类问题,采用四个分类问题常用而且可靠的评价指标,准确率、精确度、召回率和F1值对模型进行全面的评价。四个指标的计算公式如下:
其中:TP(True Positives):真正例,预测为正例而且实际上也是正例;FP(False Positives):假正例,预测为正例然而实际上却是负例;FN(false Negatives):假负例,预测为负例然而实际上却是正例;TN(True Negatives):真负例,预测为负例而且实际上也是负例。
4.2具体实现步骤
我们在80%数据上进行训练,在20%的数据上进行测试,并采用准确率、精确度、召回率和F1值四个分类问题常用而且可靠的评价指标对模型进行全方位的评价,然后在此基础上进行模型的选择。
选择模型后进一步对模型进行K折交叉验证,使用全部数据的信息,更加可靠的选择最终的预测模型。这一过程可以会有些费时。
最后使用随机搜索和网格化超参数搜索寻找最优模型的最优参数数,在所有数据上进行训练,对验证集进行预测。
#划分训练集和测试集
from sklearn.model_selection import train_test_split
X_train,X_test,y_train,y_test=train_test_split(X,y,stratify=y,test_size=0.2,random_state=0)#数据标准化
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
scaler.fit(X_train)
X_train_s = scaler.transform(X_train)
X_test_s = scaler.transform(X_test)
X2_s=scaler.transform(data2)
print('训练数据形状:')
print(X_train_s.shape,y_train.shape)
print('测试和验证数据形状:')
print(X_test_s.shape,y_test.shape,X2_s.shape)上述代码是先划分训练集和测试集,测试集的比例为20%,随机数种子为0。然后进行数据的标准化,打印查看训练集数据,测试集数据的形状。可以看到我们有14396条训练集,测试集3600条,特征变量有23个。需要预测的有3735条数据。
#采用十种模型,对比验证集精度
from sklearn.linear_model import LogisticRegression
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
from sklearn.neighbors import KNeighborsClassifier
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.ensemble import GradientBoostingClassifier
from xgboost.sklearn import XGBClassifier
from lightgbm import LGBMClassifier
from sklearn.svm import SVC
from sklearn.neural_network import MLPClassifier#逻辑回归
model1 = LogisticRegression(C=1e10,max_iter=10000)
#线性判别分析
model2 = LinearDiscriminantAnalysis()
#K近邻
model3 = KNeighborsClassifier(n_neighbors=10)
#决策树
model4 = DecisionTreeClassifier(random_state=77)
#随机森林
model5= RandomForestClassifier(n_estimators=1000, max_features='sqrt',random_state=10)
#梯度提升
model6 = GradientBoostingClassifier(random_state=123)
#极端梯度提升
model7 = XGBClassifier(use_label_encoder=False,eval_metric=['logloss','auc','error'],
objective='multi:softmax',random_state=0)
#轻量梯度提升
model8 = LGBMClassifier(objective='multiclass',num_class=3,random_state=1)
#支持向量机
model9 = SVC(kernel="rbf", random_state=77)
#神经网络
model10 = MLPClassifier(hidden_layer_sizes=(16,8), random_state=77, max_iter=10000)
model_list=[model1,model2,model3,model4,model5,model6,model7,model8,model9,model10]
model_name=['逻辑回归','线性判别','K近邻','决策树','随机森林','梯度提升','极端梯度提升','轻量梯度提升','支持向量机','神经网络']计算所有评价指标,定义评估函数
from sklearn.metrics import confusion_matrix
from sklearn.metrics import classification_report
from sklearn.metrics import cohen_kappa_score
from sklearn.model_selection import KFolddef evaluation(y_test, y_predict):
accuracy=classification_report(y_test, y_predict,output_dict=True)['accuracy']
s=classification_report(y_test, y_predict,output_dict=True)['weighted avg']
precision=s['precision']
recall=s['recall']
f1_score=s['f1-score']
#kappa=cohen_kappa_score(y_test, y_predict)
return accuracy,precision,recall,f1_score #, kappa
def evaluation2(lis):
array=np.array(lis)
return array.mean() , array.std()训练拟合模型,计算评价指标
df_eval=pd.DataFrame(columns=['Accuracy','Precision','Recall','F1_score'])
for i in range(10):
model_C=model_list[i]
name=model_name[i]
model_C.fit(X_train_s, y_train)
pred=model_C.predict(X_test_s)
s=classification_report(y_test, pred)
s=evaluation(y_test,pred)
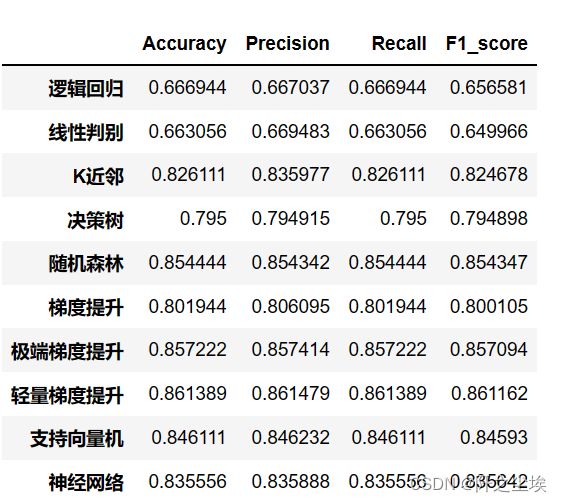
df_eval.loc[name,:]=list(s)查看:
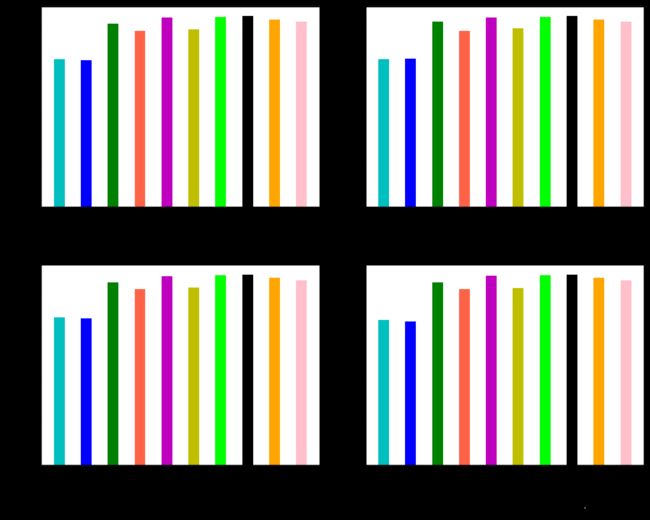
df_eval进一步,对上面的数据可视化,画出柱状图分布对比:
bar_width = 0.4
colors=['c', 'b', 'g', 'tomato', 'm', 'y', 'lime', 'k','orange','pink','grey','tan']
fig, ax = plt.subplots(2,2,figsize=(10,8),dpi=128)
for i,col in enumerate(df_eval.columns):
n=int(str('22')+str(i+1))
plt.subplot(n)
df_col=df_eval[col]
m =np.arange(len(df_col))
plt.bar(x=m,height=df_col.to_numpy(),width=bar_width,color=colors)
#plt.xlabel('Methods',fontsize=12)
names=df_col.index
plt.xticks(range(len(df_col)),names,fontsize=10)
plt.xticks(rotation=40)
plt.ylabel(col,fontsize=14)
plt.tight_layout()
#plt.savefig('柱状图.jpg',dpi=512)
plt.show()对于这种表格数据,一般经验来说,集成方法都最好。本设计的实验结果来看,在验证集上的表现最好的是随机森林,极端梯度提升,轻量梯度提升三个集成模型。其准确率、精确度、召回率和F1值综合来看最高。后面只选三个模型,随机森林,极端梯度提升,轻量梯度提升进行交叉验证,来进一步选择最优模型。
4.4.1 重复K折交叉验证
我们定义了一个重复K折交叉验证的函数,将三个模型分别都进行6次5折交叉验证,并且计算每一次的准确率、精确度、召回率和F1值,画出混淆矩阵和其评价指标。
def cross_val(model=None,X=None,Y=None,K=5,repeated=1,show_confusion_matrix=True):
df_mean=pd.DataFrame(columns=['Accuracy','Precision','Recall','F1_score'])
df_std=pd.DataFrame(columns=['Accuracy','Precision','Recall','F1_score'])
for n in range(repeated):
print(f'正在进行第{n+1}次重复K折.....随机数种子为{n}\n')
kf = KFold(n_splits=K, shuffle=True, random_state=n)
Accuracy=[]
Precision=[]
Recall=[]
F1_score=[]
print(f" 开始本次在{K}折数据上的交叉验证.......\n")
i=1
for train_index, test_index in kf.split(X):
print(f' 正在进行第{i}折的计算')
X_train=X.to_numpy()[train_index]
y_train=np.array(y)[train_index]
X_test=X.to_numpy()[test_index]
y_test=np.array(y)[test_index]
model.fit(X_train,y_train)
pred=model.predict(X_test)
score=list(evaluation(y_test,pred))
Accuracy.append(score[0])
Precision.append(score[1])
Recall.append(score[2])
F1_score.append(score[3])
if show_confusion_matrix:
#数据透视表,混淆矩阵
print("混淆矩阵:")
table = pd.crosstab(y_test, pred, rownames=['Actual'], colnames=['Predicted'])
#print(table)
plt.figure(figsize=(4,3))
sns.heatmap(table,cmap='Blues',fmt='.20g', annot=True)
plt.tight_layout()
plt.show()
#计算混淆矩阵的各项指标
print('混淆矩阵的各项指标为:')
print(classification_report(y_test, pred))
print(f' 第{i}折的准确率为:{round(score[0],4)},Precision为{round(score[1],4)},Recall为{round(score[2],4)},F1_score为{round(score[3],4)}')
i+=1
print(f' ———————————————完成本次的{K}折交叉验证———————————————————\n')
Accuracy_mean,Accuracy_std=evaluation2(Accuracy)
Precision_mean,Precision_std=evaluation2(Precision)
Recall_mean,Recall_std=evaluation2(Recall)
F1_score_mean,F1_score_std=evaluation2(F1_score)
print(f'第{n+1}次重复K折,本次{K}折交叉验证的总体准确率均值为{Accuracy_mean},方差为{Accuracy_std}')
print(f' 总体Precision均值为{Precision_mean},方差为{Precision_std}')
print(f' 总体Recall均值为{Recall_mean},方差为{Recall_std}')
print(f' 总体F1_score均值为{F1_score_mean},方差为{F1_score_std}')
print("\n====================================================================================================================\n")
df1=pd.DataFrame(dict(zip(['Accuracy','Precision','Recall','F1_score'],[Accuracy_mean,Precision_mean,Recall_mean,F1_score_mean])),index=[n])
df_mean=pd.concat([df_mean,df1])
df2=pd.DataFrame(dict(zip(['Accuracy','Precision','Recall','F1_score'],[Accuracy_std,Precision_std,Recall_std,F1_score_std])),index=[n])
df_std=pd.concat([df_std,df2])
return df_mean,df_std运行一次的截图如下:
model = LGBMClassifier(objective='multiclass',num_class=3,random_state=1)
lgb_crosseval,lgb_crosseval2=cross_val(model=model,X=X,Y=y,K=5,repeated=6)上图只展示了一次重复K折交叉验证的过程,更多的混淆矩阵和评价指标的计算结果太长,受于篇幅就不展示,可以在代码文件里面查看。
查看每次K折交叉验证的评价指标的运行结果
lgb_crosseval计算XGoost的K折交叉验证
model = XGBClassifier(use_label_encoder=False,eval_metric=['logloss','auc','error'],
objective='multi:softmax',random_state=0)
xgb_crosseval,xgb_crosseval2=cross_val(model=model,X=X,Y=y,K=5,repeated=6,show_confusion_matrix=False)随机森林的K折交叉验证
model = RandomForestClassifier(n_estimators=500, max_features='sqrt',random_state=0)
rf_crosseval,rf_crosseval2=cross_val(model=model,X=X,Y=y,K=5,repeated=6,show_confusion_matrix=False)最后对三个模型的每一次K折交叉验证的四个指标计算其均值和方差,来全方面地对比他们的预测性能。
plt.subplots(1,4,figsize=(16,3),dpi=128)
for i,col in enumerate(lgb_crosseval.columns):
n=int(str('14')+str(i+1))
plt.subplot(n)
plt.plot(lgb_crosseval[col], 'k', label='LGB')
plt.plot(xgb_crosseval[col], 'b-.', label='XGB')
plt.plot(rf_crosseval[col], 'r-^', label='RF')
plt.title(f'不同模型的{col}对比')
plt.xlabel('重复交叉验证次数')
plt.ylabel(col,fontsize=16)
plt.legend()
plt.tight_layout()
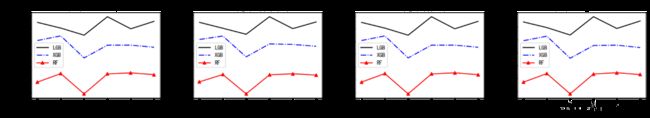
plt.show()黑色的实线是LGBM模型,蓝色的虚线是XGB模型,红色的点线是RF模型。从上图可以看到从准确率、精确度、召回率和F1值四个指标上来看,LGBM在每一次的K折交叉验证中都全面比其他两个模型高。说明LGBM模型在这个数据集上的表现能力由于其他模型。
再来看他们的方差图对比:
plt.subplots(1,4,figsize=(16,3),dpi=128)
for i,col in enumerate(lgb_crosseval2.columns):
n=int(str('14')+str(i+1))
plt.subplot(n)
plt.plot(lgb_crosseval2[col], 'k', label='LGB')
plt.plot(xgb_crosseval2[col], 'b-.', label='XGB')
plt.plot(rf_crosseval2[col], 'r-^', label='RF')
plt.title(f'不同模型的{col}方差对比')
plt.xlabel('重复交叉验证次数')
plt.ylabel(col,fontsize=16)
plt.legend()
plt.tight_layout()
plt.show()方差代表稳定性,可以看到三个模型的方差大小都差不多,说明他们的稳定性差不多。
而且在实际运行过程中,LGBM运行时间最短,综合他们的表现效果、稳定性、运行时间来看——LGBM优于XGB优于RF。 下面对LGBM进行超参数搜索。
4.4.2 超参数搜索
LGBM也属于决策树的基础算法,所以超参数搜索思路是先对决策树的超参数进行搜索,先对下面四个超参数进行随机搜索。Subsample,num_leaves,max_depth,colsample_bytree。
#利用K折交叉验证搜索最优超参数
from sklearn.model_selection import KFold, StratifiedKFold
from sklearn.model_selection import GridSearchCV,RandomizedSearchCV# Choose best hyperparameters by RandomizedSearchCV
#随机搜索决策树的参数
param_distributions = {'max_depth': range(3, 10), 'subsample':np.linspace(0.5,1,5 ),'num_leaves': [15, 31, 63, 127],
'colsample_bytree': [0.6, 0.7, 0.8, 0.9,1.0]}
# 'min_child_weight':np.linspace(0,0.1,2 ),
kfold = KFold(n_splits=3, shuffle=True, random_state=1)
model =RandomizedSearchCV(estimator= LGBMClassifier(objective='multiclass',num_class=3,random_state=1),
param_distributions=param_distributions, n_iter=200)
model.fit(X_train_s, y_train)搜索200次得到
最优参数
model.best_params_测试集上评估
model = model.best_estimator_
pred=model.predict(X_test_s)
evaluation(y_test,pred)准确率为85.86%。得到的最优参数为'subsample': 0.875, 'num_leaves': 63, 'max_depth': 9, 'colsample_bytree': 0.6。下面再对学习率和基础学习器的的个数进行网格化搜索。
param_grid={'learning_rate': np.linspace(0.05,0.3,6 ), 'n_estimators':[100,200,300,500,1000]}
model =GridSearchCV(estimator= LGBMClassifier(objective='multiclass',num_class=3,random_state=1), param_grid=param_grid, cv=3)
model.fit(X_train_s, y_train)model.best_params_得到的最优参数为learning_rate': 0.1, 'n_estimators': 100。
然后将寻找到的最优参数传入模型,再次进行拟合评价,轻微修改调试。
#利用找出来的最优超参数在所有的训练集上训练,然后预测
model=LGBMClassifier(objective='multiclass',num_class=3,subsample=0.875,learning_rate= 0.1,n_estimators= 190,
num_leaves= 63,max_depth= 9,colsample_bytree=0.6,random_state=0)
model.fit(X_train_s, y_train)
pred=model.predict(X_test_s)
evaluation(y_test,pred)可以看到测试集的拟合优度上升了一点。
上面的模型参数确定为最优的模型参数,使用该模型在全部数据上进行训练,然后对验证集预测,结果保存。
model=LGBMClassifier(objective='multiclass',num_class=3,subsample=0.875,learning_rate= 0.1,n_estimators= 190,
num_leaves= 63,max_depth= 9,colsample_bytree=0.6,random_state=0)
model.fit(np.r_[X_train_s,X_test_s],np.r_[y_train,y_test]) #使用所有数据训练
pred=model.predict(np.r_[X_train_s,X_test_s])
evaluation(np.r_[y_train,y_test],pred)将y的预测出来的0,1,2映射回分子类别的文本形式。
y_dict={}
for i,v in enumerate(uniques):
y_dict[i]=v
y_dict预测储存
pred = model.predict(X2_s)
df=pd.DataFrame(formula)
df['predicted type']=pred
df['predicted type']=df['predicted type'].map(y_dict)
df.to_csv('全部数据预测结果.csv',index=False)这样就预测完成了,储存完毕:
查看预测出来的三种分子类别的数量柱状图:
df['predicted type'].value_counts().plot.bar()和训练集的y差不多,比较合理。
4.5变量重要性
基础模型还可以根据不同特征变量分裂让损失函数下降的程度来对特征变量的重要性进行排序,利用上述最优模型在全部数据上训练后,得到的变量重要性排序如下:
model=LGBMClassifier(objective='multiclass',num_class=3,subsample=0.875,learning_rate= 0.1,n_estimators= 190,
num_leaves= 63,max_depth= 9,colsample_bytree=0.6,random_state=0)
model.fit(np.r_[X_train_s,X_test_s],np.r_[y_train,y_test]) #使用所有数据训练
pred=model.predict(np.r_[X_train_s,X_test_s])
evaluation(np.r_[y_train,y_test],pred)sorted_index = model.feature_importances_.argsort()[::-1]
mfs=model.feature_importances_[sorted_index]
plt.figure(figsize=(4,4),dpi=128)
sns.barplot(y=np.array(range(len(mfs))),x=mfs,orient='h')
plt.yticks(np.arange(X.shape[1]), X.columns[sorted_index])
plt.xlabel('Feature Importance')
plt.ylabel('Feature')
plt.title('LGBM')
plt.show()可以看到对分子种类影响最大的是MZ,NOSC,OC,DBE.O,Almod等变量。
总结与反思
整体实验得到结论如下:
- 在分子中C,H,O这些变量相关性很高,说明他们总是一起构成化学键出现的。
- 不同分子中的N和S的含量差异较大,有一些分子的N和S含量高会和别的分子具有不同差异。
- 在这个分子数据上表现最好的模型是轻量梯度提升集成模型——LGBM,我们最终选择它作为预测模型。
- LGBM超参数搜索得到最优的超参数为:'subsample': 0.875, 'num_leaves': 63, 'max_depth': 9, 'colsample_bytree': 0.6 ,’learning_rate': 0.1, 'n_estimators': 190。这些参数用于最后的预测模型。
- 从LGBM模型来看,对于分子类别影响最大的变量是MZ,NOSC,OC等变量。
本设计研究目的是从分子的特征信息中挖掘出对分子类别预测的影响。17999条样本数据。从数据的读取,预处理,再到数据的探索,可视化描述性统计,相关性分析,异常值处理模型的选择,评价指标的计算,交叉验证,超参数搜索到最终的预测构建,完整的完成了一个机器学习模型构建的项目。并对验证集的3735条数据进行了预测。
实验过程中,我们发现集成模型对于这种表格数据的分类效果最好,并且轻量梯度提升模型运算时间短,这对于做K折交叉验证和超参数搜索是一个不错的优点。虽然最在测试集上的数据精度可能只能维持到86%左右,但是这应该是这个数据特征变量所能发挥的全部性能,模型应该是充分的挖掘了特征变量中的隐藏信息。如果还需要再进一步提高准确率,可能需要从数据本身入手,可以尝试构建更多的特征变量和做更优的特征工程。