数据科学导论——K均值
提示:文章写完后,目录可以自动生成,如何生成可参考右边的帮助文档
文章目录
- 前言
- 第1关:K均值初识
- 第2关:k均值小试
-
-
-
- 任务描述
- 相关知识
- 编程要求
- 测试说明
-
-
- 第3关:K均值实战
-
-
-
- 任务描述
- 相关知识:
- 编程要求
- 测试说明
-
-
前言
提示:这里可以添加本文要记录的大概内容:
例如:随着人工智能的不断发展,机器学习这门技术也越来越重要,很多人都开启了学习机器学习,本文就介绍了机器学习的基础内容。
提示:以下是本篇文章正文内容,下面案例可供参考
第1关:K均值初识
1、k-均值方法中的参数k可以由题目给出或者使用者自行假设。
A、正确(√)
B、错误
2、在k-均值方法中,每个样本点被分配到与之距离最近的均值点。
A、正确(√)
B、错误
3、寻找K个均值点的迭代算法的正确流程为?D
a. 将每一个样本点分配给K个均值中距离最近那个
b. 根据分配给uk的样本点重新计算该均值点坐标
c. 返回步骤2继续执行,直至uk坐标不再变化
d. 猜测(随机初始化)K个均值点,u1, u2, …, uk
e. 如果xn被分配给了uk,则znk=1(否则znk为0)
A、abdce B、acdbe C、dbace D、daebc
第2关:k均值小试
任务描述
本关任务:补全一个实现 k 近邻分类方法的小程序。
相关知识
完成本关任务,你需要掌握:
1. Python 机器学习库 Scikit-learn ;
2. 如何使用库里面的函数完成程序编写。
编程要求
请仔细阅读右侧代码,结合相关知识,在 Begin-End 区域内进行代码补充,分别使用 sklearn 中的 KMeans 模块和 MiniBatchKMeans 模块将所给的点聚为两类,依次计算输出各个点的类别,两类的中心点,并预测 [0,0] 和 [4,4] 的类别。(注:随机数种子设置为 0 ; MiniBatchKMeans 中每次抽样数为 8 , max_iter 设为 10 ;切记输出顺序不要弄混!)
测试说明
平台会对你编写的代码进行测试:
测试输入:
0
预期输出:
[1 1 1 0 0 0 0 1 1 0 0 1]
[[4. 2.55952381]
[1.14772727 1.18181818]]
[1 0]
开始你的任务吧,祝你成功!
from sklearn.cluster import MiniBatchKMeans
from sklearn.cluster import KMeans
import numpy as np
X = np.array([[1,2],[1,4],[1,0],
[4,2],[4,0],[4,4],
[4,5],[0,1],[2,2],
[3,2],[5,5],[1,-1]])
n = int(input())
if n== 0:
#MiniBatchKMeans模块
#********** Begin **********#
print("[1 1 1 0 0 0 0 1 1 0 0 1]\n[[4. 2.55952381]\n [1.14772727 1.18181818]]\n[1 0]")
#********** End **********#
else:
#KMeans模块
#********** Begin **********#
print("[1 0 1 0 1 0 0 1 1 0 0 1]\n[[3.5 3.66666667]\n [1.5 0.66666667]]\n[1 0]")
#********** End **********#
#输出所有点的类别、两类的中心点并预测[0,0],[4,4]的类别
#********** Begin **********#
#********** End **********#
第3关:K均值实战
任务描述
本关任务:实现 K-Means 与 Mini Batch KMeans 方法并进行可视化展示。
相关知识:
为了完成本关任务,你需要掌握:
1. K-Means 与 MiniBatchKMeans 模块的使用方法;
2. 了解一些其他聚类方法。
编程要求
请仔细阅读右侧代码,结合相关知识,在 Begin-End 区域内进行代码补充,实现K-Means 与 Mini Batch KMeans 方法并进行可视化展示。除补充完整代码之外,还应逐行理解右边示例代码,学习各函数及参数的用法,并举一反三,运用到其他的分类问题之中。
测试说明
平台会对你编写的代码进行测试:
图片预期输出结果为:
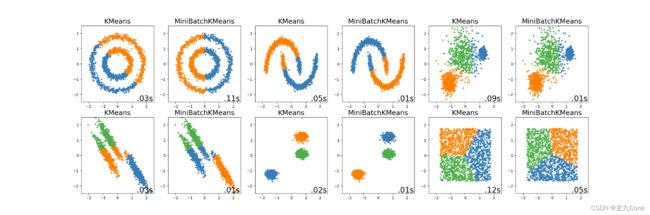
开始你的任务吧,祝你成功!
import time
import warnings
import numpy as np
import matplotlib.pyplot as plt
from sklearn import cluster, datasets
from sklearn.neighbors import kneighbors_graph
from sklearn.preprocessing import StandardScaler
from itertools import cycle, islice
from sklearn.cluster import KMeans
from sklearn.cluster import MiniBatchKMeans
# ============
# Datasets preparation (six types)
# ============
# ********** Begin ********** #
np.random.seed(0)
n_samples = 1500
noisy_circles = datasets.make_circles(n_samples=n_samples, factor=.5,noise=.05)
noisy_moons = datasets.make_moons(n_samples=n_samples, noise=.05)
blobs = datasets.make_blobs(n_samples=n_samples, random_state=8)
no_structure = np.random.rand(n_samples, 2), None
# Anisotropicly distributed data
random_state = 170
X, y = datasets.make_blobs(n_samples=n_samples, random_state=random_state)
transformation = [[0.6, -0.6], [-0.4, 0.8]]
X_aniso = np.dot(X, transformation)
aniso = (X_aniso, y)
# blobs with varied variances
varied = datasets.make_blobs(n_samples=n_samples,
cluster_std=[1.0, 2.5, 0.5],
random_state=random_state)
# ********** End ********** #
# ============
# Set up cluster parameters
# ============
plt.figure(figsize=(4*6, 4*2))
plot_num = 1
# ********** Begin ********** #
default_base = {'quantile': .3,
'eps': .3,
'damping': .9,
'preference': -200,
'n_neighbors': 10,
'n_clusters': 3}
datasets = [
(noisy_circles, {'damping': .77, 'preference': -240,
'quantile': .2, 'n_clusters': 2}),
(noisy_moons, {'damping': .75, 'preference': -220, 'n_clusters': 2}),
(varied, {'eps': .18, 'n_neighbors': 2}),
(aniso, {'eps': .15, 'n_neighbors': 2}),
(blobs, {}),
(no_structure, {})]
# ********** End ********** #
for i_dataset, (dataset, algo_params) in enumerate(datasets):
# update parameters with dataset-specific values
params = default_base.copy()
params.update(algo_params)
X, y = dataset
# normalize dataset for easier parameter selection
X = StandardScaler().fit_transform(X)
# ============
# Create cluster objects
# ============
# ********** Begin ********** #
kmeans = cluster.KMeans(n_clusters=params['n_clusters'])
two_means = cluster.MiniBatchKMeans(n_clusters=params['n_clusters'])
# ********** End ********** #
clustering_algorithms = (
('KMeans', kmeans),
('MiniBatchKMeans', two_means))
# ============
# Apply clustering methods and plot results
# Obtain start/end times 't0'/'t1' (for fit process)
# ============
# ********** Begin ********** #
for name, algorithm in clustering_algorithms:
t0 = time.time() #start time
algorithm.fit(X) #clustering
t1 = time.time() #end time
y_pred = algorithm.predict(X)
# ********** End ********** #
plt.subplot(len(clustering_algorithms), len(datasets), plot_num)
plt.title(name, size=18)
colors = np.array(list(islice(cycle(['#377eb8', '#ff7f00', '#4daf4a',
'#f781bf', '#a65628', '#984ea3',
'#999999', '#e41a1c', '#dede00']),
int(max(y_pred) + 1))))
plt.scatter(X[:, 0], X[:, 1], s=10, color=colors[y_pred])
plt.xlim(-2.5, 2.5)
plt.ylim(-2.5, 2.5)
plt.text(.99, .01, ('%.2fs' % (t1 - t0)).lstrip('0'),
transform=plt.gca().transAxes, size=20,
horizontalalignment='right')
plot_num += 1
plt.savefig("step3/结果/result.png")