已知距离矩阵,R语言画聚类图
已知距离矩阵x,怎么进行系统聚类并画出谱系图
查了网上不少资料,都是随机生成样本点,然后计算距离矩阵,最后再画出聚类图。
但我现在已知距离矩阵,怎么画聚类图呢?(此处采用最短距离(single))
R语言代码:
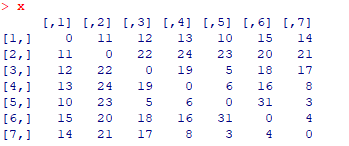
x <- matrix(c(0, 11,12,13,10,15,14,11, 0, 22, 24,23,20,21,12,22,0,19,5,18,17,13,24,19,0,6,16,8,10,23,5,6,0,31,3,15,20,18,16,31,0,4,14,21,17,8,3,4,0),nrow=7,ncol=7)
查看x:
可见已经输入进去原始的距离矩阵。
这个时候直接用*hclust()*进行操作会报错:
需要转换为一般用dist()计算距离矩阵的样式,用如下代码转换:
d<-as.dist(x)
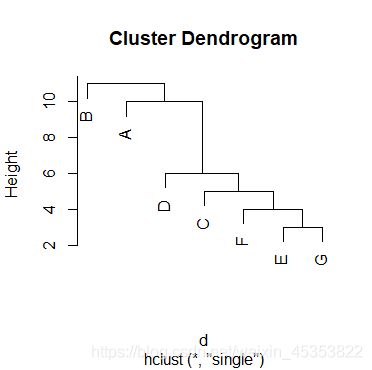
此时在进行聚类图画图就ok了。
hc1=hclust(d,"single");
plot(hc1);
但是,样本一般有名字,本次的样本为“A,B,C,D,E,F,G”七个样本,需要对矩阵行标进行命名:
rownames(x) <- LETTERS[1:7]
此时查看x:
搞定。
完整代码:
x <- matrix(c(0, 11,12,13,10,15,14,11, 0, 22, 24,23,20,21,12,22,0,19,5,18,17,13,24,19,0,6,16,8,10,23,5,6,0,31,3,15,20,18,16,31,0,4,14,21,17,8,3,4,0),nrow=7,ncol=7)
rownames(x) <- LETTERS[1:7]
d<-as.dist(x)
hc1=hclust(d,"single");
plot(hc1);