针对MNE画脑电地形图出现ValueError: DigMontage is only a subset of info问题
脑电地形图的绘制对后面的ICA分析挺有用的,所以想自己学会绘制。
Python中MNE库的脑电地形图绘制我是从这里面了解到可以直接用一条代码即可。
我直接用下面这条代码,就出现了ValueError: DigMontage is only a subset of info. There are 22 channel positions not present in the DigMontage. The required channels are:
这个问题,这个问题说是:ch_names中的你写的各通道名称不是标准名称。
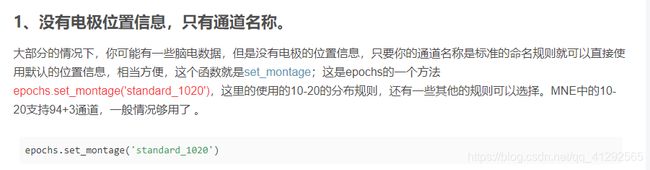
info.set_montage('standard_1020')
不过要用上面这条代码有个小前提,就是需要有你的脑电数据的各电极位置,如:我是从bci_IV_2a中获得数据,这个desc_2a.pdf有各通道的电极位置,如下图就是各通道的电极位置。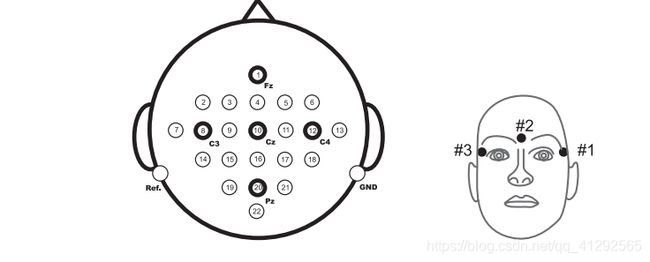
有了各电极的位置,就可以将通道名称按顺序改为标准的通道名称。上图电极位置标准是采用国际10/20系统,关于国际10/20系统的一些介绍可以看下添加链接描述和添加链接描述
解决流程:
1从含有标准电极位置图找到下面这张图

**2:**按着上面标准的电极位置,将自己数据的各通道也改为标准的,如'EEG-Fz', 'EEG-0', 'EEG-1', 'EEG-2', 'EEG-3', 'EEG-4', 'EEG-5', 'EEG-C3', 'EEG-6', 'EEG-Cz', 'EEG-7', 'EEG-C4', 'EEG-8', 'EEG-9', 'EEG-10', 'EEG-11', 'EEG-12', 'EEG-13', 'EEG-14', 'EEG-Pz', 'EEG-15', 'EEG-16'我的数据里默认的通道名称是这样的,
所以要按顺序改为标准的:'Fz', 'FC3', 'FC1', 'FCz', 'FC2', 'FC4', 'C5', 'C3', 'C1', 'Cz', 'C2', 'C4', 'C6', 'CP3', 'CP1','CPz', 'CP2', 'CP4', 'P1', 'Pz', 'P2', 'POz'这就是我按顺序改的
具体的代码是:
原本的代码
ch_names=['EEG-Fz', 'EEG-0', 'EEG-1', 'EEG-2', 'EEG-3', 'EEG-4', 'EEG-5', 'EEG-C3', 'EEG-6', 'EEG-Cz', 'EEG-7', 'EEG-C4', 'EEG-8', 'EEG-9', 'EEG-10', 'EEG-11', 'EEG-12', 'EEG-13', 'EEG-14', 'EEG-Pz', 'EEG-15', 'EEG-16',]
改了后的代码
ch_names=['Fz', 'FC3', 'FC1', 'FCz', 'FC2', 'FC4', 'C5', 'C3', 'C1', 'Cz', 'C2', 'C4', 'C6', 'CP3', 'CP1','CPz', 'CP2', 'CP4', 'P1', 'Pz', 'P2', 'POz']
之后在加上下面这条代码就不会报错,
info.set_montage('standard_1020')
我的数据集是BCI_Competition_IV_dataSet 2,可以从BCI_Competition_IV中获得
整个代码下面也会提供,下面这个代码是从.gdf中获得数据,通道名称等,利用MNE库构造raw,最后显示脑电数据。要利用MNE库绘出脑电地形图,需要将数据构造为raw,epochs等格式。
import mne
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
from mne.preprocessing import (ICA, create_eog_epochs, create_ecg_epochs,corrmap)
# path='F:/python_test/CNN-test/Deep-Learning-BCI-IV-2a-master/1111111111111python_test/Escalgrams_CWT/CWT_CNN/dataset/'
# y_train=pd.read_csv(path+'A01T-labbel.txt',header=None)
filename='E:/软件安装/python1111111111111111/dataset/2008_BCI_Competition_IV/BCI_Competition_IV_dataSet 2/'
raw=mne.io.read_raw_gdf(filename+'A01T.gdf')
#
#['EEG-Fz', 'EEG-0', 'EEG-1', 'EEG-2', 'EEG-3', 'EEG-4', 'EEG-5', 'EEG-C3', 'EEG-6', 'EEG-Cz', 'EEG-7', 'EEG-C4', 'EEG-8', 'EEG-9', 'EEG-10',
# 'EEG-11', 'EEG-12', 'EEG-13', 'EEG-14', 'EEG-Pz', 'EEG-15', 'EEG-16', 'EOG-left', 'EOG-central', 'EOG-right']
#
events1,event1_id=mne.events_from_annotations(raw)
print(events1,'111111',event1_id)
print(raw.info['ch_names'])
raw.drop_channels(['EOG-left', 'EOG-central', 'EOG-right'])
data=raw.get_data()
print(data.shape)
#'Fz', 'FC3', 'FC1', 'FCz', 'FC2', 'FC4', 'C5', 'C3', 'C1', 'Cz', 'C2', 'C6', 'CP3', 'CP1', 'CPz',
#'CP2', 'CP4', 'CP6', 'P1', 'Pz', 'P2', 'POz'
ch_names=['Fz', 'FC3', 'FC1', 'FCz', 'FC2', 'FC4', 'C5', 'C3', 'C1', 'Cz', 'C2', 'C4', 'C6', 'CP3', 'CP1','CPz', 'CP2', 'CP4', 'P1', 'Pz', 'P2', 'POz']
sfreq=raw.info['sfreq']
ch_types=['eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg','eeg']
info=mne.create_info(ch_names=ch_names,ch_types=ch_types,sfreq=sfreq)
info.set_montage('standard_1020')
custom_raw=mne.io.RawArray(data,info)
custom_raw.plot(show_scrollbars=False,show_scalebars=False)
plt.show()
通过解决这些问题我也知道了问题基本上都能自己解决,要耐下心来,好好找资料,不断的尝试,总会解决问题的。