讯飞-糖尿病遗传风险检测挑战赛
讯飞-糖尿病遗传风险检测挑战赛
- 前言
- 相关库
- 一、比赛报名
-
- 1.1 赛事任务
- 1.2 读取数据
-
- 训练数据集
- 测试数据集
- 1.3 数据集基本信息
-
- 数据集维度
- 数据表基本信息
- 二、比赛数据分析
-
- 2.1 缺失值统计
- 2.2 分析字段类型
-
- 每一列数据的类型
- int 和 float 类型数据的描述性统计
- 2.3 字段相关性
- 2.4 小结
- 三、逻辑回归尝试
-
- 3.1 使用名义变量重构糖尿病家族史和性别
- 3.2 处理缺失值
- 3.3 网格搜索查找最佳超参数
- 3.4 初步评分
- 四、特征工程
-
- 4.1 增加年龄特征
- 4.2 增加个体与性别平均值之间的差值字段
前言
本文章是在Coggle 30 Days of ML(22年7月)活动中创作的
截至2022年,中国糖尿病患者近1.3亿。中国糖尿病患病原因受生活方式、老龄化、城市化、家族遗传等多种因素影响。同时,糖尿病患者趋向年轻化。
糖尿病可导致心血管、肾脏、脑血管并发症的发生。因此,准确诊断出患有糖尿病个体具有非常重要的临床意义。糖尿病早期遗传风险预测将有助于预防糖尿病的发生。
根据《中国2型糖尿病防治指南(2017年版)》,糖尿病的诊断标准是具有典型糖尿病症状(烦渴多饮、多尿、多食、不明原因的体重下降)且随机静脉血浆葡萄糖≥11.1mmol/L或空腹静脉血浆葡萄糖≥7.0mmol/L或口服葡萄糖耐量试验(OGTT)负荷后2h血浆葡萄糖≥11.1mmol/L。
在这次比赛中,需要通过训练数据集构建糖尿病遗传风险预测模型,然后预测出测试数据集中个体是否患有糖尿病,和我们一起帮助糖尿病患者解决这“甜蜜的烦恼”。
相关库
- pandas(1.1.5)
- numpy(1.19.5)
- matplotlib(3.3.4)
- seaborn(0.11.2)
- scikit-learn(0.24.2)
%matplotlib inline
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.linear_model import LogisticRegression
from sklearn.model_selection import train_test_split, GridSearchCV
from sklearn.preprocessing import MinMaxScaler
from sklearn.pipeline import make_pipeline
plt.rcParams['font.sans-serif'] = ['FangSong'] # 用来正常显示中文标签
plt.rcParams['axes.unicode_minus'] = False # 用来正常显示负号
一、比赛报名
1.1 赛事任务
以下介绍从官网上复制而来
数据集字段说明:
- 编号:标识个体身份的数字;
- 性别:1 表示男性,0 表示女性;
- 出生年份,出生的年份,XXXX 格式;
- 体重指数:体重除以身高的平方,单位 k g / m 2 kg/m^2 kg/m2;
- 糖尿病家族史,标识糖尿病的遗传特征,记录家族里面患有糖尿病的家属;
- 舒张压:心脏舒张时,动脉血管弹性回缩时,产生的压力成为舒张压,单位 m m H g mmHg mmHg;
- 口服耐糖量测试:比赛数据采用 120 分钟耐糖测试后的血糖值,单位 m m o l / L mmol/L mmol/L;
- 胰岛素释放实验:空腹时定量口服葡萄糖刺激 β \beta β 细胞释放胰岛素,比赛数据采用服糖后 120 分钟的血浆胰岛素水平,单位 pmol/L;
- 肱三头肌皮褶厚度:在右上臂后面肩峰与鹰嘴连线的重点处,夹取与上肢衣袖平行的皮褶,纵向测量,单位 cm;
- 患有糖尿病标识:数据标签,1 表示患有糖尿病,0 表示未患有糖尿病。
1.2 读取数据
训练数据集
df_train = pd.read_csv(filepath_or_buffer='../data/比赛训练集.csv', encoding='gbk', index_col='编号')
df_train.head()
| 性别 | 出生年份 | 体重指数 | 糖尿病家族史 | 舒张压 | 口服耐糖量测试 | 胰岛素释放实验 | 肱三头肌皮褶厚度 | 患有糖尿病标识 | |
|---|---|---|---|---|---|---|---|---|---|
| 编号 | |||||||||
| 1 | 0 | 1996 | 30.1 | 无记录 | 106.0 | 3.818 | 7.89 | 0.0 | 0 |
| 2 | 0 | 1988 | 27.5 | 无记录 | 84.0 | -1.000 | 0.00 | 14.7 | 0 |
| 3 | 1 | 1988 | 36.5 | 无记录 | 85.0 | 7.131 | 0.00 | 40.1 | 1 |
| 4 | 1 | 1992 | 29.5 | 无记录 | 91.0 | 7.041 | 0.00 | 0.0 | 0 |
| 5 | 0 | 1998 | 42.0 | 叔叔或者姑姑有一方患有糖尿病 | NaN | 7.134 | 0.00 | 0.0 | 1 |
测试数据集
df_test = pd.read_csv(filepath_or_buffer='../data/比赛测试集.csv', encoding='gbk', index_col='编号')
df_test.head()
| 性别 | 出生年份 | 体重指数 | 糖尿病家族史 | 舒张压 | 口服耐糖量测试 | 胰岛素释放实验 | 肱三头肌皮褶厚度 | |
|---|---|---|---|---|---|---|---|---|
| 编号 | ||||||||
| 1 | 0 | 1987 | 33.1 | 无记录 | 72.0 | 6.586 | 24.16 | 2.94 |
| 2 | 0 | 1998 | 20.6 | 叔叔或者姑姑有一方患有糖尿病 | 68.0 | 3.861 | 0.00 | 0.00 |
| 3 | 1 | 1979 | 42.1 | 无记录 | 98.0 | 5.713 | 0.00 | 3.53 |
| 4 | 0 | 1999 | 34.6 | 无记录 | 66.0 | 4.684 | 0.00 | 3.14 |
| 5 | 0 | 1997 | 27.7 | 无记录 | 89.0 | 7.948 | 14.65 | 2.65 |
1.3 数据集基本信息
数据集维度
print(df_train.shape)
print(df_test.shape)
(5070, 9)
(1000, 8)
数据表基本信息
print('训练集:')
df_train.info()
训练集:
Int64Index: 5070 entries, 1 to 5070
Data columns (total 9 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 性别 5070 non-null int64
1 出生年份 5070 non-null int64
2 体重指数 5070 non-null float64
3 糖尿病家族史 5070 non-null object
4 舒张压 4823 non-null float64
5 口服耐糖量测试 5070 non-null float64
6 胰岛素释放实验 5070 non-null float64
7 肱三头肌皮褶厚度 5070 non-null float64
8 患有糖尿病标识 5070 non-null int64
dtypes: float64(5), int64(3), object(1)
memory usage: 396.1+ KB
df_train['糖尿病家族史'].unique()
array(['无记录', '叔叔或者姑姑有一方患有糖尿病', '叔叔或姑姑有一方患有糖尿病', '父母有一方患有糖尿病'],
dtype=object)
print('测试集:')
df_test.info()
测试集:
Int64Index: 1000 entries, 1 to 1000
Data columns (total 8 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 性别 1000 non-null int64
1 出生年份 1000 non-null int64
2 体重指数 1000 non-null float64
3 糖尿病家族史 1000 non-null object
4 舒张压 951 non-null float64
5 口服耐糖量测试 1000 non-null float64
6 胰岛素释放实验 1000 non-null float64
7 肱三头肌皮褶厚度 1000 non-null float64
dtypes: float64(5), int64(2), object(1)
memory usage: 70.3+ KB
df_test['糖尿病家族史'].unique()
array(['无记录', '叔叔或者姑姑有一方患有糖尿病', '父母有一方患有糖尿病', '叔叔或姑姑有一方患有糖尿病'],
dtype=object)
二、比赛数据分析
2.1 缺失值统计
分析 1.3 数据集基本信息和下面的统计,发现训练集一共有 5070 条数据,但舒张压字段却只有 4823 个非空数据,其他字段都有 5070 个非空数据;测试集一共有 1000 条数据,舒张压字段只有 951 个非空数据。因此,可以发现无论是训练集还是测试集,只有舒张压字段存在缺失值,其中训练集存在 247 条带缺失值的数据,测试集 存在 49 条带缺失值的数据。
print('训练集每一列缺失值统计个数:')
df_train.isnull().sum()
训练集每一列缺失值统计个数:
性别 0
出生年份 0
体重指数 0
糖尿病家族史 0
舒张压 247
口服耐糖量测试 0
胰岛素释放实验 0
肱三头肌皮褶厚度 0
患有糖尿病标识 0
dtype: int64
print('测试集每一列缺失值统计个数:')
df_test.isnull().sum()
测试集每一列缺失值统计个数:
性别 0
出生年份 0
体重指数 0
糖尿病家族史 0
舒张压 49
口服耐糖量测试 0
胰岛素释放实验 0
肱三头肌皮褶厚度 0
dtype: int64
训练集和测试集各列缺失比例计算:
df_train.isnull().mean(0)
性别 0.000000
出生年份 0.000000
体重指数 0.000000
糖尿病家族史 0.000000
舒张压 0.048718
口服耐糖量测试 0.000000
胰岛素释放实验 0.000000
肱三头肌皮褶厚度 0.000000
患有糖尿病标识 0.000000
dtype: float64
df_test.isnull().mean(0)
性别 0.000
出生年份 0.000
体重指数 0.000
糖尿病家族史 0.000
舒张压 0.049
口服耐糖量测试 0.000
胰岛素释放实验 0.000
肱三头肌皮褶厚度 0.000
dtype: float64
从上面两处结果可以看出训练集和测试集的缺失值分布差异不大,唯一包含缺失值的是舒张压这一列,且缺失值占比不大。
2.2 分析字段类型
每一列数据的类型
训练集数据类型
df_train.dtypes
性别 int64
出生年份 int64
体重指数 float64
糖尿病家族史 object
舒张压 float64
口服耐糖量测试 float64
胰岛素释放实验 float64
肱三头肌皮褶厚度 float64
患有糖尿病标识 int64
dtype: object
测试集数据类型
df_test.dtypes
性别 int64
出生年份 int64
体重指数 float64
糖尿病家族史 object
舒张压 float64
口服耐糖量测试 float64
胰岛素释放实验 float64
肱三头肌皮褶厚度 float64
dtype: object
int 和 float 类型数据的描述性统计
训练集描述性统计
df_train[['性别', '出生年份', '体重指数', '舒张压', '口服耐糖量测试', '胰岛素释放实验', '肱三头肌皮褶厚度']].describe()
| 性别 | 出生年份 | 体重指数 | 舒张压 | 口服耐糖量测试 | 胰岛素释放实验 | 肱三头肌皮褶厚度 | |
|---|---|---|---|---|---|---|---|
| count | 5070.000000 | 5070.000000 | 5070.000000 | 4823.000000 | 5070.000000 | 5070.000000 | 5070.000000 |
| mean | 0.456805 | 1986.869231 | 37.986785 | 89.423595 | 5.612839 | 4.114321 | 6.994371 |
| std | 0.498180 | 8.919737 | 11.447095 | 9.266992 | 2.257649 | 8.726001 | 13.651442 |
| min | 0.000000 | 1943.000000 | 0.000000 | 30.000000 | -1.000000 | 0.000000 | 0.000000 |
| 25% | 0.000000 | 1980.000000 | 28.400000 | 85.000000 | 4.314000 | 0.000000 | 0.000000 |
| 50% | 0.000000 | 1987.000000 | 36.550000 | 89.000000 | 5.760000 | 0.000000 | 0.000000 |
| 75% | 1.000000 | 1995.000000 | 47.600000 | 96.000000 | 7.193000 | 7.100000 | 4.120000 |
| max | 1.000000 | 2009.000000 | 65.900000 | 126.000000 | 10.839000 | 108.960000 | 45.000000 |
测试集描述性统计
df_test[['性别', '出生年份', '体重指数', '舒张压', '口服耐糖量测试', '胰岛素释放实验', '肱三头肌皮褶厚度']].describe()
| 性别 | 出生年份 | 体重指数 | 舒张压 | 口服耐糖量测试 | 胰岛素释放实验 | 肱三头肌皮褶厚度 | |
|---|---|---|---|---|---|---|---|
| count | 1000.000000 | 1000.000000 | 1000.000000 | 951.000000 | 1000.000000 | 1000.000000 | 1000.000000 |
| mean | 0.481000 | 1986.386000 | 39.439000 | 89.638275 | 5.872314 | 4.102700 | 7.064240 |
| std | 0.499889 | 8.816163 | 11.284861 | 9.379124 | 1.930880 | 8.594005 | 13.900938 |
| min | 0.000000 | 1958.000000 | 0.000000 | 28.000000 | -1.000000 | 0.000000 | 0.000000 |
| 25% | 0.000000 | 1979.000000 | 29.975000 | 85.000000 | 4.516000 | 0.000000 | 0.000000 |
| 50% | 0.000000 | 1987.000000 | 38.900000 | 89.000000 | 5.851500 | 0.000000 | 0.000000 |
| 75% | 1.000000 | 1994.000000 | 48.950000 | 96.000000 | 7.465000 | 7.202500 | 3.820000 |
| max | 1.000000 | 2003.000000 | 60.000000 | 112.000000 | 10.613000 | 123.890000 | 44.900000 |
对训练集和测试集进行描述性统计,发现两个数据集在平均值、方差、四分位数、中位数等方面差异不大。
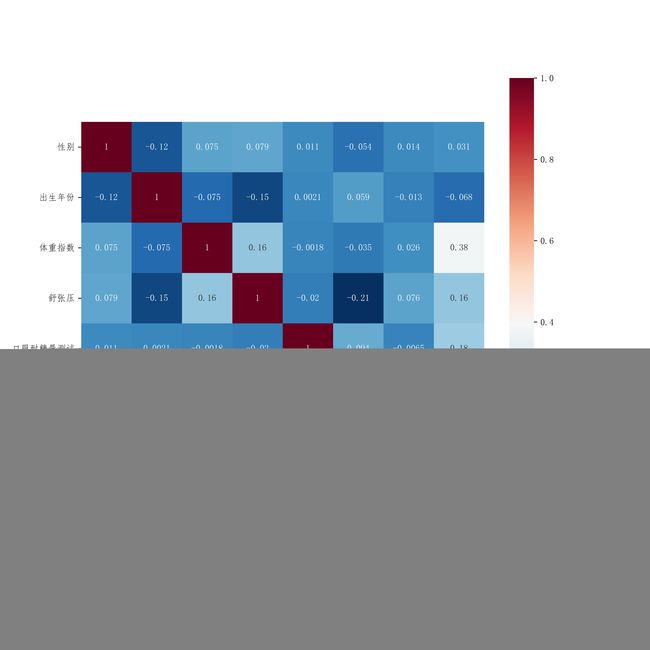
2.3 字段相关性
训练集各字段及标签之间的相关性
# 训练集相关性热力图矩阵
plt.subplots(figsize=(10,10))
sns.heatmap(df_train.corr(method='pearson'), annot=True, vmax=1, square=True, cmap='RdBu_r')
plt.savefig('../images/train_pearson.jpg', dpi=800)
测试集各字段相关性
# 测试集相关性热力图矩阵
plt.subplots(figsize=(10,10))
sns.heatmap(df_test.corr(method='pearson'), annot=True, vmax=1, square=True, cmap='RdBu_r')
plt.savefig('../images/test_pearson.jpg', dpi=800)
从 2.3 中的两个热力图可以看出,训练集中体重指数和肱三头肌皮褶厚度与标签的相关性相对较高,肱三头肌皮褶厚度与标签的相关性最高。各字段之间的相关性普遍不高。
2.4 小结
通过对训练集和测试集计算缺失值比例、分析字段类型、进行描述性统计、计算字段相关性,可以发现训练集和测试集各项指标间的差异很小,甚至几乎没有,实现控制异常数据对模型的干扰。
三、逻辑回归尝试
3.1 使用名义变量重构糖尿病家族史和性别
dict_糖尿病家族史 = {
'无记录': 0,
'叔叔或姑姑有一方患有糖尿病': 1,
'叔叔或者姑姑有一方患有糖尿病': 1,
'父母有一方患有糖尿病': 2
}
df_train['糖尿病家族史'] = df_train['糖尿病家族史'].map(dict_糖尿病家族史)
df_train['糖尿病家族史'] = df_train['糖尿病家族史'].astype('category')
df_test['糖尿病家族史'] = df_test['糖尿病家族史'].map(dict_糖尿病家族史)
df_test['糖尿病家族史'] = df_test['糖尿病家族史'].astype('category')
# 查看重构后的结果
print(df_train['糖尿病家族史'].unique())
print(df_test['糖尿病家族史'].unique())
[0, 1, 2]
Categories (3, int64): [0, 1, 2]
[0, 1, 2]
Categories (3, int64): [0, 1, 2]
df_train['性别'] = df_train['性别'].astype('category')
df_test['性别'] = df_test['性别'].astype('category')
print(df_train['性别'].unique())
print(df_test['性别'].unique())
[0, 1]
Categories (2, int64): [0, 1]
[0, 1]
Categories (2, int64): [0, 1]
3.2 处理缺失值
这里要提一下,我是第一次做讯飞的数据挖掘题,所以对比赛数据格式不是很了解,最开始处理缺失值的时候采用的是
dropna的方法,这样做会将部分编号的数据给删掉,但是这样做会导致评测失败,失败原因:输入格式错误。所以后来换了fillna的方法。
# 用前一个非空白值填补
df_train['舒张压'].fillna(method='ffill', inplace=True)
df_test['舒张压'].fillna(method='ffill', inplace=True)
print('训练集每一列缺失值统计个数:')
print(df_train.isnull().sum())
print('\n')
print('测试集每一列缺失值统计个数:')
print(df_test.isnull().sum())
训练集每一列缺失值统计个数:
性别 0
出生年份 0
体重指数 0
糖尿病家族史 0
舒张压 0
口服耐糖量测试 0
胰岛素释放实验 0
肱三头肌皮褶厚度 0
患有糖尿病标识 0
dtype: int64
测试集每一列缺失值统计个数:
性别 0
出生年份 0
体重指数 0
糖尿病家族史 0
舒张压 0
口服耐糖量测试 0
胰岛素释放实验 0
肱三头肌皮褶厚度 0
dtype: int64
df_train.head()
| 性别 | 出生年份 | 体重指数 | 糖尿病家族史 | 舒张压 | 口服耐糖量测试 | 胰岛素释放实验 | 肱三头肌皮褶厚度 | 患有糖尿病标识 | |
|---|---|---|---|---|---|---|---|---|---|
| 编号 | |||||||||
| 1 | 0 | 1996 | 30.1 | 0 | 106.0 | 3.818 | 7.89 | 0.0 | 0 |
| 2 | 0 | 1988 | 27.5 | 0 | 84.0 | -1.000 | 0.00 | 14.7 | 0 |
| 3 | 1 | 1988 | 36.5 | 0 | 85.0 | 7.131 | 0.00 | 40.1 | 1 |
| 4 | 1 | 1992 | 29.5 | 0 | 91.0 | 7.041 | 0.00 | 0.0 | 0 |
| 5 | 0 | 1998 | 42.0 | 1 | 91.0 | 7.134 | 0.00 | 0.0 | 1 |
df_test.head()
| 性别 | 出生年份 | 体重指数 | 糖尿病家族史 | 舒张压 | 口服耐糖量测试 | 胰岛素释放实验 | 肱三头肌皮褶厚度 | |
|---|---|---|---|---|---|---|---|---|
| 编号 | ||||||||
| 1 | 0 | 1987 | 33.1 | 0 | 72.0 | 6.586 | 24.16 | 2.94 |
| 2 | 0 | 1998 | 20.6 | 1 | 68.0 | 3.861 | 0.00 | 0.00 |
| 3 | 1 | 1979 | 42.1 | 0 | 98.0 | 5.713 | 0.00 | 3.53 |
| 4 | 0 | 1999 | 34.6 | 0 | 66.0 | 4.684 | 0.00 | 3.14 |
| 5 | 0 | 1997 | 27.7 | 0 | 89.0 | 7.948 | 14.65 | 2.65 |
挑选出字段集和标签集
X = df_train.drop(columns='患有糖尿病标识', inplace=False)
y = df_train['患有糖尿病标识']
3.3 网格搜索查找最佳超参数
使用 sklearn 库的 GridSearchCV 实现网格搜索,并在上面训练集 df_train 上划分出的数据集 X_train 和 y_train 上进行 5 折交叉验证,用 X_test 和 y_test 进行检测,查找最佳超参数。
model = make_pipeline(MinMaxScaler(), LogisticRegression(penalty='l2', multi_class='ovr',max_iter=10000))
params = {
'logisticregression__solver': ['liblinear', 'sag', 'saga', 'newton-cg', 'lbfgs'],
'logisticregression__C': np.linspace(0.1, 1, 10)
}
model.get_params().keys()
dict_keys(['memory', 'steps', 'verbose', 'minmaxscaler', 'logisticregression', 'minmaxscaler__clip', 'minmaxscaler__copy', 'minmaxscaler__feature_range', 'logisticregression__C', 'logisticregression__class_weight', 'logisticregression__dual', 'logisticregression__fit_intercept', 'logisticregression__intercept_scaling', 'logisticregression__l1_ratio', 'logisticregression__max_iter', 'logisticregression__multi_class', 'logisticregression__n_jobs', 'logisticregression__penalty', 'logisticregression__random_state', 'logisticregression__solver', 'logisticregression__tol', 'logisticregression__verbose', 'logisticregression__warm_start'])
grid = GridSearchCV(estimator=model, param_grid=params, cv=5, verbose=1)
grid.fit(X=X, y=y)
Fitting 5 folds for each of 50 candidates, totalling 250 fits
GridSearchCV(cv=5,
estimator=Pipeline(steps=[('minmaxscaler', MinMaxScaler()),
('logisticregression',
LogisticRegression(max_iter=10000,
multi_class='ovr'))]),
param_grid={'logisticregression__C': array([0.1, 0.2, 0.3, 0.4, 0.5, 0.6, 0.7, 0.8, 0.9, 1. ]),
'logisticregression__solver': ['liblinear', 'sag',
'saga', 'newton-cg',
'lbfgs']},
verbose=1)
df_test['label'] = grid.predict(df_test)
df_test.head()
| 性别 | 出生年份 | 体重指数 | 糖尿病家族史 | 舒张压 | 口服耐糖量测试 | 胰岛素释放实验 | 肱三头肌皮褶厚度 | label | |
|---|---|---|---|---|---|---|---|---|---|
| 编号 | |||||||||
| 1 | 0 | 1987 | 33.1 | 0 | 72.0 | 6.586 | 24.16 | 2.94 | 0 |
| 2 | 0 | 1998 | 20.6 | 1 | 68.0 | 3.861 | 0.00 | 0.00 | 0 |
| 3 | 1 | 1979 | 42.1 | 0 | 98.0 | 5.713 | 0.00 | 3.53 | 0 |
| 4 | 0 | 1999 | 34.6 | 0 | 66.0 | 4.684 | 0.00 | 3.14 | 0 |
| 5 | 0 | 1997 | 27.7 | 0 | 89.0 | 7.948 | 14.65 | 2.65 | 0 |
df_test.index.name = 'uuid'
df_test['label'].to_csv(path_or_buf='../data/submit.csv', index='uuid')
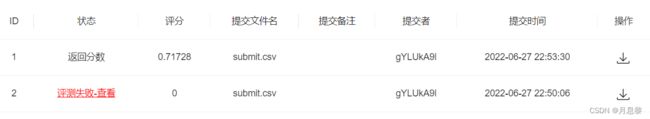
3.4 初步评分
这里的分数只有 0.7,可能是因为训练模型过拟合,也可能是模型原因,后续再加以改进
四、特征工程
4.1 增加年龄特征
df_train['年龄'] = 2022 - df_train['年龄']
df_test['年龄'] = 2022 - df_test['年龄']
4.2 增加个体与性别平均值之间的差值字段
先到这,后面再补充