R语言PCA主成分分析可视化3D版
之前详细介绍了R语言中的主成分分析,以及超级详细的主成分分析可视化方法,主要是基于factoextra和factoMineR两个神包。
-
R语言主成分分析
-
R语言主成分分析可视化(颜值高,很详细)
今天说一下如何提取数据用ggplot2画PCA图,以及三维PCA图。
提取数据
还是使用鸢尾花数据集。
rm(list = ls())
pca.res <- prcomp(iris[,-5], scale. = T, center = T)
pca.res
## Standard deviations (1, .., p=4):
## [1] 1.7083611 0.9560494 0.3830886 0.1439265
##
## Rotation (n x k) = (4 x 4):
## PC1 PC2 PC3 PC4
## Sepal.Length 0.5210659 -0.37741762 0.7195664 0.2612863
## Sepal.Width -0.2693474 -0.92329566 -0.2443818 -0.1235096
## Petal.Length 0.5804131 -0.02449161 -0.1421264 -0.8014492
## Petal.Width 0.5648565 -0.06694199 -0.6342727 0.5235971
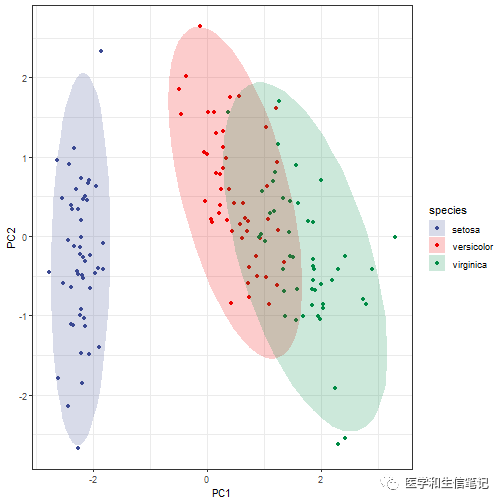
在上一篇中提到过,经典的PCA图的横纵坐标其实就是不同样本在不同主成分中的得分,只要提取出来就可以用ggplot2画了。
# 提取得分
tmp <- as.data.frame(pca.res$x)
head(tmp)
## PC1 PC2 PC3 PC4
## 1 -2.257141 -0.4784238 0.12727962 0.024087508
## 2 -2.074013 0.6718827 0.23382552 0.102662845
## 3 -2.356335 0.3407664 -0.04405390 0.028282305
## 4 -2.291707 0.5953999 -0.09098530 -0.065735340
## 5 -2.381863 -0.6446757 -0.01568565 -0.035802870
## 6 -2.068701 -1.4842053 -0.02687825 0.006586116
和原数据拼到一起就可以画图了:
tmp$species <- iris$Species
head(tmp)
## PC1 PC2 PC3 PC4 species
## 1 -2.257141 -0.4784238 0.12727962 0.024087508 setosa
## 2 -2.074013 0.6718827 0.23382552 0.102662845 setosa
## 3 -2.356335 0.3407664 -0.04405390 0.028282305 setosa
## 4 -2.291707 0.5953999 -0.09098530 -0.065735340 setosa
## 5 -2.381863 -0.6446757 -0.01568565 -0.035802870 setosa
## 6 -2.068701 -1.4842053 -0.02687825 0.006586116 setosa
library(ggplot2)
library(ggsci)
ggplot(tmp, aes(PC1, PC2))+
geom_point(aes(color = species))+
stat_ellipse(aes(fill=species), alpha = 0.2,
geom ="polygon",type = "norm")+
scale_fill_aaas()+
scale_color_aaas()+
theme_bw()
3d版
其实就是使用3个主成分,之前介绍过一种3D版:使用R语言美化PCA图,使用方法非常简单,也是在文献中学习到的。
今天再介绍下scatterplot3d包。
library(scatterplot3d)
scatterplot3d(tmp[,1:3], # 第1-3主成分
# 颜色长度要和样本长度一样,且对应!
color = rep(c("#00AFBB", "#E7B800", "#FC4E07"),each=50),
pch = 15,
lty.hide = 2
)
legend("topleft",c('Setosa','Versicolor','Virginica'),
fill=c("#00AFBB", "#E7B800", "#FC4E07"),box.col=NA)