【论文速览】U-Net 变体模型(3D U-Net,UNet++,V-Net等)
【论文速览】U-Net 变体模型(3D U-Net,UNet++,V-Net等)
U-Net 变体模型
- 【论文速览】U-Net 变体模型(3D U-Net,UNet++,V-Net等)
-
- 【文章一】3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation
-
- abstract
- 摘要
- 感性认识
- 【文章二】UNet++: Redesigning Skip Connections to Exploit Multiscale Features in Image Segmentation
-
- abstract
- 摘要
- 感性认识
- 【文章三】V-Net: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation
-
- abstract
- 摘要
- 感性认识
- 【文章四】3D MRI Brain Tumor Segmentation Using Autoencoder Regularization
-
- abstract
- 摘要
- 感性认识
- 参考资料
【文章一】3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation
- 文章题目:3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation
- 作者:Özgün Çiçek, Ahmed Abdulkadir, Soeren S. Lienkamp, Thomas Brox, Olaf Ronneberger
- 关键词:Convolutional neural networks; 3D ;Biomedical volumetric image segmentation; Xenopus kidney; Semi-automated Fully-automated; Sparse annotation
- 时间:2016
- 来源:MICCAI
- paper:https://link.springer.com/chapter/10.1007/978-3-319-46723-8_49 , https://arxiv.org/abs/1606.06650
- code:
- 引用:Çiçek Ö., Abdulkadir A., Lienkamp S.S., Brox T., Ronneberger O. (2016) 3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation. In: Ourselin S., Joskowicz L., Sabuncu M., Unal G., Wells W. (eds) Medical Image Computing and Computer-Assisted Intervention – MICCAI 2016. MICCAI 2016. Lecture Notes in Computer Science, vol 9901. Springer, Cham. https://doi.org/10.1007/978-3-319-46723-8_49
abstract
This paper introduces a network for volumetric segmentation that learns from sparsely annotated volumetric images. We outline two attractive use cases of this method: (1) In a semi-automated setup, the user annotates some slices in the volume to be segmented. The network learns from these sparse annotations and provides a dense 3D segmentation. (2) In a fully-automated setup, we assume that a representative, sparsely annotated training set exists. Trained on this data set, the network densely segments new volumetric images. The proposed network extends the previous u-net architecture from Ronneberger et al. by replacing all 2D operations with their 3D counterparts. The implementation performs on-the-fly elastic deformations for efficient data augmentation during training. It is trained end-to-end from scratch, i.e., no pre-trained network is required. We test the performance of the proposed method on a complex, highly variable 3D structure, the Xenopus kidney, and achieve good results for both use cases.
摘要
本文介绍了一种从稀疏标注的立体图像中学习的体积分割网络。我们概述了该方法的两个有吸引力的用例:(1)在半自动化设置中,用户在要分割的体积中注释一些切片。该网络从这些稀疏注释中学习,并提供密集的3D分割。(2)在完全自动化的设置中,我们假设存在一个有代表性的、稀疏注释的训练集。在这个数据集上训练,网络密集分割新的体积图像。该网络扩展了Ronneberger等人之前提出的u-net架构,将所有2D操作都替换为3D操作。该实现在训练过程中执行动态弹性变形以增强数据。端到端训练,即不需要预先训练的网络。我们在一个复杂的、高度可变的3D结构——爪蟾肾脏上测试了该方法的性能,并在两个用例中都取得了良好的结果。
感性认识
- 研究的基本问题
3D图像分割 - 现有问题
- 3D数据的完整注释的成本,以2维切片的形式注释会出现很多重复内容
- 数据
three samples of Xenopus kidney embryos at Nieuwkoop-Faber stage 36-37。
Nieuwkoop, P., Faber, J.: Normal Table of Xenopus laevis (Daudin)(Garland, New York) (1994)
不同的结构被给予标记0:“小管内”;1:“小管”;2:“背景”,以及3:“未标记的”。未加标签的切片中的所有体素也获得标签3(“未加标签的”)。 - 主要方法
结构图:

- 3维卷积,池化。
- 将maxpool之前已有的通道数数量增加一倍来避免瓶颈(bottlenecks)
- 加权softmax损失函数
将未标记像素的权重设置为零可以仅从已标记像素学习。降低了频繁出现的背景的权重,增加了内管的权重,以达到小管和背景体素对损失的均衡影响。标签为3(未标记的)的体素不会影响损失计算,即权重为0。 - 数据增强
除了旋转、缩放和灰度值增强外,在数据和ground-truth标签上都应用了平滑的密集变形场(smooth dense deformation field)。为此,我们从网格中标准差为4的正态分布中采样随机向量,每个方向的间距为32个体素,然后应用B样条插值(B-spline interpolation)。 - Semi-Automated Segmentation 半自动分割
用户从每个体积中注释几个切片(XY,YZ,XZ方向)。
-
不足与展望
-
相关工作
1.Unet
2.Szegedy, C., Vanhoucke, V., Ioffe, S., Shlens, J., Wojna, Z.: Rethinking the inception architecture for computer vision. CoRR abs/1512.00567 (2015)
避免瓶颈
3.Tran, D., Bourdev, L.D., Fergus, R., Torresani, L., Paluri, M.: Deep end2end voxel2voxel prediction. CoRR abs/1511.06681 (2015)
Unet到3D的一次推广 -
小尾巴
1.避免瓶颈
2.密集变形场(smooth dense deformation field)
3.B样条插值(B-spline interpolation)
【文章二】UNet++: Redesigning Skip Connections to Exploit Multiscale Features in Image Segmentation
- 文章题目:UNet++: Redesigning Skip Connections to Exploit Multiscale Features in Image Segmentation
- 作者:Zongwei Zhou; Md Mahfuzur Rahman Siddiquee; Nima Tajbakhsh; Jianming Liang
- 关键词:Neuronal structure segmentation, liver segmentation, cell segmentation, nuclei segmentation, brain tumor segmentation, lung nodule segmentation, medical image segmentation, semantic segmentation, instance segmentation, deep supervision, model pruning
- 时间:2019 / 2018
- 来源:IEEE Transactions on Medical Imaging / MICCAI
- paper:https://ieeexplore.ieee.org/document/8932614 , https://arxiv.org/abs/1807.10165 , https://arxiv.org/abs/1912.05074
- code:
- 引用:Z. Zhou, M. M. R. Siddiquee, N. Tajbakhsh and J. Liang, “UNet++: Redesigning Skip Connections to Exploit Multiscale Features in Image Segmentation,” in IEEE Transactions on Medical Imaging, vol. 39, no. 6, pp. 1856-1867, June 2020, doi: 10.1109/TMI.2019.2959609.
abstract
The state-of-the-art models for medical image segmentation are variants of U-Net and fully convolutional networks (FCN). Despite their success, these models have two limitations: (1) their optimal depth is apriori unknown, requiring extensive architecture search or inefficient ensemble of models of varying depths; and (2) their skip connections impose an unnecessarily restrictive fusion scheme, forcing aggregation only at the same-scale feature maps of the encoder and decoder sub-networks. To overcome these two limitations, we propose UNet++, a new neural architecture for semantic and instance segmentation, by (1) alleviating the unknown network depth with an efficient ensemble of U-Nets of varying depths, which partially share an encoder and co-learn simultaneously using deep supervision; (2) redesigning skip connections to aggregate features of varying semantic scales at the decoder sub-networks, leading to a highly flexible feature fusion scheme; and (3) devising a pruning scheme to accelerate the inference speed of UNet++. We have evaluated UNet++ using six different medical image segmentation datasets, covering multiple imaging modalities such as computed tomography (CT), magnetic resonance imaging (MRI), and electron microscopy (EM), and demonstrating that (1) UNet++ consistently outperforms the baseline models for the task of semantic segmentation across different datasets and backbone architectures; (2) UNet++ enhances segmentation quality of varying-size objects-an improvement over the fixed-depth U-Net; (3) Mask RCNN++ (Mask R-CNN with UNet++ design) outperforms the original Mask R-CNN for the task of instance segmentation; and (4) pruned UNet++ models achieve significant speedup while showing only modest performance degradation. Our implementation and pre-trained models are available at https://github.com/MrGiovanni/UNetPlusPlus.
摘要
医学图像分割的最先进模型是U-Net和全卷积网络(FCN)的变种。尽管这些模型取得了成功,但它们有两个局限性:(1)它们的最优深度是先验未知的,需要广泛的架构搜索或对不同深度的模型进行低效的搜索;(2)它们的跳跃连接强加了一种不必要的限制性融合方案,只强制在编码器和解码器子网络的相同尺度特征映射上进行聚合。为了克服这两个限制,我们提出了一种新的用于语义和实例分割的神经结构UNet++,通过(1)利用不同深度的U-Nets的有效集成来缓解未知的网络深度,这些U-Nets部分共享一个编码器,并使用深度监督同时进行共同学习;(2)重新设计跳跃连接,在解码器子网络上聚合不同语义尺度的特征,形成高度灵活的特征融合方案;(3)设计一种剪枝方案来加快UNet++的推理速度。我们使用6种不同的医学图像分割数据集评估了UNet++,包括多种成像方式,如计算机断层扫描(CT)、磁共振成像(MRI)和电子显微镜(EM),并证明(1)UNet++在跨不同数据集和主干架构的语义分割任务中始终优于基线模型;(2)与固定深度U-Net相比,UNet++提高了不同大小目标的分割质量;(3) Mask RCNN+(采用UNet++设计的Mask R-CNN)在实例分割任务上优于原Mask R-CNN;(4)修剪后的UNet++模型实现了显著的加速,但性能下降幅度不大。我们的实现和预先培训的模型可在https://github.com/MrGiovanni/UNetPlusPlus上获得。
感性认识
- 研究的基本问题
二维医学图像分割。改进UNet。 - 现有问题
- 编解码器网络的最佳深度可能因应用程序的不同而不同,这取决于任务难度和可用于训练的标签数据量。
- 在编解码器网络中使用的跳跃连接的设计是不必要的限制,需要融合相同尺度的编码器和解码器特征地图。虽然这是一种自然的设计,但来自解码器和编码器网络的相同比例的特征地图在语义上是不同的,没有可靠的理论可以保证它们是特征融合的最佳匹配。
- 把不同深度的UNet叠加到一起,形成不同层次(上升通道)信息的判断结果。所有这些U-Net部分共享一个编码器。
- 把同一深度的层,用短的SKIP connection连接起来。再用长的skip connection把下降通道的原始语义输送到对应层次上升通道中。这样最深(最终)的上升通道可以接受所有层次的下降通道的特征,或是直接,或是通过其派生出的上升通道的中间结果。
- 附带剪枝的训练策略。
1)集成模式,其中收集所有分割分支的分割结果,然后进行平均;
2)修剪模式,其中分割输出仅从其中一个分割分支中选择,其选择决定了模型修剪的程度和速度增益。选择的分枝深度以前的部分会被保留,最终结果是一个三角形的剪枝。 - 深度监督
向节点X1、X2、X3和X4的输出附加带有C个核的1×1卷积,后跟Sigmoid激活函数,其中C是在给定数据集中观察到的类数。 - 混合损失
然后,我们针对每个语义尺度定义了由像素交叉熵损失和软骰子系数损失组成的混合分割损失。混合损失可以利用这两个损失函数必须提供的优势:平滑梯度和处理类别失衡
L ( Y , P ) = − 1 N ∑ c = 1 C ∑ n = 1 N ( y n , c log p n , c + 2 y n , c p n , c y n , c 2 + p n , c 2 ) \mathcal{L}\left( Y,P \right) =-\frac{1}{N}\sum_{c=1}^{\mathcal{C}}{\sum_{n=1}^N{\left( y_{n,c}\log p_{n,c}+\frac{2y_{n,c}p_{n,c}}{y_{n,c}^{2}+p_{n,c}^{2}} \right)}} L(Y,P)=−N1c=1∑Cn=1∑N(yn,clogpn,c+yn,c2+pn,c22yn,cpn,c)Y,P是目标标签与预测概率标签。
总损耗函数定义为来自每个单独解码器的混合损耗的加权总和: L = ∑ i = 1 d η i ⋅ L ( Y , P i ) \mathcal{L}=\sum_{i=1}^d{\eta _i\cdot \mathcal{L}\left( Y,P^i \right)} L=i=1∑dηi⋅L(Y,Pi)在实验中,对每个损失赋予相同的平衡权重 η i η_i ηi,即 η i ≡ 1 η_i≡1 ηi≡1
- 对比实验,V-Net
- 原始工作,U-Net
- 小尾巴
【文章三】V-Net: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation
- 文章题目:V-Net: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation
- 作者:Fausto Milletari; Nassir Navab; Seyed-Ahmad Ahmadi
- 关键词:
- 时间:2016
- 来源: 2016 Fourth International Conference on 3D Vision (3DV)
- paper:https://ieeexplore.ieee.org/document/7785132
- code:https://github.com/faustomilletari/VNet (caffe)
- 引用:F. Milletari, N. Navab and S. Ahmadi, “V-Net: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation,” 2016 Fourth International Conference on 3D Vision (3DV), Stanford, CA, USA, 2016, pp. 565-571, doi: 10.1109/3DV.2016.79.
abstract
Convolutional Neural Networks (CNNs) have been recently employed to solve problems from both the computer vision and medical image analysis fields. Despite their popularity, most approaches are only able to process 2D images while most medical data used in clinical practice consists of 3D volumes. In this work we propose an approach to 3D image segmentation based on a volumetric, fully convolutional, neural network. Our CNN is trained end-to-end on MRI volumes depicting prostate, and learns to predict segmentation for the whole volume at once. We introduce a novel objective function, that we optimise during training, based on Dice coefficient. In this way we can deal with situations where there is a strong imbalance between the number of foreground and background voxels. To cope with the limited number of annotated volumes available for training, we augment the data applying random non-linear transformations and histogram matching. We show in our experimental evaluation that our approach achieves good performances on challenging test data while requiring only a fraction of the processing time needed by other previous methods.
摘要
卷积神经网络(CNNs)最近被用于解决计算机视觉和医学图像分析领域的问题。尽管它们很受欢迎,但大多数方法只能处理2D图像,而临床实践中使用的大多数医疗数据都是3D体积的。在这项工作中,我们提出了一种基于体积、全卷积神经网络的三维图像分割方法。我们的CNN在描绘前列腺的MRI体积上接受端到端的训练,并学习预测整个体积的分割。我们基用骰子系数引入了一个新的目标函数,在训练过程中进行优化。这样我们就可以处理前景和背景体素数量不平衡的情况。为了应付有限数量的注释体积可用的训练,我们应用随机非线性转换和直方图匹配来增强数据。我们在实验评估中表明,我们的方法在具有挑战性的测试数据上取得了良好的性能,而只需要之前其他方法所需的一小部分处理时间。
感性认识
- 研究的基本问题
3D医学影像分割。 - 现有问题
- 数据
前列腺MRI图片。
trained our method on 50 MRI volumes, and the relative manual ground truth annotation, obtained from the ”PROMISE2012” challenge dataset [10]. - 主要方法
结构:

- 下采样通过卷积完成,2 * 2 * 2的核,2的步长。用卷积操作替换池操作也会导致网络在训练期间占用更小的内存。
- Dice 损失
公式: D = 2 ∑ i N p i g i ∑ i N p i 2 + ∑ i N g i 2 D=\frac{2\sum_i^N{p_ig_i}}{\sum_i^N{p_i^2}+\sum_i^N{g_i^2}} D=∑iNpi2+∑iNgi22∑iNpigi
导数:

使用该公式,我们不需要在前景和背景体素之间建立正确的平衡,例如通过向不同类别的样本分配损失权重。 - 数据增强
在每一次训练迭代中,通过2×2×2的控制点网格和B样条插值得到的密集变形场,将训练图像的随机变形版本输入到网络中。这些扩充是在每次优化迭代之前“在运行中”执行的,以减轻原本过多的存储需求。此外,我们在训练期间改变数据的强度,以模拟来自扫描仪的数据外观的变化。为此,我们使用直方图匹配来使每次迭代中使用的训练体的强度分布与属于该数据集的其他随机选择的扫描的强度分布相适应。
- 直方图匹配
【文章四】3D MRI Brain Tumor Segmentation Using Autoencoder Regularization
- 文章题目:3D MRI Brain Tumor Segmentation Using Autoencoder Regularization
- 作者:Andriy Myronenko
- 关键词:
- 时间:2018
- 来源:BrainLes@MICCAI
- paper:https://link.springer.com/chapter/10.1007/978-3-030-11726-9_28
- code:
- 引用:Myronenko A. (2019) 3D MRI Brain Tumor Segmentation Using Autoencoder Regularization. In: Crimi A., Bakas S., Kuijf H., Keyvan F., Reyes M., van Walsum T. (eds) Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. BrainLes 2018. Lecture Notes in Computer Science, vol 11384. Springer, Cham. https://doi.org/10.1007/978-3-030-11726-9_28
abstract
Automated segmentation of brain tumors from 3D magnetic resonance images (MRIs) is necessary for the diagnosis, monitoring, and treatment planning of the disease. Manual delineation practices require anatomical knowledge, are expensive, time consuming and can be inaccurate due to human error. Here, we describe a semantic segmentation network for tumor subregion segmentation from 3D MRIs based on encoder-decoder architecture. Due to a limited training dataset size, a variational auto-encoder branch is added to reconstruct the input image itself in order to regularize the shared decoder and impose additional constraints on its layers. The current approach won 1st place in the BraTS 2018 challenge.
摘要
从3D磁共振图像(MRI)中自动分割脑肿瘤对于疾病的诊断、监测和治疗计划是必要的。手工勾画需要解剖学知识,费用昂贵,耗时长,并且可能由于人为错误而不准确。在这里,我们描述了一个基于编码器解码器架构的基于3D-MRI的肿瘤区域语义分割网络。由于训练数据集的大小有限,为了使共享解码器正则化并对其层施加附加约束,增加了变分自动编码器分支来重建输入图像本身。目前的方法在2018年Brats挑战赛中获得第一名。
感性认识
- 研究的基本问题
3D-MRI 脑图像分割 - 现有问题
- 数据
BraTS 2018 training dataset (285 cases)
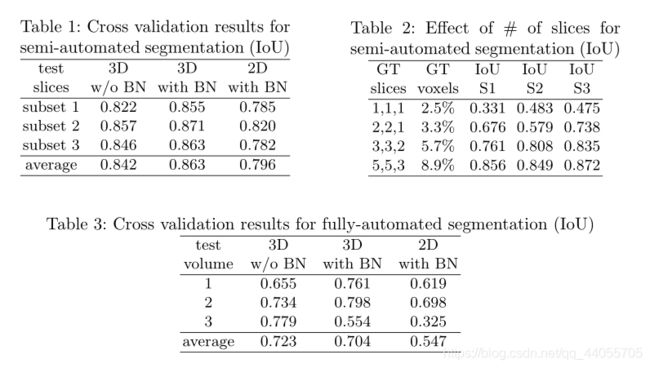
在训练期间,我们使用了大小为160x192x128的随机裁剪,以确保大多数图像内容保留在裁剪区域内。 - 主要方法
结构图:
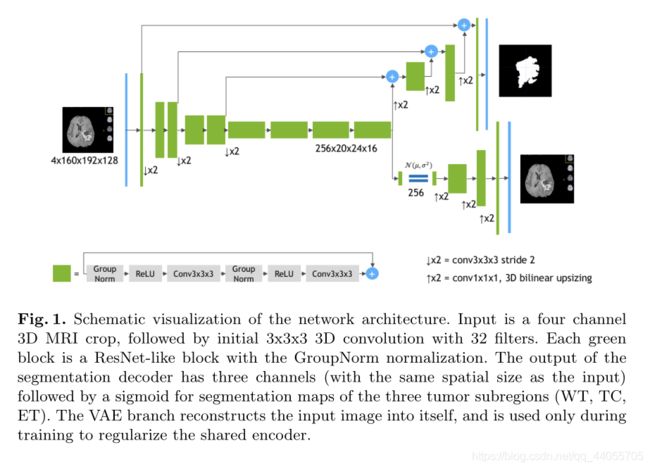
- encoder
编码器部分使用ResNet块,其中每个块由两个具有归一化和RELU的卷积组成,随后是附加的同一性skip连接。对于归一化,我们使用组归一化(Group Normalization)(GN) - decoder
解码器结构类似于编码器结构,但每个空间级别只有一个块。每个解码器级别开始于放大:将特征的数量减少2倍(使用1x1x1卷积),并将空间维度加倍(使用3D双线性上采样),然后添加等效空间级别的编码器输出。 - VAE部分
从编码器端点输出开始,我们首先将输入减少到256的低维空间(128表示平均值,128表示STD)。然后,以给定的均值和标准差从高斯分布中提取样本,并按照与解码器相同的架构将其重构为输入图像的维度,只不过我们在这里不使用编码器的层间跳跃连接。 - 加权损失
L = L D I C E + 0.1 × L L 2 + 0.1 × L K L L=L_{DICE}+0.1\times L_{L2}+0.1\times L_{KL} L=LDICE+0.1×LL2+0.1×LKL
L D I C E L_{DICE} LDICE是V-Net定义的,在分母加一个防爆系数。 L L 2 L_{L2} LL2是VAE的解码的损失: L L 2 = ∥ I i n p u t − I p r e d ∥ 2 2 L_{L2}=\lVert I_{input}-I_{pred} \rVert _{2}^{2} LL2=∥Iinput−Ipred∥22 L K L L_{KL} LKL是VAE的约束损失。 L K L = 1 N ∑ μ 2 + σ 2 − log σ 2 − 1 L_{KL}=\frac{1}{N}\sum{\mu ^2}+\sigma ^2-\log \sigma ^2-1 LKL=N1∑μ2+σ2−logσ2−1 - 数据增强
对输入图像通道应用随机(每通道)强度偏移(图像标准的−0.1…0.1)和比例(0.9…1.1)。我们还应用了概率为0.5的随机轴镜像翻转(针对所有3个轴)
- 组归一化(Group Normalization)(GN)
参考资料
1.https://zhuanlan.zhihu.com/p/44958351
2.http://www.mamicode.com/info-detail-2399938.html
3.https://blog.csdn.net/qq_36484003/article/details/108874913