宏基因组分析-基于Reads比对
- 介绍
宏基因组 ( Metagenome) 指特定环境下所有生物遗传物质的总和。它包含了可培养的和未可培养的微生物的基因。一般从环境样品中提取基因组DNA, 进行高通量测序,从而分析微生物多样性、种群结构、功能信息、与环境之间的关系等。
宏基因组的分析目前主要包括三种方法:基于组装分析、基于reads分析、基于bin分析。
下面我们介绍基于Reads比对的分析方法。
- 分析流程介绍
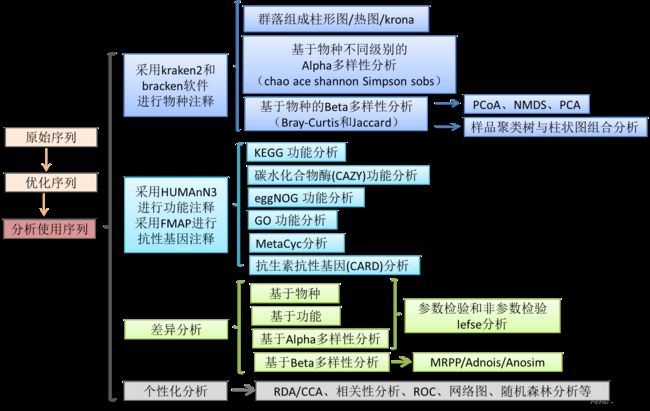
基于Reads的宏基因组分析,使用质控的优化序列与已知物种和功能数据库进行比对,从而得到每个样本的物种、功能注释信息和相对丰度。此方法的优点能够用比较科学的方法鉴定功能的物种来源,既能研究功能组成,也能研究每个功能都是来自哪些物种;分析速度快。
三、详细流程
一、Kranken2物种注释:
Kraken2是一个基于k-mer精确比对算法,并采用LCA (最小共同祖先)的⽅法对序列进行物种注释的软件。Bracken(使用Kraken对丰度的贝叶斯重新估计)是一种高度准确的统计方法,可以从宏基因组学样本中计算DNA序列中物种的丰度。
从质控的优化序列出发,使用Kranken2软件和微生物数据库(包括RefSeq最近的99版本基因组数据,涵盖细菌、真菌、病毒、古菌、原生动物)鉴别样本中所含的物种,再用Bracken对Kraken2得到的分类结果进性分类后贝叶斯重新估算丰度来估算宏基因组样本的种级别相对丰度;
物种风分布柱形图
注:横坐标为样本,纵坐标为各级别的相对丰度;其它级别的图请在下面目录查看。
使用 krona 软件对物种注释结果进行可视化展示,展示结果中,圆圈从内到外依次代表不同的分类级别,扇形的大小代表注释结果的相对比例。
Krone
Alpha 多样性(Alpha diversity)包括:chao指数、ace指数,shannon指数以及simpson指数等。其中,chao指数和ACE指数反映样品中群落的丰富度(speciesrichness),即简单指群落中物种的数量,而不考虑群落中每个物种的丰度情况。而shannon指数以及simpson指数反映群落的多样性(speciesdiversity),受样品群落中物种丰富度(speciesrichness)和物种均匀度(speciesevenness)的影响。相同物种丰富度的情况下,群落中各物种具有越大的均匀度,则认为群落具有越大的多样性。
Alpha多样性指数:采用界、门、纲、目、科、属、种级别的物种丰度进行 Alpha 多样性指数计算。
Alpha index
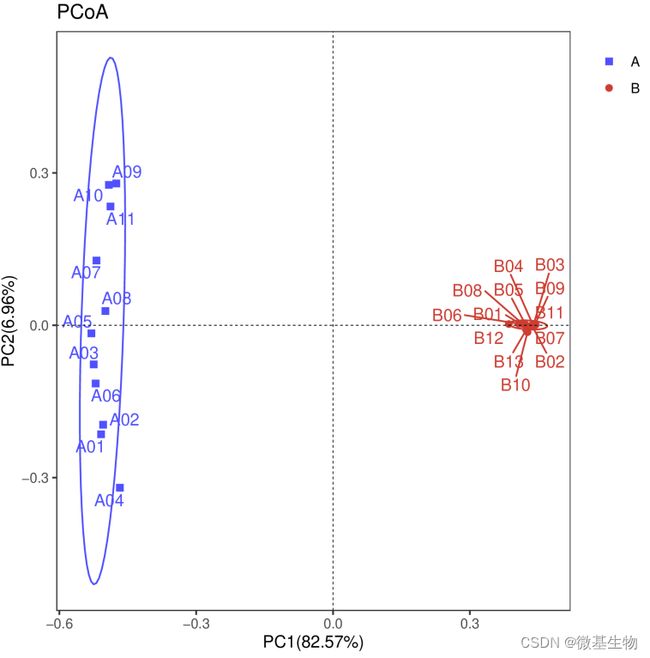
Beta多样性(Beta diversity)分析是用来比较一对样品在物种多样性方面存在的差异大小。分析各类群在样品中的含量,进而计算出不同样品间的Beta多样性值。多种指数可以衡量Beta多样性,常用的为Bray-Curtis,Jaccard。我们将以这两种方法得到的矩阵,做NMDS,PCoA及聚类树分析。
PCoA (Bray_curtis/Jaccard)
二、HUMAnN功能注释:
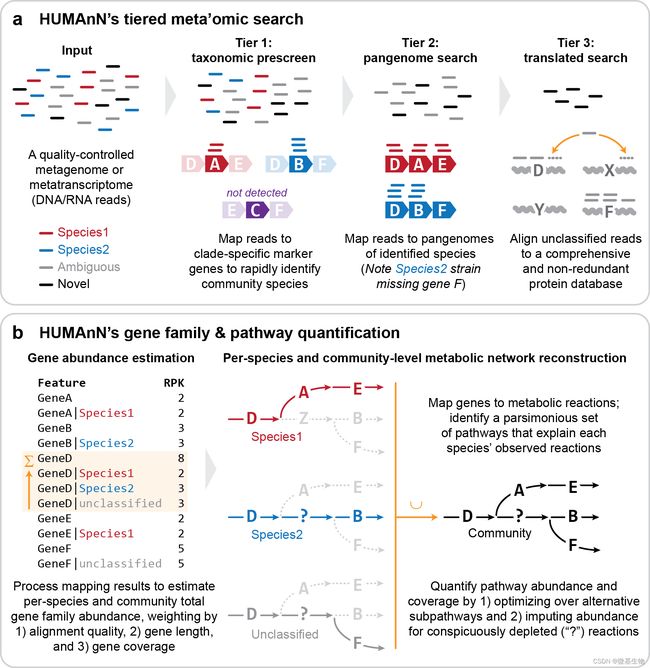
HUMAnN(全称HMP Unified Metabolic Analysis Network)最早由Huttenhower实验室针对HMP项目开发的一款可对宏基因组测序数据进行物种分类和功能分析的流程,它适用于任何类型的微生物群落,而不仅仅是人类微生物组,是目前前沿的权威的宏基因组分析软件。在2020年已更新到第三版称为HUMAnN3。
从质控的优化序列出发,使用HUMAnN3软件和蛋⽩质数据库(UniRef90)进⾏⽐对,根据UniRef90 的ID 和各个功能数据库ID的对应关系,统计各个功能数据库对应功能相对丰度;
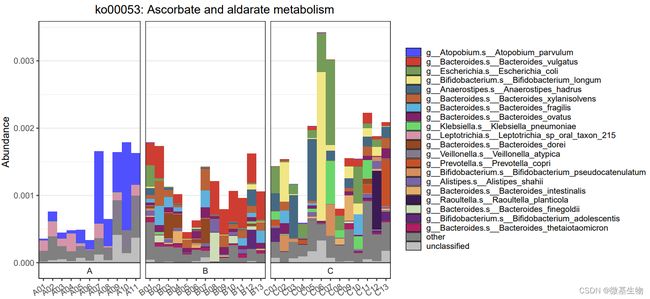
根据 HUMAnN3 的分析结果,得到功能的物种来源,绘制功能物种来源组成Stratified柱形图。我们从图中得到功能的丰度, 和功能来源于哪些物种。
Patway Stratified Barplot
给 pathway(通路)图上色,在通路图中标注出检测到的基因,有分组的再图中 展示出对应的分组信息(用不同颜色的框标注出来)。
三、FMAP功能注释:
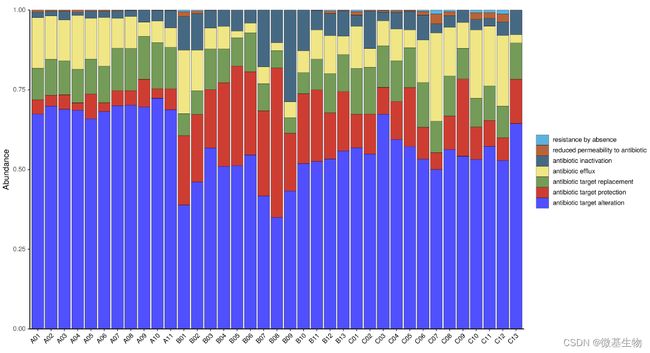
使⽤FMAP软件将各样本质控好的优化序列与CARD参考数据库进⾏⽐对(基于DIAMOND),根据⽐对结果,统计出每个样本⽐对到各ARO参考序列的reads数,从⽽计算相对丰度;通过数据库对应关系,分别计算 AMR_Gene_Family、Drug_Class、Resistance_Mechanism 分类的丰度。
Resistance Mechanism Barplot
四、差异分析
如果老师提供了分组信息,我们可以从物种、功能、alpha、beta多样性不同的角度进行差异分析。包括参数检验、非参数检验、lefse分析等。
五、 相关性分析
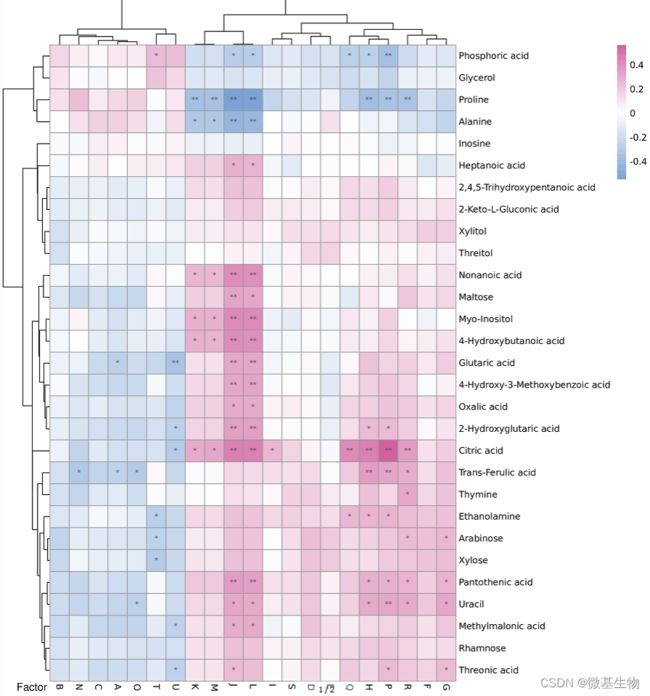
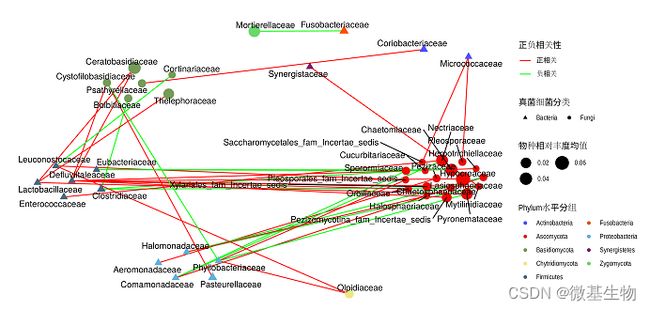
功能和物种之间的相关性、物种和临床指标之间的相关性,只要是同样的样本检测了两种不同的指标,都可以进行两个指标之间的关联分析。展示方式主要是spearman热图和网络图的形式。
相关性热图
图中
(1)颜色代表相关性系数:蓝色为负相关,且颜色越深,相关性系数越大,粉红色为正相关,且颜色越深,相关性系数越大,具体的颜色与相关性系数的对应关系见图中右上角的图注。
(2)横轴代表的是临床因子,纵轴代表的是代谢物。
(3)图中的*代表P值,*为0.05>P>0.01,**为0.01>P>0.001…..,只要是图中标注了*的,都是有显著相关的。
(4)左侧和上面的树都是根据相关性系数的相似性情况进行聚类的。
图中:
(1)图中形状代表不同的检测类型:本图是细菌和真菌,也可以是物种和不同功能之间的相关性。
(2)spearman的结果中p小于0.05,r绝对值大于0.6.
(3)节点的大小代表相对丰度的均值或是相关性程度。
(4)图中节点的颜色代表不同的门。
注意:网络图中展示的信息需要根据老师的要求来设计。