PMA的应用
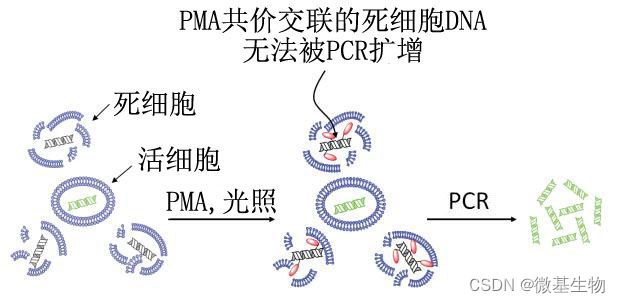
基于核酸的分子生物学检测方法,如聚合酶链式反应(Polymerase Chain Reaction,PCR),能够用于精准地分析样本中微生物的种类和丰度,但是难以区分微生物的存活状态。传统的用于检测样本中活细胞的方法都需要对样本进行培养,但是此类检测方法存在一定的局限性,无法用于检测一些难培养或是不可培养的活细胞。叠氮溴化丙锭(Propidium monoazide,PMA)是一种能与DNA结合的光反应染料,不具有细胞膜渗透性,无法穿透活细胞完整的细胞膜,只能选择性地穿透死细胞受损的细胞膜。进入细胞后,PMA会与DNA双螺旋发生共价交联,进而阻碍死细胞中目标DNA的PCR扩增,如图1所示。基于PMA的这一特性,通过对PMA处理过的样本进行PCR扩增和测序分析,即可得知样本中微生物的存活状态。
图1.PMA的作用原理示意图
现在已经有越来越多的文献报道采用了PMA-PCR或PMA-qPCR等方法对样本中的活细胞进行定性或定量分析,下面解读三篇文献。
文献一:Andreas N, Katherine ES, Anne KC. Molecular monitoring of disinfection efficacy using propidium monoazide in combination with quantitative PCR. Journal of Microbiological Methods, 2007, Volume 70, Issue 2.
文章采用了PMA-qPCR的方法,对四种常用的灭菌方法进行了效率评估。作者选择了鼠伤寒沙门氏杆菌、单核细胞增生李斯特菌、大肠杆菌和鸟分枝杆菌这四种常见的致病菌,分别采用次氯酸盐、苯扎氯铵、紫外线和加热消毒法进行灭菌处理,并用PMA进行交联处理。将处理好的样本和未经处理的对照样本一起进行基因组抽提,并分别用四株菌的特异引物对样本进行qPCR定量分析。作者还同时对样本进行了平板培养,作为PMA处理效果的对照实验。
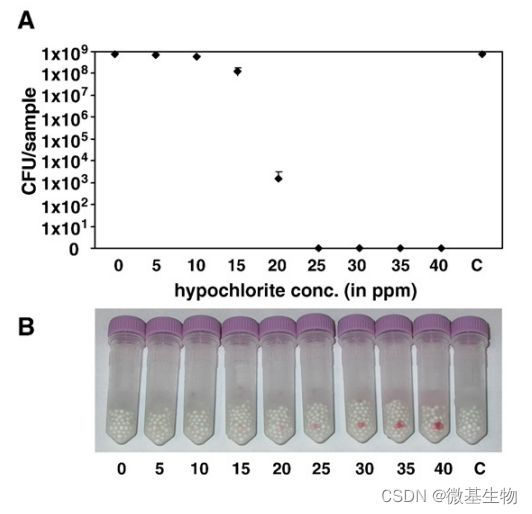
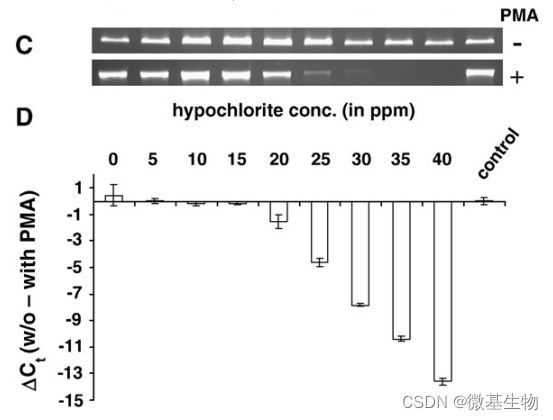
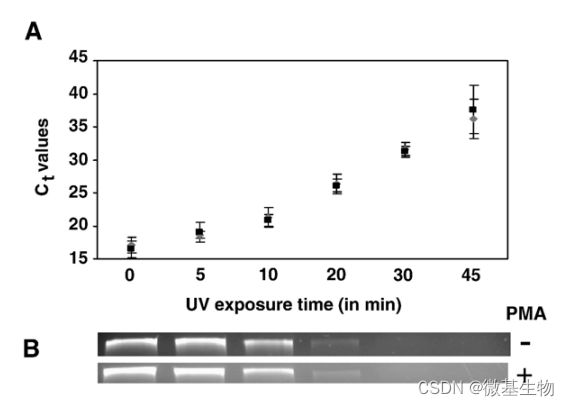
根据实验结果,次氯酸盐、苯扎氯铵和加热消毒法的灭菌效果都符合预期,PMA-qPCR和平板培养的结果基本一致,部分结果如下图1所示。而紫外线处理的样本经过PMA处理后,qPCR定量结果与未处理对照组的基本一致,如下图2所示,且PMA-qPCR的结果与平板培养的结果之间存在较大的差异,其原因可能是紫外线的灭菌原理是破坏微生物的DNA而非细胞膜,这导致了PMA无法与死细胞的DNA进行共价交联,只有用紫外线进行长时间的照射处理才能破坏细胞膜。
图1不同浓度的次氯酸盐对沙门氏菌的灭菌效果。(A)平板计数的结果,横坐标表示次氯酸盐浓度,C为未处理对照。(B)PMA处理后将菌体转移至裂解抽提管中。(C)PMA处理(+)和未处理对照组(-)基因组的凝胶电泳图。(D)qPCR定量结果,纵坐标为未处理对照组和PMA处理组的Ct值差。
图2.不同时长的紫外线处理对大肠杆菌的灭菌效果。(A)PMA处理和未处理对照组的qPCR定量结果,横坐标代表紫外线处理时长,纵坐标为Ct值。(B)PMA处理(+)和未处理对照组(-)基因组的凝胶电泳图。
文献二:Jialing N, Shingo H, Yin W, et al. Uncovering Viable Microbiome in Anaerobic Sludge Digesters by Propidium Monoazide (PMA)-PCR. Microbial Ecology, 2019, Volume 79.
厌氧消化是一种非常高效的污水处理方法,若想维持厌氧消化池的正常运作,就必须首先了解参与消化的微生物种群。本文的作者从多个污水厂采集了污泥样本,分别做了PMA处理和不处理的对照,并进行基因组抽提、16S V3-V4高通量测序和qPCR定量分析。
根据qPCR定量的结果,发现用PMA处理过的样本中16S拷贝数少于未处理过的样本,说明在样本中约有10%的死细胞。对高通量结果进行分析,发现用PMA处理过的样本中微生物的种类和多样性要明显少于未处理过的对照样本,如下图1所示,部分细菌在经过PMA处理后相对丰度降低了约90%,如α-变形菌和β-变形菌,这些微生物可以被视为污水处理过程中的残留物;而有些细菌的相对丰度则升高了,如拟杆菌门、绿弯菌门和厚壁菌门的微生物,具体可见下图2。在所有用PMA处理过的样本中,广古菌门、拟杆菌门、δ-变形菌门、绿弯菌门、厚壁菌门、WWE1、螺旋体门、互养菌门和Caldiserica门的微生物占了大多数。
图1.样品中的OUT数量、Chao 1指数和香农指数。AKT、NGT、NIT、SEN和TNN分别为5个不同的取样地点。DS为厌氧消化池中的淤泥样本,DS-PMA为用PMA处理过的厌氧消化池淤泥样本。
图2.PMA处理后样本中部分微生物门水平相对丰度的变化。黑字表示相对丰度增加,红字表示相对丰度减少。
文献三:Lanxin M, Jie Y, Hao J, et al. Investigating the bacterial microbiota of traditional fermented dairy products using propidium monoazide with single-molecule real-time sequencing. Journal of Dairy Science, 2019, Volume 102, Issue 5.
乳制品在游牧民族中是一类颇受欢迎的食品,而发酵类乳制品中的微生物,尤其是乳酸菌,能够提高乳制品的外观和口感。过去对于乳制品中微生物的鉴定往往采用培养法,但是此方法不仅费时费力,对于一些苛养菌(fastidious microbes)的检测效果也不是很理想。本文的作者对多种传统发酵乳制品进行了采样,使用PMA与三代测序结合的方法,对样本中的活菌和死菌分别进行了分析。作者同时还采用了传统的培养法对样本中的微生物进行分离和鉴定。
分析测序结果,发现乳酸杆菌的相对丰度在所有样本中都是最高的。部分样本在经过PMA处理后,乳酸杆菌的相对丰度都有所提高,链球菌的相对丰度则有所下降;另外一些样本中则是肠杆菌的相对丰度提高了,而乳酸杆菌的相对丰度发生了下降,具体可参考下图1。对比PMA处理的结果和传统培养法的结果,作者发现使用PMA处理的方法可以检测到一些使用培养法检测不到的痕量微生物,如清酒乳杆菌和肠膜明串珠菌。
图1.传统发酵乳制品中微生物分布的柱状图,上图为属水平分布图,下图为种水平分布图。KM、KIM和CM分别代表3种不同的乳制品样本,PKM、PKIM和PCM为相对的PMA处理样本。
微基生物现在也开发出了一套对样本进行PMA处理和高通量/qPCR定量分析的系统,能够对样本中的活菌进行较为精准地定性和定量分析。