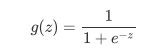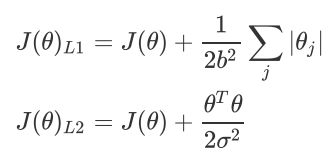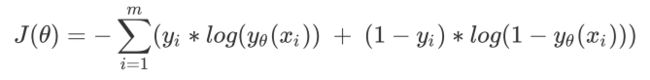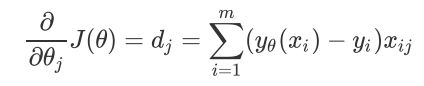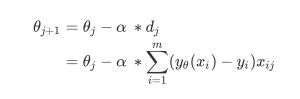sklearn专题五:逻辑回归
目录
1 概述
1.1 名为“回归”的分类器
1.2 为什么需要逻辑回归
2 linear_model.LogisticRegression
2.1 二元逻辑回归的损失函数
2.1.1 损失函数的概念与解惑
2.2 重要参数penalty & C
2.2.1 正则化
2.2.2 逻辑回归中的特征工程
2.3 梯度下降:重要参数max_iter
2.3.1 梯度下降求解逻辑回归
2.3.2 梯度下降的概念与解惑
2.4 二元回归与多元回归:重要参数solver & multi_class
2.5 样本不平衡与参数class_weight
1 概述
1.1 名为“回归”的分类器
面试高危问题:Sigmoid函数的公式和性质
1.2 为什么需要逻辑回归
线性回归对数据的要求很严格,比如标签必须满足正态分布,特征之间的多重共线性需要消除等等,而现实中很多真实情景的数据无法满足这些要求,因此线性回归在很多现实情境的应用效果有限。逻辑回归是由线性回归变化而来,因此它对数据也有一些要求,而我们之前已经学过了强大的分类模型决策树和随机森林,它们的分类效力很强,并且不需要对数据做任何预处理。
- 1. 逻辑回归对线性关系的拟合效果好到丧心病狂,特征与标签之间的线性关系极强的数据,比如金融领域中的信用卡欺诈,评分卡制作,电商中的营销预测等等相关的数据,都是逻辑回归的强项。虽然现在有了梯度提升树GDBT,比逻辑回归效果更好,也被许多数据咨询公司启用,但逻辑回归在金融领域,尤其是银行业中的统治地位依然不可动摇(相对的,逻辑回归在非线性数据的效果很多时候比瞎猜还不如,所以如果你已经知道数据之间的联系是非线性的,千万不要迷信逻辑回归)
- 2. 逻辑回归计算快:对于线性数据,逻辑回归的拟合和计算都非常快,计算效率优于SVM和随机森林,亲测表示在大型数据上尤其能够看得出区别
- 3. 逻辑回归返回的分类结果不是固定的0,1,而是以小数形式呈现的类概率数字:我们因此可以把逻辑回归返回的结果当成连续型数据来利用。比如在评分卡制作时,我们不仅需要判断客户是否会违约,还需要给出确定的”信用分“,而这个信用分的计算就需要使用类概率计算出的对数几率,而决策树和随机森林这样的分类器,可以产出分类结果,却无法帮助我们计算分数(当然,在sklearn中,决策树也可以产生概率,使用接口predict_proba调用就好,但一般来说,正常的决策树没有这个功能)。

2 linear_model.LogisticRegression
2.1 二元逻辑回归的损失函数
2.1.1 损失函数的概念与解惑
2.2 重要参数penalty & C
2.2.1 正则化
from sklearn.linear_model import LogisticRegression as LR
from sklearn.datasets import load_breast_cancer
import numpy as np
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
data = load_breast_cancer()
X = data.data
y = data.target
data.data.shape
lrl1 = LR(penalty="l1",solver="liblinear",C=0.5,max_iter=1000)
lrl2 = LR(penalty="l2",solver="liblinear",C=0.5,max_iter=1000) #逻辑回归的重要属性coef_,查看每个特征所对应的参数
lrl1 = lrl1.fit(X,y)
lrl1.coef_
(lrl1.coef_ != 0).sum(axis=1)
lrl2 = lrl2.fit(X,y)
lrl2.coef_l1 = []
l2 = []
l1test = []
l2test = []
Xtrain, Xtest, Ytrain, Ytest = train_test_split(X,y,test_size=0.3,random_state=420)
for i in np.linspace(0.05,1,19):
lrl1 = LR(penalty="l1",solver="liblinear",C=i,max_iter=1000)
lrl2 = LR(penalty="l2",solver="liblinear",C=i,max_iter=1000)
lrl1 = lrl1.fit(Xtrain,Ytrain)
l1.append(accuracy_score(lrl1.predict(Xtrain),Ytrain))
l1test.append(accuracy_score(lrl1.predict(Xtest),Ytest))
lrl2 = lrl2.fit(Xtrain,Ytrain)
l2.append(accuracy_score(lrl2.predict(Xtrain),Ytrain))
l2test.append(accuracy_score(lrl2.predict(Xtest),Ytest))
graph = [l1,l2,l1test,l2test]
color = ["green","black","lightgreen","gray"]
label = ["L1","L2","L1test","L2test"]
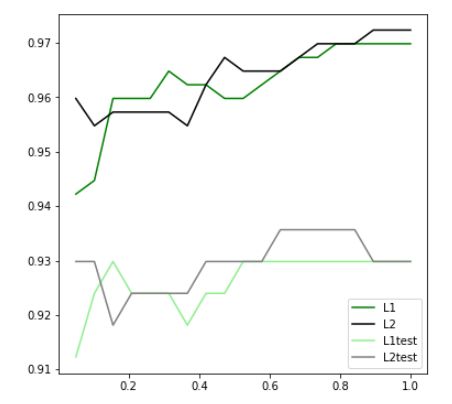
plt.figure(figsize=(6,6))
for i in range(len(graph)):
plt.plot(np.linspace(0.05,1,19),graph[i],color[i],label=label[i])
plt.legend(loc=4) #图例的位置在哪里?4表示,右下角
plt.show()
2.2.2 逻辑回归中的特征工程
- 业务选择
- PCA和SVD一般不用
- 统计方法可以使用,但不是非常必要
- 高效的嵌入法embedded
from sklearn.linear_model import LogisticRegression as LR
from sklearn.datasets import load_breast_cancer
import numpy as np
import matplotlib.pyplot as plt
from sklearn.model_selection import cross_val_score
from sklearn.feature_selection import SelectFromModel
data = load_breast_cancer()
data.data.shape
'''
(569, 30)
'''
LR_ = LR(solver="liblinear",C=0.8,random_state=420)
cross_val_score(LR_,data.data,data.target,cv=10).mean()
'''
0.9508145363408522
'''X_embedded = SelectFromModel(LR_,norm_order=1).fit_transform(data.data,data.target)
X_embedded.shape
'''
(569, 9)
'''cross_val_score(LR_,X_embedded,data.target,cv=10).mean()
'''
0.9368107769423559
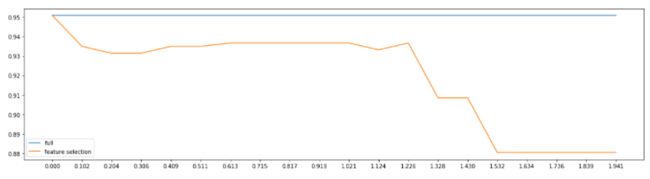
'''看看结果,特征数量被减小到个位数,并且模型的效果却没有下降太多,如果我们要求不高,在这里其实就可以停下了。但是,能否让模型的拟合效果更好呢?在这里,我们有两种调整方式:
fullx = []
fsx = []
threshold = np.linspace(0,abs((LR_.fit(data.data,data.target).coef_)).max(),20)
k=0
for i in threshold:
X_embedded = SelectFromModel(LR_,threshold=i).fit_transform(data.data,data.target)
fullx.append(cross_val_score(LR_,data.data,data.target,cv=5).mean())
fsx.append(cross_val_score(LR_,X_embedded,data.target,cv=5).mean())
print((threshold[k],X_embedded.shape[1]))
k+=1
plt.figure(figsize=(20,5))
plt.plot(threshold,fullx,label="full")
plt.plot(threshold,fsx,label="feature selection")
plt.xticks(threshold)
plt.legend()
plt.show()(0.0, 30)
(0.1021431183124225, 17)
(0.204286236624845, 12)
(0.3064293549372675, 10)
(0.40857247324969, 8)
(0.5107155915621124, 8)
(0.612858709874535, 5)
(0.7150018281869575, 5)
(0.81714494649938, 5)
(0.9192880648118025, 5)
(1.0214311831242249, 5)
(1.1235743014366475, 4)
(1.22571741974907, 3)
(1.3278605380614925, 2)
(1.430003656373915, 2)
(1.5321467746863375, 1)
(1.63428989299876, 1)
(1.7364330113111823, 1)
(1.838576129623605, 1)
(1.9407192479360273, 1)fullx = []
fsx = []
C=np.arange(0.01,10.01,0.5)
for i in C:
LR_ = LR(solver="liblinear",C=i,random_state=420)
fullx.append(cross_val_score(LR_,data.data,data.target,cv=10).mean())
X_embedded = SelectFromModel(LR_,norm_order=1).fit_transform(data.data,data.target)
fsx.append(cross_val_score(LR_,X_embedded,data.target,cv=10).mean())
print(max(fsx),C[fsx.index(max(fsx))])
plt.figure(figsize=(20,5))
plt.plot(C,fullx,label="full")
plt.plot(C,fsx,label="feature selection")
plt.xticks(C)
plt.legend()
plt.show()![]() 继续细化学习曲线:
继续细化学习曲线:
fullx = []
fsx = []
C=np.arange(6.05,7.05,0.005)
for i in C:
LR_ = LR(solver="liblinear",C=i,random_state=420)
fullx.append(cross_val_score(LR_,data.data,data.target,cv=10).mean())
X_embedded = SelectFromModel(LR_,norm_order=1).fit_transform(data.data,data.target)
fsx.append(cross_val_score(LR_,X_embedded,data.target,cv=10).mean())
print(max(fsx),C[fsx.index(max(fsx))])
plt.figure(figsize=(20,5))
plt.plot(C,fullx,label="full")
plt.plot(C,fsx,label="feature selection")
plt.xticks(C)
plt.legend()
plt.show()
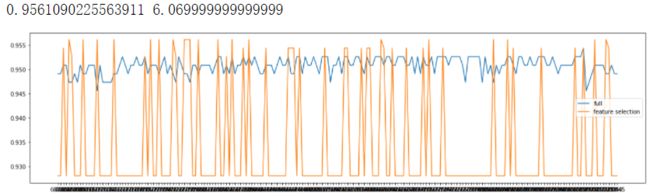
#验证模型效果:降维之前
LR_ = LR(solver="liblinear",C=6.069999999999999,random_state=420)
cross_val_score(LR_,data.data,data.target,cv=10).mean()
#验证模型效果:降维之后
LR_ = LR(solver="liblinear",C=6.069999999999999,random_state=420)
X_embedded = SelectFromModel(LR_,norm_order=1).fit_transform(data.data,data.target)
cross_val_score(LR_,X_embedded,data.target,cv=10).mean()
X_embedded.shape #(569, 11)这样我们就实现了在特征选择的前提下,保持模型拟合的高效,现在,如果有一位医生可以来为我们指点迷津,看看剩下的这些特征中,有哪些是对针对病情来说特别重要的,也许我们还可以继续降维。当然,除了嵌入法,系数累加法或者包装法也是可以使用的。
- 比较麻烦的系数累加法
系数累加法的原理非常简单。在PCA中,我们通过绘制累积可解释方差贡献率曲线来选择超参数,在逻辑回归中我们可以使用系数coef_来这样做,并且我们选择特征个数的逻辑也是类似的:找出曲线由锐利变平滑的转折点,转折点之前被累加的特征都是我们需要的,转折点之后的我们都不需要。不过这种方法相对比较麻烦,因为我们要先对特征系数进行从大到小的排序,还要确保我们知道排序后的每个系数对应的原始特征的位置,才能够正确找出那些重要的特征。如果要使用这样的方法,不如直接使用嵌入法来得方便。
- 简单快速的包装法
2.3 梯度下降:重要参数max_iter
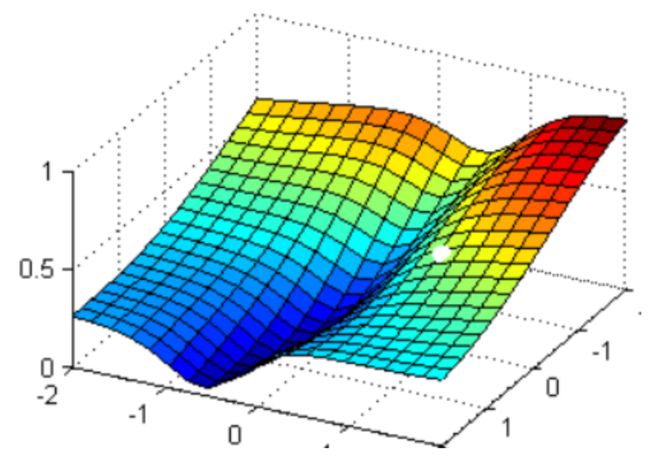
2.3.1 梯度下降求解逻辑回归
但无论如何,小球停下的就是我们在现有状况下可以获得的唯一点了。如果我们够幸运,这个点就是图像的最低点,那我们只要找到这个点的对应坐标(![]() ),就可以获取能够让损失函数最小的参数取值
),就可以获取能够让损失函数最小的参数取值![]() 了。如此,梯度下降的过程就已经完成。
了。如此,梯度下降的过程就已经完成。
的取值就会减少得最快,也就最容易找到损失函数的最小值。
我们对这个函数上的自变量![]() 求偏导,就可以得到梯度向量在第 组 的坐标点上的表示形式:
求偏导,就可以得到梯度向量在第 组 的坐标点上的表示形式:
来看看乳腺癌数据集下,max_iter的学习曲线:
l2 = []
l2test = []
Xtrain, Xtest, Ytrain, Ytest = train_test_split(X,y,test_size=0.3,random_state=420)
for i in np.arange(1,201,10):
lrl2 = LR(penalty="l2",solver="liblinear",C=0.9,max_iter=i)
lrl2 = lrl2.fit(Xtrain,Ytrain)
l2.append(accuracy_score(lrl2.predict(Xtrain),Ytrain))
l2test.append(accuracy_score(lrl2.predict(Xtest),Ytest))
graph = [l2,l2test]
color = ["black","gray"]
label = ["L2","L2test"]
plt.figure(figsize=(20,5))
for i in range(len(graph)):
plt.plot(np.arange(1,201,10),graph[i],color[i],label=label[i])
plt.legend(loc=4)
plt.xticks(np.arange(1,201,10))
plt.show()
#我们可以使用属性.n_iter_来调用本次求解中真正实现的迭代次数
lr = LR(penalty="l2",solver="liblinear",C=0.9,max_iter=300).fit(Xtrain,Ytrain)
lr.n_iter_当参数solver="sag":
![]()
2.4 二元回归与多元回归:重要参数solver & multi_class
来看看鸢尾花数据集上,multinomial和ovr的区别怎么样:
from sklearn.datasets import load_iris
iris = load_iris()
for multi_class in ('multinomial', 'ovr'):
clf = LR(solver='sag', max_iter=100, random_state=42,
multi_class=multi_class).fit(iris.data, iris.target) #打印两种multi_class模式下的训练分数
#%的用法,用%来代替打印的字符串中,想由变量替换的部分。%.3f表示,保留三位小数的浮点数。%s表示,字符串。
#字符串后的%后使用元祖来容纳变量,字符串中有几个%,元祖中就需要有几个变量
print("training score : %.3f (%s)" % (clf.score(iris.data, iris.target),multi_class))
'''
training score : 0.987 (multinomial)
training score : 0.960 (ovr)
'''2.5 样本不平衡与参数class_weight