三维荧光平行因子学习记录--(二)荧光区域积分(FRI)
三维荧光平行因子学习记录–(二)荧光区域积分(FRI)
注:本文仅作为自己的学习记录以备以后复习查阅
所有步骤和代码参考知乎上两位大佬在这里po一下他们的主页:
https://www.zhihu.com/people/uby-23
https://www.zhihu.com/people/ihy-38-65
有兴趣学习三维荧光相关数据处理和分析的可以去看看他们的文章。
前言
荧光区域积分最早是在《2003 Fluorescence Excitation-Emission Matrix Regional Integration to Quantify Spectra for Dissolved Organic Matter》这篇文章里提到的,用于分析荧光数据的方法,在此之前各种荧光光谱技术已被用于表征海洋、水生和土壤DOM(溶解性有机质)样品的来源,但量化荧光光谱特性的技术已受到限制,通常仅使用荧光光谱中的一个、两个或三个数据点。由此文章提出了区域积分的方法对荧光数据进行表征。
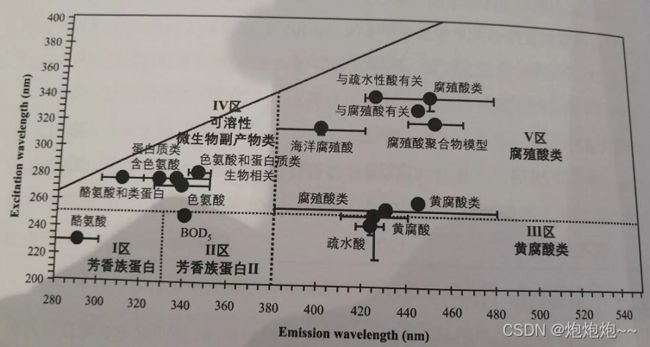
文中的积分区域如下:
一、积分公式
我们假设在选定区域内EEM下方的整合将代表具有相似性质的DOM的累积荧光响应。FRI技术用于整合EEM光谱下的区域(计算第i个区域的体积积分)。
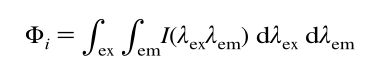
对于离散的数据,体积积分公式的计算如下:
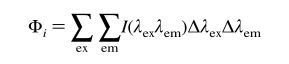
标准化激发发射面积体积(Φi,n,ΦT,n)和荧光响应百分比(Pi,n)计算如下:
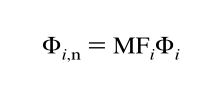
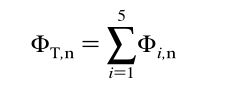
第i块区域占全区域的比例:
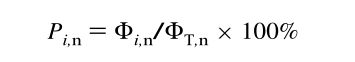
二、数据整理
所有的数据整理参考我的上一篇文章,在这里就不赘述了,默认我们已经有了做好的数据集。
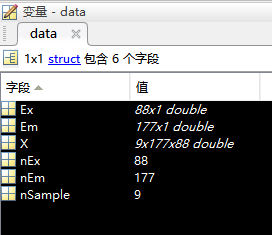
大家注意看一下自己的ex和em的大小。
三、Matlab代码
下面的代码参考知乎上一位作者的,这里po一下他的主页,(如有侵权联系我立即删除)
https://www.zhihu.com/people/ihy-38-65
下面是代码部分,简单说一下,因为代码中没有注释的部分,想要获得较好的积分和绘图效果建议ex在200-450nm,em在250-600nm这样大概的范围内,并且要保证代码中所有积分的波长范围你的数据集中都有包括,不然matlab会报错超过数组索引范围。
下面的代码保存成.m文件放在你要分析数据的路径下面方便调用。

function [P, Phi, MF] = FRI(data,plots)
% FRI:区域积分计算
% Phi:各区域的荧光积分,MF:倍增系数,P:各区域比例
% data:数据集,plots:每次画图数,如[2,3]表示两行三列图,即一次六张图,默认不连续出图而是按下任意键画下一组图
% e.g.绘制图形:[P, Phi, MF] = FRI(data,plots) 不绘制图形:[P, Phi, MF] = FRI(data)
% 只获取各区域积分比例:[P] = FRI(data)
zmin=min(reshape(data.X,(data.nSample*data.nEm*data.nEx),1));
zmax=max(reshape(data.X,(data.nSample*data.nEm*data.nEx),1));
X=data.X;
X(isnan(X))=0;
ex=data.Ex;em=data.Em;
dex=ex(2)-ex(1);dem=em(2)-em(1);
s(1)=(250-ex(1))*(330-em(1));index{1,1}=find(ex<=250);index{1,2}=find(em<=330);
s(2)=(250-ex(1))*(380-330);index{2,1}=find(ex<=250);index{2,2}=find(em>=330&em<=380);
s(3)=(250-ex(1))*(em(end)-380);index{3,1}=find(ex<=250);index{3,2}=find(em>=380);
s(4)=(ex(end)-250)*(380-em(1));index{4,1}=find(ex>=250);index{4,2}=find(em<=380);
s(5)=(ex(end)-250)*(em(end)-380);index{5,1}=find(ex>=250);index{5,2}=find(em>=380);
for i=1:data.nSample
t=squeeze(X(i,:,:));
for j=1:5
ts=t(index{j,2},index{j,1});
Phi(i,j)=(sum(ts(:))-0.5*(sum(ts(1,:))+sum(ts(:,1))+sum(ts(end,:))+sum(ts(:,end)))+0.25*(ts(1,1)+ts(1,end)+ts(end,1)+ts(end,end)))*dex*dem;
MF(i,j)=sum(s)/s(j);
S(i,j)= Phi(i,j)*MF(i,j);
end
for j=1:5
P(i,j)=S(i,j)/sum(S(i,:));
end
end
if nargin>1
figure
for i=1:(plots(1)*plots(2)):data.nSample,pause
m=min(i+plots(1)*plots(2)-1,data.nSample)-i+1;
for j=1:m
subplot(plots(1),plots(2),j)
contourf(em,ex,squeeze(X(i+j-1,:,:))'),colorbar,caxis([zmin,zmax])
line([em(1) em(end)],[250 250],'color','k','LineWidth',0.8)
line([380 380],[ex(1) ex(end)],'color','k','LineWidth',0.8)
line([330 330],[ex(1) 250],'color','k','LineWidth',0.8)
text((330+em(1))/2,(250+ex(1))/2,'Ⅰ','Color','white','FontSize',12,'FontAngle','italic','FontWeight','bold','HorizontalAlignment','center')
text((330+380)/2,(250+ex(1))/2,'Ⅱ','Color','white','FontSize',12,'FontAngle','italic','FontWeight','bold','HorizontalAlignment','center')
text((380+em(end))/2,(250+ex(1))/2,'Ⅲ','Color','white','FontSize',12,'FontAngle','italic','FontWeight','bold','HorizontalAlignment','center')
text((em(1)+380)/2,(250+ex(end))/2,'Ⅳ','Color','white','FontSize',12,'FontAngle','italic','FontWeight','bold','HorizontalAlignment','center')
text((380+em(end))/2,(250+ex(end))/2,'Ⅴ','Color','white','FontSize',12,'FontAngle','italic','FontWeight','bold','HorizontalAlignment','center')
xlabel('Em/nm')
ylabel('Ex/nm')
title(['Sample' num2str(i+j-1)])
end
end
end
end
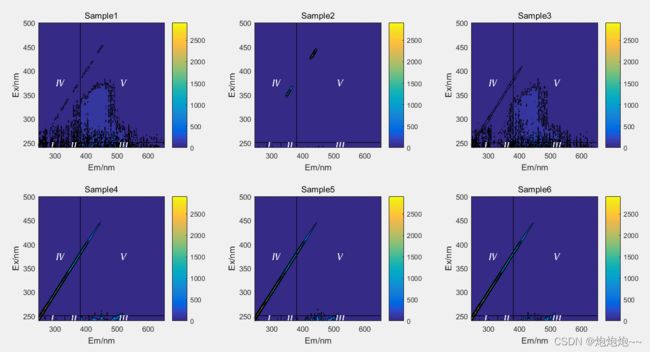
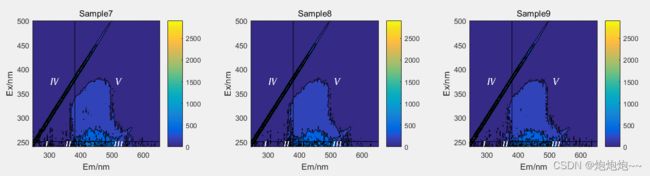
运行效果:
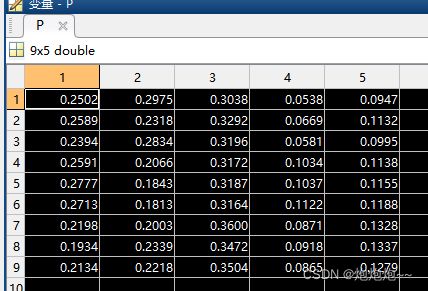
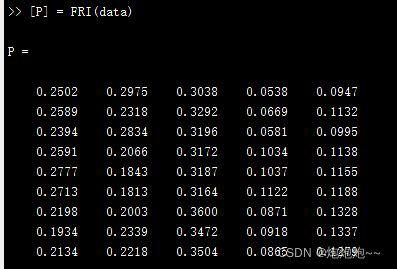
P中生成的列分别对应五个积分区域,行就代表你的样本数量,比如我这里只有九个样本。(下面的也是一样)
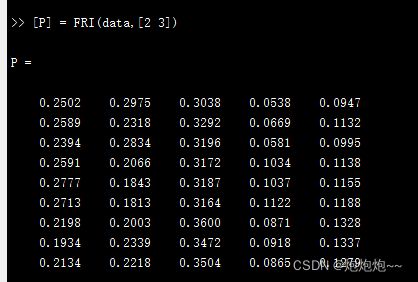
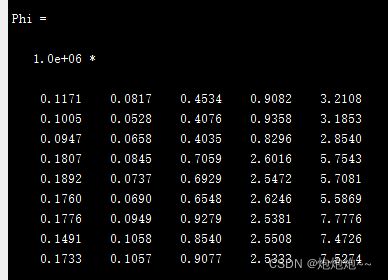
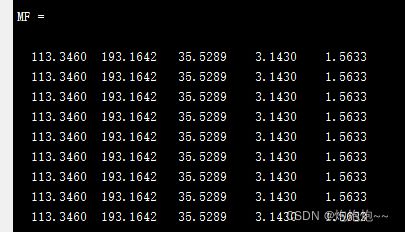
运行代码:[P] = FRI(data,[2 3])
表示画一个两行三列的图来显示数据,运行后默认是不会画的,需要你点击任意键来切换,每一张图都是如此。
注意:在进行荧光区域积分之前建议大家都对数据进行去除散射的处理
我的数据量不是很多,并且从图中也能看出数据处理的并不是特别好,可能也是我做的仪器的原因,实验室仪器的氙灯用的太久了,水拉曼测试两个指标都不通过,勉强为了学习数据分析去收集的这九个数据,还是建议大家在数据分析前也重视数据的预处理,这样才能得到一个较好的分析效果。
以上。