图像特征提取_影像组学特征提取——知行合一(工具篇)
在上一篇"理论篇"中,咱们一起讨论了保证影像组学特征提取的可重复一致性,所需要具备的各项理论知识,包括实现过程中需要注意的重要细节和避免的错误。但大家心中是否始终存有一个疑问:理论我是清楚了,但真的要我自己动手去编程实现吗?我的目标是完成毕业论文或者发表学术论文,特征提取只是其中一步,真的没时间(没实力)去实现这一大套啊。
重复造轮子的事情当然不能干。不用担心,Radiomics的大神们也早已想到了这一点。光说不练假把式,连说带练才是真把式。大咖们虽然没听说过王阳明,也不懂心学,但对“知行合一”的追求,却是共通和共同的。
PyRadiomics
废话铺垫这里,必须引出工具篇的主角,PyRadiomics工具箱了。这是Radiomics网站团队开发和推出的一个开源Python工具包。
Github:https://github.com/Radiomics/pyradiomics
文档网站:https://pyradiomics.readthedocs.io/en/latest/
官方参考文献:Griethuysen J J M V , Fedorov A , Parmar C , et al. Computational Radiomics System to Decode the Radiographic Phenotype[J]. Cancer Research, 2017, 77(21):e104-e107. https://cancerres.aacrjournals.org/content/77/21/e104
如果想系统了解这个工具,推荐阅读上面的官方参考文献。这篇文章发表在Cancer Research期刊上,这个期刊的影响因子(IF)8.7。
一群理工男(Geek),一起写了一个Python库,然后就发了一篇IF 8.7的文章,也算是值得传颂的“佳话”了。也再次印证了之前提到的,影像组学在医工结合、独辟蹊径发表高分文章领域的威力。(多说一句,文章发表在2017年,现在想要走这个捷径,可要慎重了。)
目标
PyRadiomics的研发目标很明确:影像组学研究长期苦于缺少一套标准化的图像预处理和特征提取工具,所以,Radiomics团队要解决这个痛点,并且开源给大家使用。发现痛点、研发工具、开源共享,绝对的极客精神。
功能
目标明确了,那么按照这个目标,PyRadiomics实现了哪些具体功能呢?
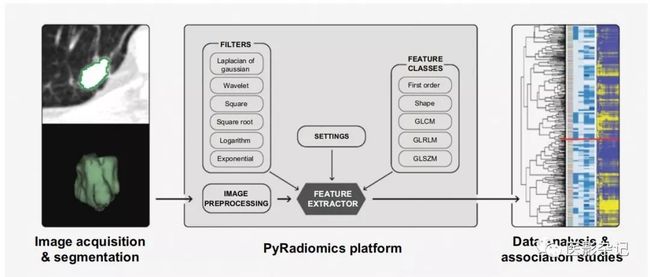
来自参考文献的下图,给出了很好的说明。大家注意,图片中间灰色方框中的内容是PyRadiomics覆盖的功能范围,方框以外的都不在PyRadiomics范围内。说的更具体一些,一个完整的影像组学分析过程包括:1)左侧的图像采集和分割;2)中间的图像处理和特征提取;3)右侧的特征分析。PyRadiomics只负责中间的图像处理和特征提取。
从图片中也可以看到,PyRadiomics主要提供两大类功能:图像过滤和特征提取。下面分别简单介绍。
图像滤波
这部分相对简单。PyRadiomics提供了一系列函数,用于对原始图像进行滤波变换,从而生成小波图像、Laplacian of Gaussian (LoG)图像、以及square, square root, logarithm, 和exponential 图像。最新版本PyRadiomics在2017年论文的基础上,扩展增加了新的滤波输出图像类型。详细信息大家可以直接参考官方在线文档。
https://pyradiomics.readthedocs.io/en/latest/radiomics.html#module-radiomics.imageoperations
对影像组学不熟悉的同学可能有疑惑。直接在原始图像上进行特征提取就可以了,为什么还需要这些不同类型的滤波图像?大家应该看过一些影像组学的论文,里面动辄就提取出上千种特征。这上千种怎么来的?秘诀就是在原始图像以外,还对原始图像衍生出的多种类型滤波图像,并对滤波图像进行了特征提取。
额外说两句。
滤波即卷积。做不同类型的卷积,然后再提取特征,组合成一个大特征向量组,对向量组做筛选,然后进行分析和拟合。这和深度卷积网络在基本思想上,可以说已经达到80%的趋同了。不严格的说,影像组学类似于深度卷积网络的简化版;深度卷积网络则类似于影像组学的扩充版,二者颇有些“大道趋同”的意思。
在图像滤波部分,很多函数是调用SimpleITK来实现的。SimpleITK是基于ITK开发的一个Python库。而ITK则是从1999年开始开发的一个C++开源库,主要提供图像分割和配准功能。从ITK到SimpleITK,再到PyRadiomics,我们既看到了开源软件“九层之台,起于垒土”,也看到了开源软件的“薪火相传”。
特征提取
特征提取是影像组学中最重要的步骤。PyRadiomics工具箱提供了下述7大类特征(Shape-based 3D/2D 作为一类)。
First Order Statistics(19 features)Shape-based (3D)(16 features)Shape-based (2D)(10 features)Gray Level Cooccurence Matrix(24 features)Gray Level Run Length Matrix(16 features)Gray Level Size Zone Matrix(16 features)Neighbouring Gray Tone Difference Matrix(5 features)Gray Level Dependence Matrix(14 features)
上面列出的所有特征种类,除了形状特征,都既可以应用于原始图像,也可以应用于滤波变换后的图像,如小波图像。而形状特征直接且只能在形状Mask上计算和提取。最重要的,PyRadiomics实现的大部分特征都与“理论篇”中“Imaging Biomarker Standardization Initiative” (IBSI)参考文献中的定义一致。也就是说,PyRadiomics是IBSI的官方推荐实现版本。
对于每一类特征,PyRadiomics参考手册网站中,都给出了说明和定义。相比理论篇IBSI,参考手册中的内容更加贴近编程和实现。介绍和讨论了很多编程中需要注意的具体问题,或特殊处理方式。
使用方法
了解PyRadiomics的功能之后,接下来的问题就是,如何使用呢?应该说,开发团队非常体贴用户,提供了两种使用方式:命令行模式和编程模式。
命令行模式
命令行方式就是在搭建好Python和PyRadiomics环境之后,可以像调用DOS命令一样,去启动程序,分析单张或批量影像。命令行方式的最大价值就是,让完全不熟悉Python编程的人员(医生),也可以使用PyRadiomics这个工具,完成自己的研究。(当然,还是需要有一个Python工程师先把环境搭建好。)
单次提取模式
例如,可以使用下面的命令,从单张影像中提取组学特征。
pyradiomics 第一个参数代表原始图像(Image)的存储路径;第二个参数代表对应的分割蒙板(Mask)的存储路径。
批量提取模式
在真正的影像组学研究中,提取单张图像的特征是没有意义。必须能够批量提取大批影像的特征,也就是,需要能够对影像进行“高通量”处理。下面的命令就可以用来分析批量影像。
pyradiomics 指向一个CSV文件,CSV文件的最简单形式是N行2列。每一行的第一列是原始图像(Image)的存储路径;第二列是对应的分割蒙板(Mask)的存储路径。
此外单次提取和批量提取模式都支持额外指定一个参数文件,对特征提取的各项参数进行指定。具体可以参考参考手册网站。
编程模式
编程模式面向具备Python基础编程能力的同学,可以进行更多灵活参数配置,以及自定义处理步骤。对于程序员来说,这部分应该不需要介绍了。身为程序员,看文档、自学上手,应该是基本功,也是乐趣所在了。
补充材料
以上,就是关于PyRadiomics这个工具箱的主要介绍内容。
大家平常总说,一图胜千言。如果有视频的话,那应该能胜“万言”了。这一点,大神们也想到了。专门制作了两段视频,来介绍PyRadiomics工具箱。
一段是偏向科普的:
https://cancerres.aacrjournals.org/content/canres/suppl/2017/11/18/77.21.e104.DC1/178435_3_video_4115604_jrwm6j.mp4
另一段是偏向程序员的:
https://cancerres.aacrjournals.org/content/canres/suppl/2017/11/18/77.21.e104.DC1/178435_3_video_4134730_jsd7cd.mp4
既然视频胜万言,那也就无需多说了。祝大家观影愉快。