传统机器学习(四)聚类算法DBSCAN
传统机器学习(四)聚类算法DBSCAN
1.1 算法概述
DBSCAN(Density-Based Spatial Clustering of Applications with Noise,具有噪声的基于密度的聚类方法)是一种基于密度的空间聚类算法。
该算法将具有足够密度的区域划分为簇,并在具有噪声的空间数据库中发现任意形状的簇,它将簇定义为密度相连的点的最大集合。
1.1.1 基本概念
核心对象: 若某个点的密度达到算法设定的阈值,则其为核心点(即r 邻域内点的数量不小于 minPts )
e-邻域的距离阈值:设定的半径
直接密度可达: 若某点p在点q的 r 域内,且q是核心点,则p-q直接密度可达
密度可达: 若有一个点的序列q0、q1、…qk,对任意qi-qi-1是直接密度可达的则称从q0到qk密度可达,这实际上是直接密度可达的“传播”
边界点:属于某一个类的非核心点,不能发展下线了
噪声点: 不属于任何一个类簇的点,从任何一个核心点出发都是密度不可达的
上面这些点是分布在样本空间的众多样本,现在我们的目标是把这些在样本空间中距离相近的聚成一类。
我们发现A点附近的点密度较大,红色的圆圈根据一定的规则在这里滚啊滚,最终收纳了A附近的5个点,标记为红色也就是定为同一个簇。
其它没有被收纳的根据一样的规则成簇。
形象来说,我们可以认为这是系统在众多样本点中随机选中一个,围绕这个被选中的样本点画一个圆,规定这个圆的半径以及圆内最少包含的样本点,如果在指定半径内有足够多的样本点在内,那么这个圆圈的圆心就转移到这个内部样本点,继续去圈附近其它的样本点,类似传销一样,继续去发展下线。
等到这个滚来滚去的圈发现所圈住的样本点数量少于预先指定的值(minPts),就停止了。那么我们称最开始那个点为核心点,如A,停下来的那个点为边界点,如B、C,没得滚的那个点为离群点,如N)。
基于密度这点有什么好处呢?
我们知道kmeans聚类算法只能处理球形的簇,也就是一个聚成实心的团(这是因为算法本身计算平均距离的局限)。但往往现实中还会有各种形状,比如下面图,环形和不规则形,这个时候,那些传统的聚类算法显然就悲剧了。
1.1.2 DBSCAN工作流程
1.1.3 参数选择及可视化
参数选择
DBSCAN算法的两个参数,这两个参数比较难指定:
-
半径: 半径是最难指定的 ,大了,圈住的就多了,簇的个数就少了;反之,簇的个数就多了,这对我们最后的结果是有影响的。这个时候,K距离可以帮助我们来设定半径r,也就是要找到突变点。
首先选中一个点,计算它和所有其它点的距离,从小到大排序,d1,d2,d3,d4等等,假如我们发现 d3 和 d4 之间的差异很大,于是认为前面的距离是比较合适的,那么就可以指定出来 r 半径的大小。虽然是一个可取的方式,但是有时候比较麻烦 ,大部分还是都试一试进行观察,用k距离需要做大量实验来观察,很难一次性把这些值都选准。
- MinPts: 这个参数就是圈住的点的个数,也相当于是一个密度,一般这个值都是偏小一些,然后进行多次尝试。
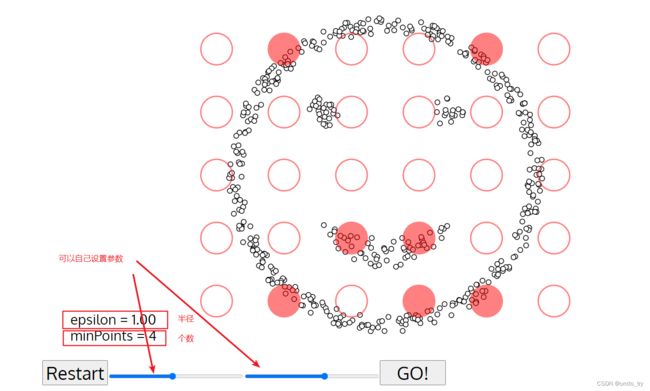
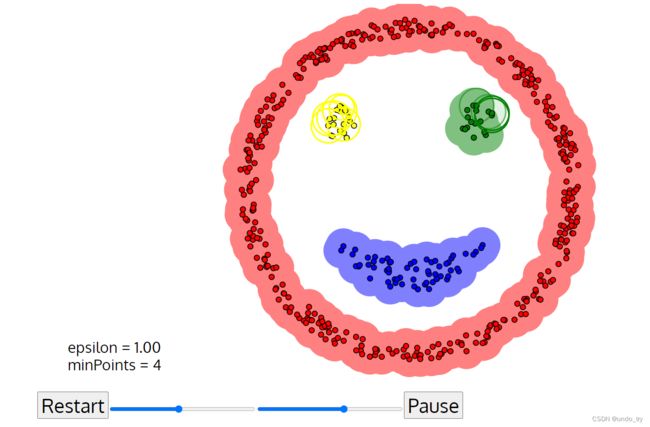
可视化
有一个网站(https://www.naftaliharris.com/blog/visualizing-dbscan-clustering/)可以把我们DBSCAN的迭代过程动态图画出来
设置好参数,点击GO! 就开始聚类了!
1.1.4 DNSCAN优缺点
优点
-
不需要指定簇个数
-
可以发现任意形状的簇
-
擅长找到离群点( 检测任务)
-
两个参数就够了
缺点
-
高维数据有些困难(可以做降维)
-
参数难以选择(参数对结果的影响非常大)
-
Sklearn中效率很慢(数据削减策略)
1.2 DBScan算法案例
1.2.1 不同半径对算法结果影响
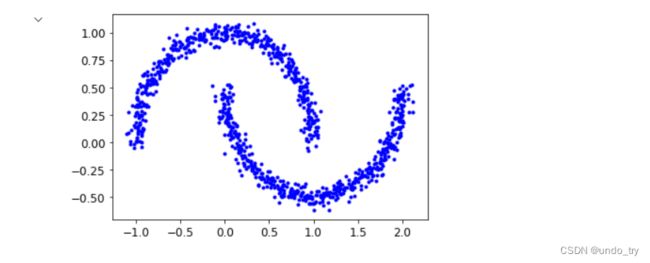
1.2.1.1 利用sklearn中make_moons方法产生数据集
make_moons是函数用来生成数据集,在sklearn.datasets里,具体用法如下:
sklearn.datasets.make_moons(n_samples=100, *, shuffle=True, noise=None, random_state=None)
主要参数作用如下:
n_numbers:生成样本数量
shuffle:是否打乱,类似于将数据集random一下
noise:默认是false,数据集是否加入高斯噪声
random_state:生成随机种子,给定一个int型数据,能够保证每次生成数据相同。
import numpy as np
import os
%matplotlib inline
import matplotlib
import matplotlib.pyplot as plt
plt.rcParams['axes.labelsize'] = 14
plt.rcParams['xtick.labelsize'] = 12
plt.rcParams['ytick.labelsize'] = 12
import warnings
warnings.filterwarnings('ignore')
np.random.seed(42)
# 导入make_moons
from sklearn.datasets import make_moons
X, y = make_moons(n_samples=1000, noise=0.05, random_state=42)
# 绘制图像
plt.plot(X[:,0],X[:,1],'b.')
1.2.2.2 DBScan算法
from sklearn.cluster import DBSCAN
# 设置半径为0.05,阈值为5
dbscan = DBSCAN(eps = 0.05,min_samples=5)
dbscan.fit(X)
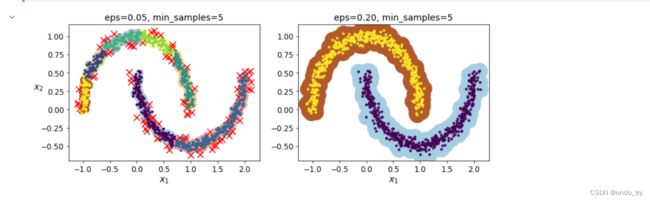
不同半径对聚类结果的影响
dbscan2 = DBSCAN(eps = 0.2,min_samples=5)
dbscan2.fit(X)
# 图像展示
def plot_dbscan(dbscan, X, size, show_xlabels=True, show_ylabels=True):
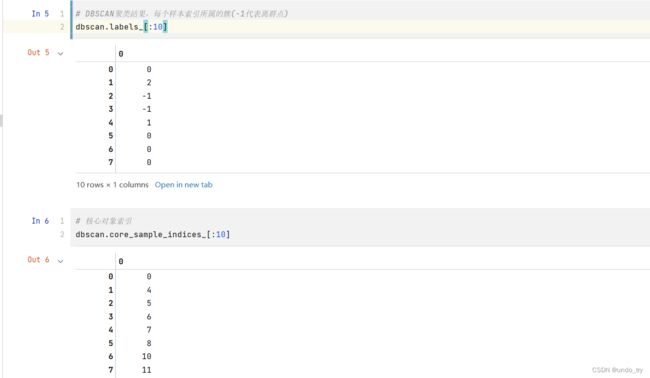
core_mask = np.zeros_like(dbscan.labels_, dtype=bool)
core_mask[dbscan.core_sample_indices_] = True
anomalies_mask = dbscan.labels_ == -1
non_core_mask = ~(core_mask | anomalies_mask)
cores = dbscan.components_
anomalies = X[anomalies_mask]
non_cores = X[non_core_mask]
plt.scatter(cores[:, 0], cores[:, 1],
c=dbscan.labels_[core_mask], marker='o', s=size, cmap="Paired")
plt.scatter(cores[:, 0], cores[:, 1], marker='*', s=20, c=dbscan.labels_[core_mask])
plt.scatter(anomalies[:, 0], anomalies[:, 1],
c="r", marker="x", s=100)
plt.scatter(non_cores[:, 0], non_cores[:, 1], c=dbscan.labels_[non_core_mask], marker=".")
if show_xlabels:
plt.xlabel("$x_1$", fontsize=14)
else:
plt.tick_params(labelbottom='off')
if show_ylabels:
plt.ylabel("$x_2$", fontsize=14, rotation=0)
else:
plt.tick_params(labelleft='off')
plt.title("eps={:.2f}, min_samples={}".format(dbscan.eps, dbscan.min_samples), fontsize=14)
plt.figure(figsize=(12, 4))
plt.subplot(121)
plot_dbscan(dbscan, X, size=100)
plt.subplot(122)
plot_dbscan(dbscan2, X, size=600, show_ylabels=False)
plt.show()
1.2.2 鸢尾花数据集的DBSCAN的聚类案例
import pandas as pd
from sklearn.datasets import load_iris
# 导入数据,sklearn自带鸢尾花数据集
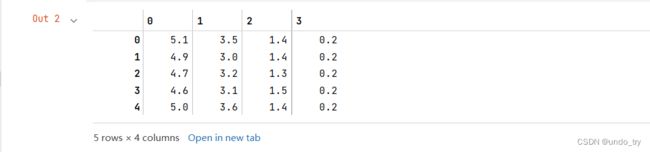
iris = load_iris().data
iris[:5]
from sklearn.cluster import DBSCAN
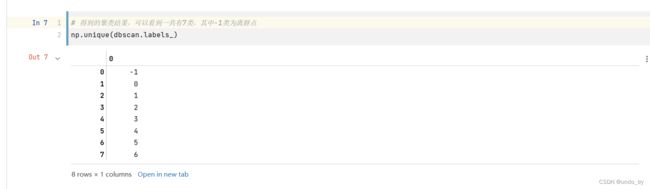
iris_db = DBSCAN(eps=0.6,min_samples=4).fit_predict(iris)
# 设置半径为0.6,最小样本量为2,建模
db = DBSCAN(eps=10, min_samples=2).fit(iris)
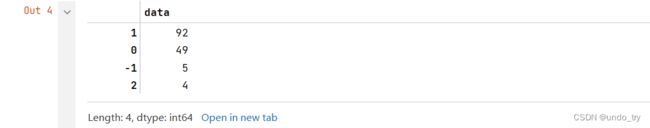
# 统计每一类的数量
counts = pd.value_counts(iris_db,sort=True)
counts
# 可视化
import matplotlib.pyplot as plt
plt.rcParams['font.sans-serif'] = [u'Microsoft YaHei']
fig,ax = plt.subplots(1,2,figsize=(12,12))
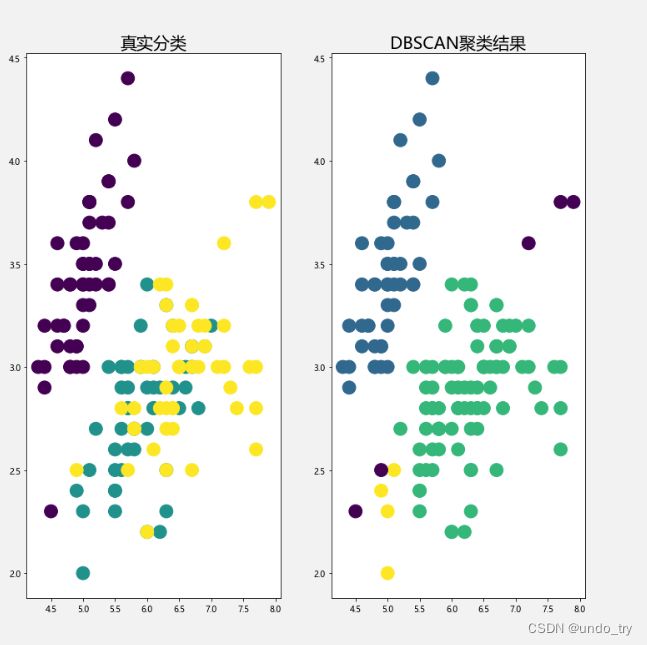
# 画真实数据结果
ax1 = ax[0]
ax1.scatter(x=iris[:,0],y=iris[:,1],s=250,c=load_iris().target)
ax1.set_title('真实分类',fontsize=20)
# 画聚类后的结果
ax2 = ax[1]
ax2.scatter(x=iris[:,0],y=iris[:,1],s=250,c=iris_db)
ax2.set_title('DBSCAN聚类结果',fontsize=20)
plt.show()
我们可以从上面这个图里观察聚类效果的好坏,但是当数据量很大,或者指标很多的时候,观察起来就会非常麻烦。
这时候可以使用轮廓系数来判定结果好坏,聚类结果的轮廓系数,定义为S,是该聚类是否合理、有效的度量。
聚类结果的轮廓系数的取值在[-1,1]之间,值越大,说明同类样本相距约近,不同样本相距越远,则聚类效果越好。
轮廓系数以及其他的评价函数都定义在sklearn.metrics模块中,在sklearn中函数silhouette_score()计算所有点的平均轮廓系数。
from sklearn import metrics
# 就是下面这个函数可以计算轮廓系数
score = metrics.silhouette_score(iris,iris_db)
score
# 0.42300259179079075
1.2.3 k-means和DBSCAN聚类对比
import matplotlib.pyplot as plt
from sklearn.cluster import DBSCAN
from sklearn.cluster import KMeans
from sklearn import datasets
# 生成数据
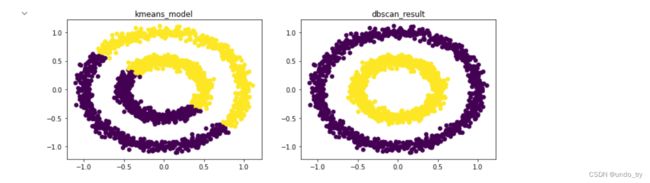
x, y = datasets.make_circles(n_samples=2000, factor=0.5, noise=0.05)
# k-means方法聚类
kmeans_model = KMeans(n_clusters=2)
kmeans_model.fit(x)
kmeans_result = kmeans_model.predict(x)
# DBSCAN方法聚类
dbscan_model = DBSCAN(eps=0.2, min_samples=50)
dbscan_model.fit(x)
dbscan_result = dbscan_model.fit_predict(x)
# 绘制图像
plt.figure(figsize=(12, 4))
plt.subplot(121)
plt.scatter(x[:, 0], x[:, 1], c=kmeans_result)
plt.title('kmeans_model')
plt.subplot(122)
plt.scatter(x[:, 0], x[:, 1], c=dbscan_result)
plt.title('dbscan_result')
plt.show()