【大数据平台】基于Spark的美国新冠肺炎疫情数据分析及预测
(本实验系中国地质大学(武汉)2022年秋期大数据平台及应用课程设计)
一、选题背景
新型冠状病毒疫情是由严重急性呼吸系统综合征冠状病毒2(SARS-CoV-2)导致的2019冠状病毒病(COVID-19)所引发的全球大流行疫情。该疾病在2019年末于中华人民共和国湖北省武汉市首次爆发,随后在2020年初迅速扩散至全球多国,逐渐变成一场全球性的大瘟疫。截至到2022年12月7日,全球已累计报告超过6.43亿例确诊病例,其中超过663.7万人死亡,是人类历史上最大规模的流行病之一。
这次疫情导致严重的全球性的社会和经济混乱,被视为人类自第二次世界大战以来面临的最严峻危机,并使全球经济陷入自从1930年代的大萧条以来最大的衰退。危机爆发的初期,亦遇上全球医疗与民生用品因为恐慌性消费导致供应不足、传播假新闻与针对不同族裔产生种族或地域等歧视的问题。许多教育机构和公共区域被部分或完全关闭,很多活动被取消或推迟。而疫情扩散对全球航空、旅游、娱乐、体育、石油市场、金融市场等方面造成巨大影响并在经济重启后仍持续多年。
鉴于新冠肺炎疫情的巨大影响,我们只有先做到客观而全面地认识,才能谋求科学有效的应对方法。本项目使用Spark等大数据处理工具,对美国逾两年的疫情状况进行分析,以期得到数据背后的疫情发展规律,更加客观全面地看待疫情现状,为中国的疫情防控举措提供科学合理的参考意见。
二、数据集介绍
链接:https://pan.baidu.com/s/1ke36AeJ0ThB_6zpNZwW4fQ?pwd=0294
提取码:0294
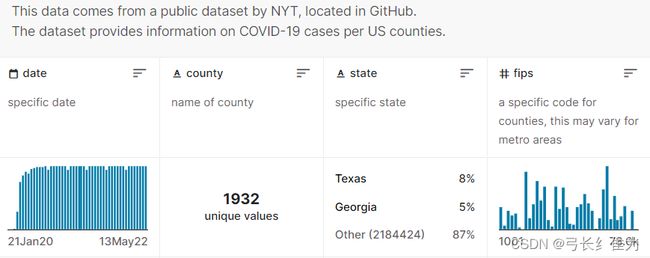
本项目的数据集来自数据竞赛网站Kaggle的美国各县疫情数据集US counties COVID 19 dataset,包含了从2020年1月21日美国新增第一例确诊病例到2022年5月13日停止更新期间的250万多条记录。该数据集以csv格式组织,各属性的含义如下:
date表示日期,county表示区县名称,state表示州名,fips表示各县位置编码,cases表示截止当前日期累计确诊病例数,deaths表示截止当前日期累计死亡病例数。FIPS(Federal Information Processing System)编码是美国国内各地区各自的唯一编码,用以区分不同地理实体。每个州各自均有一个唯一的二位FIPS编码,每个州下的所有郡县级地区同样有各自唯一的五位FIPS编码(其中前两位是一样的,都是该州所属FIPS编码)。
三、Spark简介
Spark最初由美国加州伯克利大学的AMP 实验室于2009年开发,是基于内存计算的大数据并行计算框架,可用于构建大型的、低延迟的数据分析应用程序。 Spark于2013年加入Apache孵化器项目后发展迅猛,如今已成为Apache软件基金会最重要的三大分布式计算系统开源项目之一(Hadoop、Spark、Storm)。值得一提的是,Spark在2014年打破了Hadoop保持的基准排序记录。Spark使用206个节点,用时23分钟完成了100TB数据的排序,而Hadoop使用了2000个节点,耗时72分钟,完成了100TB数据的排序。也就是说Spark用十分之一的计算资源,获得了比Hadoop快3倍的速度。
Spark具有如下几个主要特点:运行速度快:使用DAG执行引擎以支持循环数据流与内存计算;容易使用:支持使用Scala、Java、Python和R语言进行编程,可以通过 Spark Shell进行交互式编程;通用性:Spark提供了完整而强大的技术栈,包括SQL查询、流式计算、机器学习和图算法组件;运行模式多样:可运行于独立的集群模式中,可运行于Hadoop中,也可运行于Amazon EC2等云环境中,并且可以访问HDFS、Cassandra、HBase和Hive等多种数据源。
传统大数据处理框架Hadoop存在如下一些缺点:表达能力有限;磁盘IO开销大;延迟高;任务之间的衔接涉及IO开销;在前一个任务执行完成之前,其他任务就无法开始,难以胜任复杂、多阶段的计算任务。Spark在借鉴Hadoop MapReduce优点的同时,很好地解决了MapReduce所面临的问题。相比于Hadoop MapReduce,Spark主要具有如下优点:Spark的计算模式也属于MapReduce,但不局限于Map和Reduce操作,还提供了多种数据集操作类型,编程模型比Hadoop MapReduce更灵活;Spark提供了内存计算,可将中间结果放到内存中,对于迭代运算效率更高;Spark基于DAG的任务调度执行机制,要优于Hadoop MapReduce的迭代执行机制。
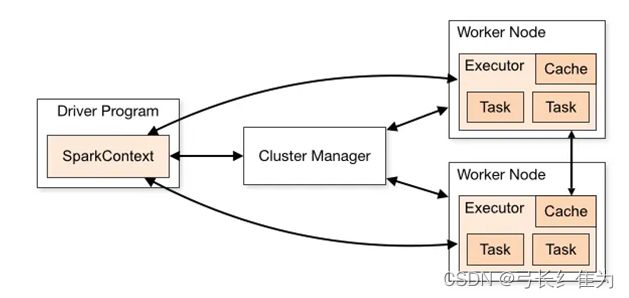
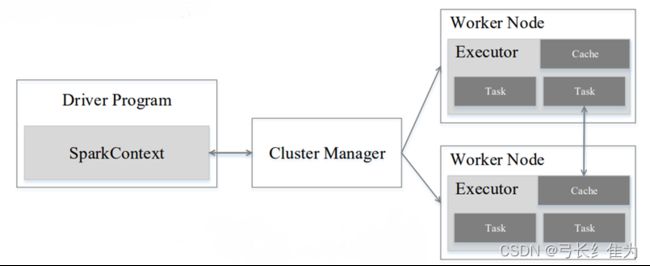
Spark的运行架构包括集群管理器(Cluster Manager)、运行作业任务的工作节点(Worker Node)、每个应用的任务控制节点(Driver)和每个工作节点上负责具体任务的执行进程(Executor)。其中,集群管理器可以是Spark自带的资源管理器,也可以是YARN或Mesos等资源管理框架。与Hadoop MapReduce计算框架相比,Spark所采用的Executor有两个优点。一是利用多线程来执行具体的任务(HadoopMapReduce采用的是进程模型),减少任务的启动开销。二是 Executor中有一个BlockManager存储模块,会将内存和磁盘共同作为存储设备,当需要多轮迭代计算时,可以将中间结果存储到这个存储模块里,下次需要时就可以直接读该存储模块里的数据,而不需要读写到 HDFS 等文件系统里,因而有效减少了 IO 开销;或者在交互式查询场景下,Executor预先将表缓存到该存储系统上,从而可以提高读写IO的性能。
Spark运行基本流程如下图所示,流程如下。
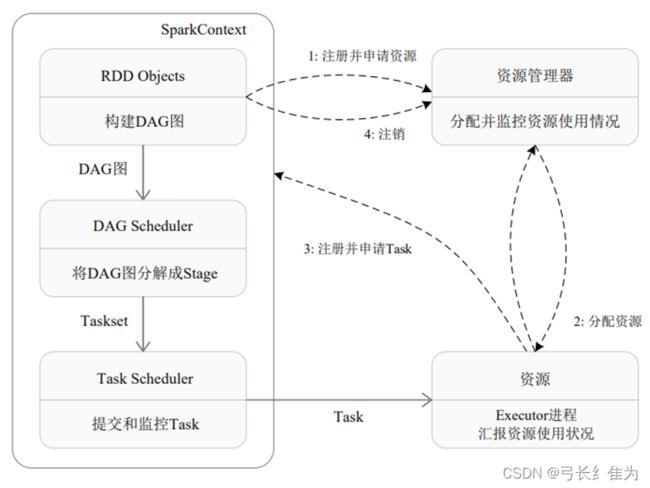
(1)当一个Spark应用被提交时,首先需要为这个应用构建起基本的运行环境,即由任务控制节点(Driver)创建一个SparkContext,由SparkContext负责和资源管理器—Cluster Manager的通信,以及进行资源的申请、任务的分配和监控等。SparkContext 会向资源管理器注册并申请运行Executor的资源。
(2)资源管理器为Executor分配资源,并启动Executor进程,Executor运行情况将随着“心跳”发送到资源管理器上。
(3)SparkContext 根据 RDD 的依赖关系构建 DAG,并将 DAG 提交给 DAG 调度器(DAGScheduler)进行解析,将DAG分解成多个“阶段”(每个阶段都是一个任务集),并且计算出各个阶段之间的依赖关系,然后把一个个“任务集”提交给底层的任务调度器(TaskScheduler)进行处理;Executor 向 SparkContext 申请任务,任务调度器将任务分发给 Executor 运行,同时SparkContext将应用程序代码发放给Executor。
(4)任务在Executor上运行,把执行结果反馈给任务调度器,然后反馈给DAG调度器,运行完毕后写入数据并释放所有资源。
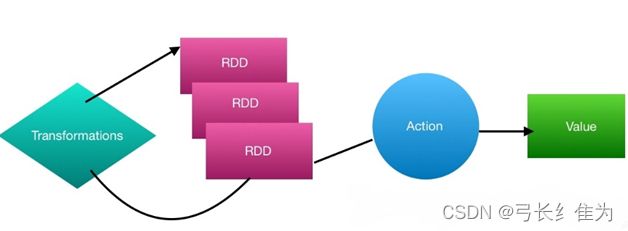
Spark的核心建立在统一的抽象RDD之上,这使得Spark的各个组件可以无缝地进行集成,在同一个应用程序中完成大数据计算任务。一个RDD就是一个分布式对象集合,本质上是一个只读的分区记录集合。每个RDD可以分成多个分区,每个分区就是一个数据集片段,并且一个 RDD 的不同分区可以被保存到集群中不同的节点上,从而可以在集群中的不同节点上进行并行计算。RDD提供了一组丰富的操作以支持常见的数据运算,分为“行动”(Action)和“转换”(Transformation)两种类型,前者用于执行计算并指定输出的形式,后者指定RDD之间的相互依赖关系。两类操作的主要区别是,转换操作(如map、filter、groupBy、join等)接受RDD并返回RDD,而行动操作(如count、collect等)接受RDD但是返回非RDD(即输出一个值或结果)。RDD采用了惰性调用,即在RDD的执行过程中,真正的计算发生在RDD的“行动”操作,对于“行动”之前的所有“转换”操作,Spark只是记录下“转换”操作应用的一些基础数据集以及RDD生成的轨迹,即相互之间的依赖关系,而不会触发真正的计算。
四、实验环境搭建
由于虚拟机内存大小的限制,在虚拟机中运行程序会出现运行速度过慢甚至运行失败等问题,所以直接在Windows操作系统中重新搭建实验环境。
1、安装JDK1.8。
2、配置环境变量。点击我的电脑-属性-高级系统设置-环境变量,点击新建,创建JAVA_HOME。
3、安装Hadoop2.7.1。
不要忘记配置环境变量:
4、安装winutils。Hadoop主要基于Linux编写,winutil.exe主要用于模拟Linux下的目录环境,因此Hadoop若想要在Windows下运行,需要winutil.exe的帮助。下载完成后找到相应的Hadoop版本,这里我们安装的是2.7.1,进入该目录,将bin目录下的所有内容复制,粘贴到Hadoop2.7.1安装目录的bin目录下。
5、修改hadoop-env.cmd。打开Hadoop安装目录,找到hadoop-env.cmd文件,指定JDK的路径。
6、安装Python3.6。由于版本问题,使用Hadoop2.7和Spark2.4版本只能使用Python3.6,所以需要安装Python3.6。建议使用Anaconda来下载Python。
7、安装Spark并配置环境变量。
8、进入Spark的安装目录,将pyspark复制到Anaconda下的python36目录。
9、安装Py4J。在Python3.6环境的Scripts文件夹打开cmd窗口使用pip安装。Py4J 是Python和Java的互调接口,使得Python程序可以利用Python解释器直接调用Java虚拟机中的Java对象,也可以让Java调用Python对象。
这样,环境搭建已经完成。我们打开cmd窗口输入spark-shell:
说明安装成功。
五、基于Spark的数据分析及可视化
首先导入所需要的包:
import pandas as pd
from pyspark import SparkConf,SparkContext
from pyspark.sql import Row
from pyspark.sql.types import *
from pyspark.sql import SparkSession
from datetime import datetime
import pyspark.sql.functions as func原始数据集是csv格式的,为方便Spark读取,需要转化为txt格式:
data = pd.read_csv('C:\\Users\\26909\\Desktop\\us-counties.csv')
with open('us-counties.txt', 'a+', encoding='utf-8') as f:
for line in data.values:
f.write((str(line[0]) + '\t' + str(line[1]) + '\t'
+ str(line[2]) + '\t' + str(line[3]) + '\t' + str(line[4]) + '\n'))
下面创建一个spark对象作为编程入口:
spark = SparkSession.builder.config(conf=SparkConf()).getOrCreate()因为其构造函数私有,所以需要用builder方法创建SparkSession对象。SparkSession内部封装了SparkContext,所以计算实际上是由SparkContext完成的。
在Spark2.0版本之前,使用Spark必须先创建SparkConf和SparkContext;在Spark2.0中只要创建一个SparkSession就够了,SparkConf、SparkContext和SQLContext都已经被封装在SparkSession当中。
下图说明了SparkContext在Spark中的主要功能:
从图中可以看到SparkContext起到中介的作用,通过它来使用Spark其他功能。每一个JVM都有一个对应的SparkContext,Driver Program通过SparkContext连接到集群管理器来实现对集群中任务的控制。Spark配置参数的设置以及对SQLContext、HiveContext和StreamingContext的控制也要通过SparkContext进行。然而在Spark2.0中上述的所有功能都是通过SparkSession来完成的,同时SparkSession也简化了DataFrame/Dataset API的使用和对数据的操作。
fields = [StructField("date", DateType(), False), StructField("county", StringType(), False),
StructField("state", StringType(), False),
StructField("cases", IntegerType(), False), StructField("deaths", IntegerType(), False), ]
schema = StructType(fields)
一个StructField记录了列名、列类型、列是否运行为空;多个StructField组成一个StructType对象,一个StructType对象描述一个DataFrame。
rdd0 = spark.sparkContext.textFile("us-counties.txt")
rdd1 = rdd0.map(lambda x: x.split("\t")).map(lambda p: Row(toDate(p[0]), p[1], p[2], int(p[3]), int(p[4])))
SparkContext.textFile()从HDFS、本地文件系统或任何Hadoop支持的文件系统读取文本文件,并将其作为字符串的RDD返回。同时,一行数据被描述为Row对象。rdd1是rdd0进行map transformation的结果,由于惰性机制,并不是立即执行计算。
如上图所示,Spark对RDD的操作可以整体分为两类:Transformation和Action。这里的Transformation可以翻译为转换,表示是针对RDD中数据的转换操作,主要会针对已有的RDD创建一个新的RDD,常见的有map、flatMap、filter等。Action可以翻译为执行,表示是触发任务执行的操作,主要对RDD进行最后的操作,比如遍历、 reduce、保存到文件等,并且还可以把结果返回给Driver程序。
不管是Transformation里面的操作还是Action里面的操作,我们一般会把它们称之为算子,例如:map 算子、reduce算子,其中Transformation算子有一个特性:lazy,lazy特性在这里指的是如果一个Spark任务中只定义了transformation算子,那么即使你执行这个任务,任务中的算子也不会执行,也就是说,transformation是不会触发Spark任务的执行,它们只是记录了对RDD所做的操作。只有当transformation之后,接着执行了一个action操作,那么所有的transformation才会执行。Spark通过惰性机制来进行底层的Spark任务执行的优化,避免产生过多中间结果。
shemaUsInfo = spark.createDataFrame(rdd1, schema)在SQLContext中使用createDataFrame可以创建DataFrame。这里通过row+schema来创建Dataframe。
shemaUsInfo.createOrReplaceTempView("usInfo")
df = shemaUsInfo.groupBy("date").agg(func.sum("cases"), func.sum("deaths")).sort(shemaUsInfo["date"].asc())
# 列重命名
df1 = df.withColumnRenamed("sum(cases)", "cases").withColumnRenamed("sum(deaths)", "deaths")
df1.repartition(1).write.json("result1.json") # 写入hdfs
# 注册为临时表供下一步使用
df1.createOrReplaceTempView("ustotal")
将原始数据注册为一个名为usInfo的表,再将同一日期的病例数量和死亡数量进行聚合,注册为ustotal表。
这样,准备工作已全部完成,接下来就可以使用SQL语句对usInfo、ustotal等表进行筛选,从而进行分析。
下面是可视化分析:
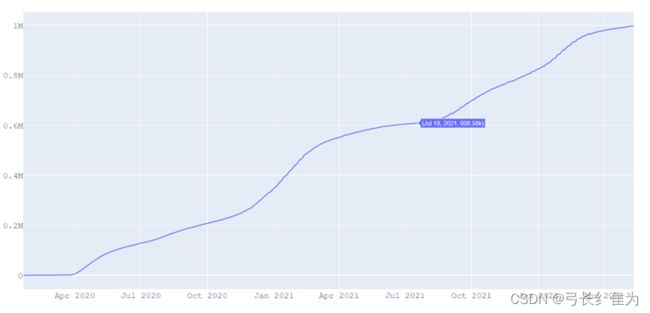
图1 美国确诊病例增长曲线
可以看出美国疫情总体经历了两波疫情增长高峰,第一波出现在2020年与2021年之交,第二波开始于2021年秋天,一直持续到2022年春天。截止2022年5月13日,全美已累计确诊8236.71万例。
图2 美国死亡病例增长曲线
截止2022年5月13日,全美已累计死亡998279例。与确诊病例增长曲线对比可以看出,死亡病例增长最快的时期与新增病例增速最快的时期基本重合。
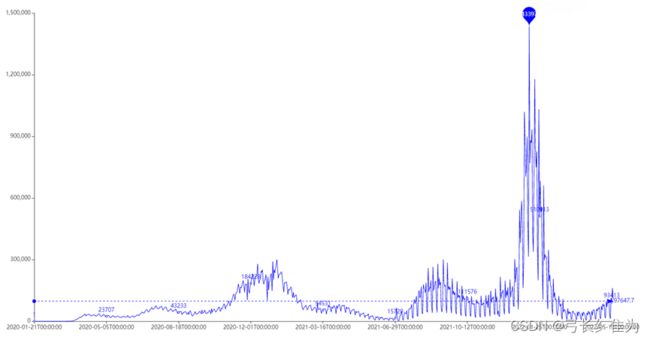
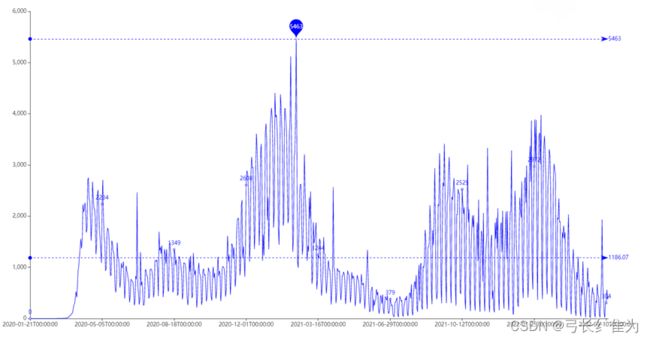
图3 美国每日新增确诊曲线图
图4 新华社有关美国日增新冠确诊病例超百万例的报道
每年10月至次年3月为疫情高发期,在2022年1月尤为明显,该月确诊病例暴增,甚至某些天日增超过百万例,与新闻报道的时间吻合。
图5 美国每日新增死亡病例数曲线图
冬季和春季是死亡病例的高发期,且死亡病例增长最快的时期与新增确诊病例增速最快的时期基本重合。
图6 美国分州疫情地图
图7 美国分县疫情地图
美国东西部沿海地区经济发达,人口稠密,确诊人数较多,如加利福尼亚、佛罗里达、德克萨斯、纽约等州。
图8 确诊数量词云图
全美确诊人数位居前列的州有加利福尼亚州、德克萨斯州、纽约州、伊利诺伊州、宾夕法尼亚州、新泽西州、北卡罗莱纳州、马萨诸塞州和亚利桑那州等。
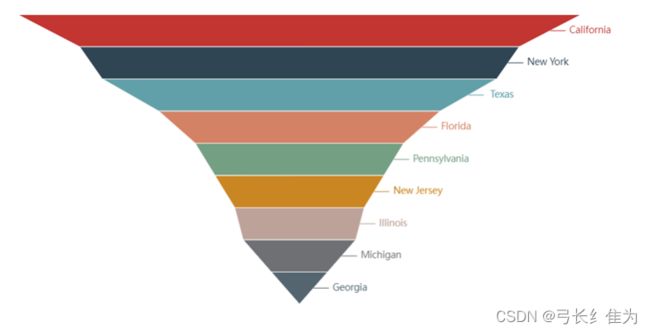
图9 各州死亡病例Top10
死亡病例最多的前10个州依次为加利福尼亚州、纽约州、德克萨斯州、佛罗里达州、宾夕法尼亚州、新泽西州、伊利诺伊州、密歇根州和佐治亚州。
图10 美国确诊病例各月份占比饼状图
从该饼状图可以看出,美国确诊病例3和4月占比最大,其次是1和2月。究其原因,可能是春季气候变化无常,早晚温差较大,是传染病的高发季节;而冬季温度较低,更利于新冠肺炎病毒存活。
我们还可以对美国新冠肺炎疫情数据进行聚类分析。DBSCAN(Density-Based Spatial Clustering of Applications with Noise,具有噪声的基于密度的聚类方法)是一种典型的基于密度的空间聚类算法。和K-Means这样的一般只适用于凸样本集的聚类相比,DBSCAN既可以适用于凸样本集,也可以适用于非凸样本集。该算法将具有足够密度的区域划分为簇,并在具有噪声的空间数据库中发现任意形状的簇,它将簇定义为密度相连的点的最大集合。
该算法利用基于密度的聚类的概念,即要求聚类空间中的一定区域内所包含对象(点或其他空间对象)的数目不小于某一给定阈值。DBSCAN算法的显著优点是聚类速度快且能够有效处理噪声点和发现任意形状的空间聚类。但是当空间聚类的密度不均匀、聚类间距差相差很大时,聚类质量较差。
DBSCAN的优点在于,首先它可以对任意形状的稠密数据集进行聚类,而K-Means之类的聚类算法一般只适用于凸数据集;其次它可以在聚类的同时发现异常点,对数据集中的异常点不敏感;且DBSCAN聚类结果没有偏倚,而K-Means之类的聚类算法初始值对聚类结果有很大影响。
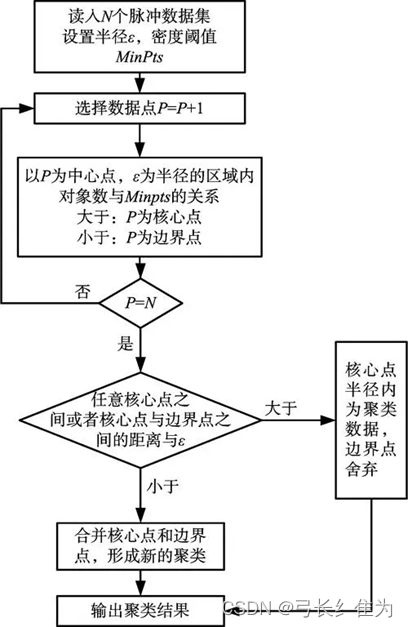
DBSCAN是基于一组邻域来描述样本集的紧密程度的,参数(ϵ, MinPts)用来描述邻域的样本分布紧密程度。其中,ϵ描述了某一样本的邻域距离阈值,MinPts描述了某一样本的距离为ϵ的邻域中样本个数的阈值。下面是DBSCAN聚类算法的流程图:
下图为可视化的聚类结果:
其中x、y轴分别为病例人数和死亡人数。为方便显示,将坐标轴比例设置为log,坐标取值以指数量级增长。被归为一类的样本具有一定程度的相似性,可以针对这些时间节点的确诊病例进行进一步的深入研究。
六、疫情数据建模预测
从前面的分析可以看出,疫情数据表现出了一定的季节特性,可以认为是一种时间序列数据。在对时间序列数据进行预测时,一个简单的思路是认为离预测点越近的点所起的作用越大。例如对下个月的体重进行预测,这个月的数据影响力更大些。假设随着时间变化权重以指数方式下降,最终年代久远的数据权重将接近于0。将权重按照指数级进行衰减,这就是指数平滑法的基本思想。
指数平滑法有几种不同形式:一次指数平滑法针对没有趋势和季节性的序列,二次指数平滑法针对有趋势但没有季节性的序列,三次指数平滑法针对有趋势也有季节性的序列。Holt-Winters特指三次指数平滑法。
所有的指数平滑法都要更新上一时间步长的计算结果,并使用当前时间步长的数据中包含的新信息。它们通过混合新信息和旧信息来实现,而相关的新旧信息的权重由一个可调整的参数来控制。
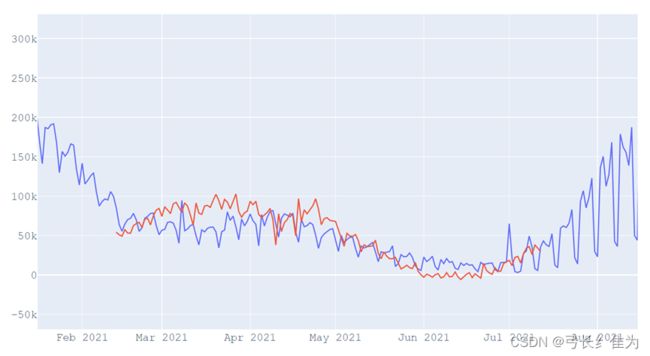
Statsmodels是Python进行拟合多种统计模型、进行统计试验和数据探索可视化的库,它提供了实现指数平滑法的ExponentialSmoothing模型,需要指定5个参数,第一个endog就是时间序列数据;第二个trend是趋势,有三种可选项,就是加法趋势、乘法趋势还有None;第三个damped是衰减,Boolean决定是否对趋势进行衰减;第四个seasonal是季节性(周期),也是三种选项,加法、乘法还有None;第五个seasonal_periods,季节性周期,int型,holt-winter要考虑的季节的数量。经过训练集划分和参数调整,最终的预测结果与真实数据可视化如下:
其中红色曲线为预测值,蓝色曲线为真实值。可以看出二者变化趋势基本一致,但预测精度有待改善。
八、源代码
Spark01.py
import pandas as pd
from pyspark import SparkConf,SparkContext
from pyspark.sql import Row
from pyspark.sql.types import *
from pyspark.sql import SparkSession
from datetime import datetime
import pyspark.sql.functions as func
# .csv->.txt
data = pd.read_csv('C:\\Users\\26909\\Desktop\\us-counties.csv')
with open('us-counties.txt', 'a+', encoding='utf-8') as f:
for line in data.values:
f.write((str(line[0]) + '\t' + str(line[1]) + '\t'
+ str(line[2]) + '\t' + str(line[3]) + '\t' + str(line[4]) + '\n'))
def toDate(inputStr):
newStr = ""
if len(inputStr) == 8:
s1 = inputStr[0:4]
s2 = inputStr[5:6]
s3 = inputStr[7]
newStr = s1+"-"+"0"+s2+"-"+"0"+s3
else:
s1 = inputStr[0:4]
s2 = inputStr[5:6]
s3 = inputStr[7:]
newStr = s1+"-"+"0"+s2+"-"+s3
date = datetime.strptime(newStr, "%Y-%m-%d")
return date
spark = SparkSession.builder.config(conf=SparkConf()).getOrCreate()
fields = [StructField("date", DateType(), False), StructField("county", StringType(), False),
StructField("state", StringType(), False),
StructField("cases", IntegerType(), False), StructField("deaths", IntegerType(), False), ]
schema = StructType(fields)
rdd0 = spark.sparkContext.textFile("us-counties.txt")
rdd1 = rdd0.map(lambda x: x.split("\t")).map(lambda p: Row(toDate(p[0]), p[1], p[2], int(p[3]), int(p[4])))
shemaUsInfo = spark.createDataFrame(rdd1, schema)
shemaUsInfo.createOrReplaceTempView("usInfo")
# 1.计算每日的累计确诊病例数和死亡数
df = shemaUsInfo.groupBy("date").agg(func.sum("cases"), func.sum("deaths")).sort(shemaUsInfo["date"].asc())
# 列重命名
df1 = df.withColumnRenamed("sum(cases)", "cases").withColumnRenamed("sum(deaths)", "deaths")
df1.repartition(1).write.json("result1.json") # 写入hdfs
# 注册为临时表供下一步使用
df1.createOrReplaceTempView("ustotal")
# 2.计算每日较昨日的新增确诊病例数和死亡病例数
df2 = spark.sql(
"select t1.date,t1.cases-t2.cases as caseIncrease,t1.deaths-t2.deaths as deathIncrease from ustotal t1,ustotal t2 where t1.date = date_add(t2.date,1)")
df2.sort(df2["date"].asc()).repartition(1).write.json("result2.json") # 写入hdfs
# 3.统计截止5.19日 美国各州的累计确诊人数和死亡人数
df3 = spark.sql(
"select date,state,sum(cases) as totalCases,sum(deaths) as totalDeaths,round(sum(deaths)/sum(cases),4) as deathRate from usInfo where date = to_date('2020-05-19','yyyy-MM-dd') group by date,state")
df3.sort(df3["totalCases"].desc()).repartition(1).write.json("result3.json") # 写入hdfs
df3.createOrReplaceTempView("eachStateInfo")
# 4.找出美国确诊最多的10个州
df4 = spark.sql("select date,state,totalCases from eachStateInfo order by totalCases desc limit 56")
df4.repartition(1).write.json("result4.json")
# 5.找出美国死亡最多的10个州
df5 = spark.sql("select date,state,totalDeaths from eachStateInfo order by totalDeaths desc limit 10")
df5.repartition(1).write.json("result5.json")
# 6.找出美国确诊最少的10个州
df6 = spark.sql("select date,state,totalCases from eachStateInfo order by totalCases asc limit 10")
df6.repartition(1).write.json("result6.json")
# 7.找出美国死亡最少的10个州
df7 = spark.sql("select date,state,totalDeaths from eachStateInfo order by totalDeaths asc limit 10")
df7.repartition(1).write.json("result7.json")
# 8.统计截止5.19全美和各州的病死率
df8 = spark.sql(
"select 1 as sign,date,'USA' as state,round(sum(totalDeaths)/sum(totalCases),4) as deathRate from eachStateInfo group by date union select 2 as sign,date,state,deathRate from eachStateInfo").cache()
df8.sort(df8["sign"].asc(), df8["deathRate"].desc()).repartition(1).write.json("result8.json")
Spark02.py
from pyecharts import options as opts
from pyecharts.charts import Bar
from pyecharts.charts import Line
from pyecharts.components import Table
from pyecharts.charts import WordCloud
from pyecharts.charts import Pie
from pyecharts.charts import Funnel
from pyecharts.charts import Scatter
from pyecharts.charts import PictorialBar
from pyecharts.options import ComponentTitleOpts
from pyecharts.globals import SymbolType
import json
# 1.画出每日的累计确诊病例数和死亡数——>双柱状图
def drawChart_1(index):
root = "D:\PyProject\Pyspark\keshe\\result" + str(index) + "/part-00000.json"
date = []
cases = []
deaths = []
with open(root, 'r') as f:
while True:
line = f.readline()
if not line: # 到 EOF,返回空字符串,则终止循环
break
js = json.loads(line)
date.append(str(js['date']))
cases.append(int(js['cases']))
deaths.append(int(js['deaths']))
d = (
Bar()
.add_xaxis(date)
.add_yaxis("累计确诊人数", cases, stack="stack1")
.add_yaxis("累计死亡人数", deaths, stack="stack1")
.set_series_opts(label_opts=opts.LabelOpts(is_show=False))
.set_global_opts(title_opts=opts.TitleOpts(title="美国每日累计确诊和死亡人数"))
.render("result1.html")
)
# 2.画出每日的新增确诊病例数和死亡数——>折线图
def drawChart_2(index):
root = "D:\PyProject\Pyspark\keshe\\result" + str(index) + "/part-00000.json"
date = []
cases = []
deaths = []
with open(root, 'r') as f:
while True:
line = f.readline()
if not line: # 到 EOF,返回空字符串,则终止循环
break
js = json.loads(line)
date.append(str(js['date']))
cases.append(int(js['caseIncrease']))
deaths.append(int(js['deathIncrease']))
(
Line(init_opts=opts.InitOpts(width="1600px", height="800px"))
.add_xaxis(xaxis_data=date)
.add_yaxis(
series_name="新增确诊",
y_axis=cases,
markpoint_opts=opts.MarkPointOpts(
data=[
opts.MarkPointItem(type_="max", name="最大值")
]
),
markline_opts=opts.MarkLineOpts(
data=[opts.MarkLineItem(type_="average", name="平均值")]
),
)
.set_global_opts(
title_opts=opts.TitleOpts(title="美国每日新增确诊折线图", subtitle=""),
tooltip_opts=opts.TooltipOpts(trigger="axis"),
toolbox_opts=opts.ToolboxOpts(is_show=True),
xaxis_opts=opts.AxisOpts(type_="category", boundary_gap=False),
)
.render("result2.html")
)
(
Line(init_opts=opts.InitOpts(width="1600px", height="800px"))
.add_xaxis(xaxis_data=date)
.add_yaxis(
series_name="新增死亡",
y_axis=deaths,
markpoint_opts=opts.MarkPointOpts(
data=[opts.MarkPointItem(type_="max", name="最大值")]
),
markline_opts=opts.MarkLineOpts(
data=[
opts.MarkLineItem(type_="average", name="平均值"),
opts.MarkLineItem(symbol="none", x="90%", y="max"),
opts.MarkLineItem(symbol="circle", type_="max", name="最高点"),
]
),
)
.set_global_opts(
title_opts=opts.TitleOpts(title="美国每日新增死亡折线图", subtitle=""),
tooltip_opts=opts.TooltipOpts(trigger="axis"),
toolbox_opts=opts.ToolboxOpts(is_show=True),
xaxis_opts=opts.AxisOpts(type_="category", boundary_gap=False),
)
.render("result2.html")
)
# 3.画出截止5.19,美国各州累计确诊、死亡人数和病死率--->表格
def drawChart_3(index):
root = "D:\PyProject\Pyspark\keshe\\result" + str(index) + "/part-00000.json"
allState = []
with open(root, 'r') as f:
while True:
line = f.readline()
if not line: # 到 EOF,返回空字符串,则终止循环
break
js = json.loads(line)
row = []
row.append(str(js['state']))
row.append(int(js['totalCases']))
row.append(int(js['totalDeaths']))
row.append(float(js['deathRate']))
allState.append(row)
table = Table()
headers = ["State name", "Total cases", "Total deaths", "Death rate"]
rows = allState
table.add(headers, rows)
table.set_global_opts(
title_opts=ComponentTitleOpts(title="美国各州疫情一览", subtitle="")
)
table.render("result3.html")
# 4.画出美国确诊最多的10个州——>词云图
def drawChart_4(index):
root = "D:\PyProject\Pyspark\keshe\\result" + str(index) + "/part-00000.json"
data = []
with open(root, 'r') as f:
while True:
line = f.readline()
if not line: # 到 EOF,返回空字符串,则终止循环
break
js = json.loads(line)
row = (str(js['state']), int(js['totalCases']))
data.append(row)
c = (
WordCloud()
.add("", data, word_size_range=[20, 100], shape=SymbolType.DIAMOND)
.set_global_opts(title_opts=opts.TitleOpts(title="美国各州确诊Top10"))
.render("result4.html")
)
# 5.画出美国死亡最多的10个州——>象柱状图
def drawChart_5(index):
root = "D:\PyProject\Pyspark\keshe\\result" + str(index) + "/part-00000.json"
state = []
totalDeath = []
with open(root, 'r') as f:
while True:
line = f.readline()
if not line: # 到 EOF,返回空字符串,则终止循环
break
js = json.loads(line)
state.insert(0, str(js['state']))
totalDeath.insert( 0, int(js['totalDeaths']))
c = (
PictorialBar()
.add_xaxis(state)
.add_yaxis(
"",
totalDeath,
label_opts=opts.LabelOpts(is_show=False),
symbol_size=18,
symbol_repeat="fixed",
symbol_offset=[0, 0],
is_symbol_clip=True,
symbol=SymbolType.ROUND_RECT,
)
.reversal_axis()
.set_global_opts(
title_opts=opts.TitleOpts(title="PictorialBar-美国各州死亡人数Top10"),
xaxis_opts=opts.AxisOpts(is_show=False),
yaxis_opts=opts.AxisOpts(
axistick_opts=opts.AxisTickOpts(is_show=False),
axisline_opts=opts.AxisLineOpts(
linestyle_opts=opts.LineStyleOpts(opacity=0)
),
),
)
.render("result5.html")
)
# 6.找出美国确诊最少的10个州——>词云图
def drawChart_6(index):
root = "D:\PyProject\Pyspark\keshe\\result" + str(index) + "/part-00000.json"
data = []
with open(root, 'r') as f:
while True:
line = f.readline()
if not line: # 到 EOF,返回空字符串,则终止循环
break
js = json.loads(line)
row = (str(js['state']), int(js['totalCases']))
data.append(row)
c = (
WordCloud()
.add("", data, word_size_range=[100, 20], shape=SymbolType.DIAMOND)
.set_global_opts(title_opts=opts.TitleOpts(title="美国各州确诊最少的10个州"))
.render("result6.html")
)
# 7.找出美国死亡最少的10个州——>漏斗图
def drawChart_7(index):
root = "D:\PyProject\Pyspark\keshe\\result" + str(index) + "/part-00000.json"
data = []
with open(root, 'r') as f:
while True:
line = f.readline()
if not line: # 到 EOF,返回空字符串,则终止循环
break
js = json.loads(line)
data.insert(0, [str(js['state']), int(js['totalDeaths'])])
c = (
Funnel()
.add(
"State",
data,
sort_="ascending",
label_opts=opts.LabelOpts(position="inside"),
)
.set_global_opts(title_opts=opts.TitleOpts(title=""))
.render("result7.html")
)
# 8.美国的病死率--->饼状图
def drawChart_8(index):
root = "D:\PyProject\Pyspark\keshe\\result" + str(index) + "/part-00000.json"
values = []
with open(root, 'r') as f:
while True:
line = f.readline()
if not line: # 到 EOF,返回空字符串,则终止循环
break
js = json.loads(line)
if str(js['state']) == "USA":
values.append(["Death(%)", round(float(js['deathRate']) * 100, 2)])
values.append(["No-Death(%)", 100 - round(float(js['deathRate']) * 100, 2)])
c = (
Pie()
.add("", values)
.set_colors(["blcak", "orange"])
.set_global_opts(title_opts=opts.TitleOpts(title="全美的病死率"))
.set_series_opts(label_opts=opts.LabelOpts(formatter="{b}: {c}"))
.render("result8.html")
)
# 可视化主程序:
index = 1
while index < 9:
funcStr = "drawChart_" + str(index)
eval(funcStr)(index)
index += 1
plot01.py
"""
本py文件:
绘制美国分县疫情地图
绘制美国新增病例增长曲线
绘制美国疫情死亡病例增长曲线
使用三次指数平滑法进行预测
"""
import matplotlib.pyplot as plt
import numpy as np
import plotly.graph_objs as go
import pandas as pd
import plotly.express as px
import datetime
from statsmodels.tsa.api import ExponentialSmoothing, SimpleExpSmoothing, Holt
from plotly.offline import init_notebook_mode, iplot
from urllib.request import urlopen
import json
import plotly.io as pio
import plotly.offline as py
pio.renderers.default = "notebook_connected"
init_notebook_mode(connected=True)
ds=pd.read_csv("C:\\Users\\26909\\Desktop\\us-counties-latest.csv")
# 绘制美国疫情分县地图
with urlopen('https://raw.githubusercontent.com/plotly/datasets/master/geojson-counties-fips.json') as response:
counties = json.load(response)
fig = px.choropleth_mapbox(ds, geojson=counties, locations='fips', color='cases',
color_continuous_scale="Turbo",
range_color=(0, 20000),
mapbox_style="carto-positron",
hover_name ="county",
zoom=3, center = {"lat": 37.0902, "lon": -95.7129},
opacity=0.5
)
fig.update_layout(margin={"r":0,"t":0,"l":0,"b":0})
fig.show()
py.plot(fig, filename='1.html')
# 绘制美国新增病例数增长曲线
ds['date']= pd.to_datetime(ds['date'])
ds.Timestamp=ds["date"]
ds.index = ds.Timestamp
df = ds.resample('D').sum()
fig = go.Figure()
fig.add_trace(go.Scatter(
x=df.index,
y=df.cases,
name="Cases in USA"
))
fig.update_layout(
font=dict(
family="Courier New, monospace",
size=18,
)
)
fig.show()
py.plot(fig, filename='2.html')
# 绘制美国疫情死亡病例增长曲线
fig = go.Figure()
fig.add_trace(go.Scatter(
x=df.index,
y=df.deaths,
name="Cases in USA"
))
fig.update_layout(
font=dict(
family="Courier New, monospace",
size=18,
)
)
fig.show()
py.plot(fig, filename='3.html')
# 三次指数平滑法预测模型
start = datetime.datetime.strptime("2021-02-14", "%Y-%m-%d")
end = datetime.datetime.strptime("2021-07-13", "%Y-%m-%d")
date_generated = [start + datetime.timedelta(days=x) for x in range(0, (end-start).days)]
dt=[]
for date in date_generated:
dt.append(date.strftime("%Y-%m-%d"))
dtd=pd.DataFrame()
dtd["Date"]=dt
df = pd.read_csv("C:\\Users\\26909\\Desktop\\us-counties-latest.csv")
fig, axes = plt.subplots(2, 1, figsize = (10, 10))
data=pd.DataFrame(df.groupby("date").agg({"cases": "sum","deaths": "sum"}))
ddf=pd.DataFrame(df.groupby("date").agg({"cases": "sum","deaths": "sum"}))
data.insert(2,'increasecases','0')
data.insert(3,'increasedeaths','0')
data=np.asarray(data)
for i in range(1,844):
data[i][2]=data[i][0]-data[i-1][0]
data[i][3] = data[i][1] - data[i - 1][1]
data[0][2]=1
data[0][3]=0
fit1 = ExponentialSmoothing(data[:640,2] ,seasonal_periods=130 ,trend='add', seasonal='add',).fit()
pred = fit1.forecast(149)
pred=pred.astype(int)
dtd["Holt"]=pred
dtd.Timestamp=dtd["Date"]
dtd.index = dtd.Timestamp
fig = go.Figure()
fig.add_trace(go.Scatter(
x=ddf.index,
y=data[:,2],
name="Confirmed Cases"
))
fig.add_trace(go.Scatter(
x=dtd.index,
y=dtd.Holt,
name="Future Prediction"
))
fig.update_layout(
font=dict(
family="Courier New, monospace",
size=18,
)
)
fig.show()
py.plot(fig, filename='4.html')
plot02.py
"""
本py文件:
绘制美国疫情分州地图
绘制美国确诊病例各月份占比饼状图
"""
import pandas as pd
from plotly.offline import init_notebook_mode, iplot
init_notebook_mode(connected=True)
import plotly.graph_objs as go
import plotly.offline as py
import matplotlib.pyplot as plt
import warnings
warnings.filterwarnings("ignore")
plt.style.use('ggplot')
dataset=pd.read_csv("C:\\Users\\26909\\Desktop\\us-counties-latest.csv")
data = dataset.groupby("state").sum().reset_index()
# 绘制美国疫情分州地图
dat = [dict(
type="choropleth",
locations=['AL', 'AK', 'AS','AZ', 'AR', 'CA', 'CO', 'CT', 'DE', "DC", 'FL', 'GA', "GU", 'HI', 'ID', 'IL', 'IN', 'IA',
'KS', 'KY', 'LA', 'ME', 'MD', 'MA', 'MI', 'MN', 'MS', 'MO', 'MT', 'NE', 'NV', 'NH', 'NJ', 'NM', 'NY',
'NC', 'ND', "NM", 'OH', 'OK', 'OR', 'PA', "PR", 'RI', 'SC', 'SD', 'TN', 'TX', 'UT', 'VT', "VI", 'VA',
'WA', 'WV', 'WI', 'WY'],
locationmode='USA-states',
z=data["cases"],
text=data["state"],
colorscale=[[0, "#641b0c"], [0.85, "#f85466"], [0.9, "#fa7483"],
[0.94, "#f88f9a"], [0.97, "#f7bbc1"], [1, "#f1c6cb"]],
autocolorscale=False,
reversescale=True,
marker=dict(
line=dict(
width=0.5,
color='rgba(100,100,100)',
),
),
colorbar=dict(
title="Total Cases",
)
)]
layout = dict(
title={
'text': "Total Cases of States",
'y': 0.9,
'x': 0.5,
'xanchor': 'center',
'yanchor': 'top'},
geo=dict(
showframe=False,
showcoastlines=True,
projection=dict(
type="albers usa"
),
scope="usa"
)
)
fig = go.Figure(data=dat, layout=layout)
iplot(fig)
fig.show()
py.plot(fig, filename='5.html')
# 绘制美国确诊病例各月份占比饼状图
a = []
for x in dataset["date"]:
a.append(x.split("-")[1])
dataset["Month"] = a
data = dataset.groupby("Month").sum().reset_index()
data.replace("01", "January", inplace=True)
data.replace("02", "February", inplace=True)
data.replace("03", "March", inplace=True)
data.replace("04", "April", inplace=True)
data.replace("05", "May", inplace=True)
data.replace("06", "June", inplace=True)
data.replace("07", "July", inplace=True)
data.replace("08", "August", inplace=True)
data.replace("09", "Semptember", inplace=True)
data.replace("10", "October", inplace=True)
data.replace("11", "November", inplace=True)
data.replace("12", "December", inplace=True)
cases = [each for each in data.cases]
deaths = [each for each in data.deaths]
pie_list = [each for each in data.cases]
labels = data.Month
fig = {
"data": [
{
"values": pie_list,
"labels": labels,
"domain": {"x": [0, 0.5]},
"name": "Cases per Month",
"hoverinfo": "label+percent+name",
"hole": .3,
"type": "pie"}, ],
"layout": {
"title": "Cases per Month",
"annotations": [
{"font": {"size": 20},
"showarrow": False,
"text": "Number of Cases",
"x": 0.20,
"y": 1.19
}, ]
}
}
iplot(fig)
py.plot(fig, filename='6.html')
DBSCAN.py
import pandas as pd
import scipy
def fit_dbscan_iterable(X, minPts=3, radius=1, verbose=True, anim=True, animFrames=100, animFramesAfter=40,
animFramesBefore=10):
"""Find clusters using dbscan algorithm
Parameters
----------
X : list of points
array of points to find the clusters in (accept: list, numpy or pandas)
minPts : int (default=3)
minimum points to be considered a core
radius : float (default=1)
radius to find neighbors
verbose : bool (default=True)
print some messages during processing
anim : bool (default=True)
return intermediary frames to make a animation
Returns
-------
generator object -> (y, cores, cursor)
returns a generator object (iterable) that will
return a tuple of (y, cores and cursor) for
each frame
"""
radius = float(radius)
if isinstance(X, pd.DataFrame):
X = X.to_numpy()
def locate_neigbors(x1):
nonlocal X, radius
result = []
for x2i, x2 in enumerate(X):
d = scipy.spatial.distance.euclidean(x1, x2)
if d < radius:
result.append([x2i, x2])
return result;
animEach = len(X) / animFrames
animFrame = 0;
# dados gerais
cluster_id = 0
y = [0 for _ in X]
cores = [0 for _ in X]
if anim:
for i in range(animFramesBefore):
yield (y, cores, None)
for x1i, x1 in enumerate(X):
if y[x1i] == 0:
N = locate_neigbors(x1)
if len(N) >= minPts:
cluster_id = cluster_id + 1
visited = []
while len(N) > 0:
(x2i, x2) = N.pop()
if x2i in visited: continue
visited.append(x2i)
animFrame = animFrame + 1
if (animFrame % animEach) == 0:
if anim: yield (y, cores, x2)
if cores[x2i] == 0:
y[x2i] = cluster_id
N2 = locate_neigbors(x2)
if (len(N2) >= minPts):
cores[x2i] = 1
for x3i, x3 in N2:
if y[x3i] != cluster_id and cores[x3i] == 0:
N.insert(0, [x3i, x3])
if anim:
for i in range(animFramesAfter):
yield (y, cores, None)
# finalizado, retorna última frame
yield (y, cores, None)
def fit_dbscan(X, minPts=3, radius=1, verbose=True, anim=False):
"""Find clusters using dbscan algorithm
Parameters
----------
X : list of points
array of points to find the clusters in (accept: list, numpy or pandas)
minPts : int (default=3)
minimum points to be considered a core
radius : float (default=1)
radius to find neighbors
verbose : bool (default=True)
print some messages during processing
anim : bool (default=True)
return intermediary frames to make a animation
Returns
-------
if anim=False:
tuple (y, cores, cursor):
y: list of scalars
the clusterids which each point belongs to
cores: list of scalars
1 if the corresponding X is a core
cursor: tensor (point)
None in the last frame
if anim=True:
list of tuples (y, cores, cursor)
"""
if anim:
return list(fit_dbscan_iterable(X, minPts, radius, verbose, anim=True))
else:
return next(fit_dbscan_iterable(X, minPts, radius, verbose, anim=False))
def bloco1():
global df
from datetime import datetime
import pandas as pd
import os
pd.options.display.max_columns = None
df = pd.read_csv("C:\\Users\\26909\\Desktop\\us-counties-latest.csv"
)
df.insert(1, "wday", df['date'].apply(lambda x: int(datetime.strptime(x, '%Y-%m-%d').strftime('%w'))))
df.insert(1, "mday", df['date'].apply(lambda x: int(datetime.strptime(x, '%Y-%m-%d').strftime('%d'))))
df = df.dropna()
df = df.sample(1500)
return df
def bloco():
import matplotlib.pyplot as plt
X = df[['cases', 'deaths']].to_numpy()
y, cores, cursor = fit_dbscan(X, radius=20, minPts=5, anim=False)
fig = plt.figure(figsize=(12,5), dpi=300)
hsv = plt.cm.get_cmap('hsv', 10)
plt.clf()
plt.title(f"Covid-19 casos x mortes",fontsize=7)
plt.rcParams.update({'font.size': 6})
plt.xticks(())
plt.yticks(())
ax = fig.subplots()
ax.set_xlabel('casos (log)')
ax.set_ylabel('mortes (log)')
fig.patch.set_visible(False)
ax.patch.set_visible(False)
plt.yscale('log')
plt.xscale('log')
res = []
ax.scatter(
[X[j][0] for j in range(len(X)) if y[j] == 0],
[X[j][1] for j in range(len(X)) if y[j] == 0],
color='#c0c0c0',
label='Outlier',
marker='o',
s=1
)
for i in range(1, 10):
ax.scatter(
[X[j][0] for j in range(len(X)) if y[j] == i and cores[j] == 0],
[X[j][1] for j in range(len(X)) if y[j] == i and cores[j] == 0],
color=hsv(i),
marker='o',
s=1
)
ax.scatter(
[X[j][0] for j in range(len(X)) if y[j] == i and cores[j] == 1],
[X[j][1] for j in range(len(X)) if y[j] == i and cores[j] == 1],
color=hsv(i),
label=f'Cluster {i}',
marker='o',
s=1
)
if not (cursor is None):
ax.scatter(
[cursor[0]],
[cursor[1]],
color='#000000',
label='Cursor',
marker='o',
s=1
)
ax.legend()
plt.show()
bloco1()
bloco()